
Una nueva técnica permitirá detectar virus en el campo. En este caso, una parcela de ñame en Guadalupe. Crédito:D. Filloux, Cirad
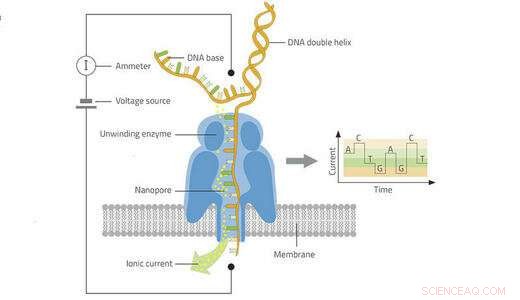
Por primera vez en virología vegetal, Un equipo del CIRAD utilizó recientemente la tecnología de nanoporos para secuenciar los genomas completos de dos virus de ARN de ñame. Esta técnica de biología molecular poco utilizada pero prometedora allana el camino para nuevas herramientas para el diagnóstico de campo de plantas, enfermedades animales y humanas.
Un nuevo alto rendimiento La técnica de secuenciación portátil se ha desarrollado en los últimos años con fines de salud humana y animal. Utiliza laboratorios móviles para diagnosticar virus como el Ébola o el Zika casi al instante. en el campo. El diagnóstico es rápido y temprano, lo que evita la necesidad de transferir muestras contaminadas.
"La tecnología se caracteriza por la producción de largas secuencias de nucleótidos, que permite secuenciar todo el genoma viral, "Philippe Roumagnac, un virólogo del CIRAD, explica. CIRAD fue uno de los primeros laboratorios del mundo en probar y validar su uso en virología vegetal. "Usando una planta de ñame enferma, Solo nos llevó unas pocas horas secuenciar el genoma completo de dos virus de ARN monocatenario, un macluravirus y un potyvirus, ", añade su colega Denis Filloux.
Como ocurre con la virología humana, el hecho de que la técnica haya sido validada en un laboratorio de virología vegetal allana el camino para el tiempo real, detección móvil de crónicas, virus vegetales estacionales o emergentes, incluso en áreas aisladas. Al acortar el tiempo que transcurre entre el muestreo y el diagnóstico, la tecnología ayudará a las redes de vigilancia epidemiológica a detectar organismos nocivos en una etapa más temprana.
Crédito:Kerstin Göpfrich