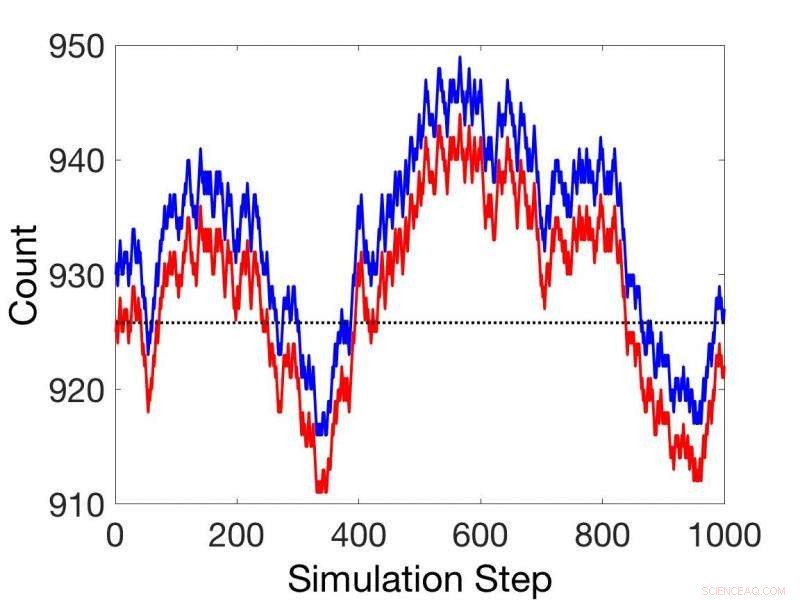
Comparaciones entre simulaciones cinéticas estocásticas en estado no estacionario (rojo) y simulaciones utilizando el teorema de reacción acoplada (azul). Crédito:Laboratorio Nacional del Noroeste del Pacífico
La ley de la acción de masas se describió por primera vez hace 150 años. Hoy dia, sigue siendo un concepto básico en todos los textos de química de primer año. La ley establece que la velocidad de una reacción química es proporcional a la concentración de los reactivos. Para soluciones en equilibrio dinámico, la ley predice cómo se comportarán esas soluciones.
Pero es un desafío aplicar la ley de acción de masas a sistemas complejos, como el metabolismo biológico. Para uno, la mayoría de las constantes de velocidad no están disponibles, y medir los que faltan es muy laborioso.
En investigación biológica, medir las constantes de velocidad requiere aislar y purificar enzimas individuales, luego determinando las constantes de velocidad con espectrofotometría, es decir, por la cantidad de luz que absorbe una sustancia química. Esto ralentiza las velocidades de rendimiento, lo que dificulta la caracterización de cada nuevo genoma. Después de todo, hacer esto requiere miles de parámetros de tasa.
Debido a estos problemas, Se ha determinado un gran número de parámetros de velocidad enzimática para unos pocos sistemas bien descritos. como Escherichia coli. Para la mayoría de los sistemas biológicos, los científicos aún no han obtenido constantes de velocidad.
Mientras tanto, el método de elección para modelar procesos biológicos a gran escala (como el metabolismo) implica modelos de flujo basados en restricciones. Pero estos modelos adolecen de una falta de física con respecto a la ley de acción de masas, limitar la capacidad de los enfoques basados en restricciones incluso cuando se amplían para incluir la termodinámica y otros factores.
En respuesta, algunos investigadores han propuesto estudiar reacciones elementales utilizando la ley de reacción de masas a través de afinidades de reacción en lugar de constantes de velocidad.
Un nuevo artículo en la revista Biologia fisica por William R. Cannon y Scott E. Baker en el Pacific Northwest National Laboratory revisa la relación entre la afinidad de reacción y la energía libre para reacciones aisladas utilizando conceptos de integración termodinámica estadística. Cannon y Baker se propusieron demostrar explícitamente la relación entre las formulaciones cinéticas de tasas que utilizan constantes de velocidad y una formulación de termodinámica estadística que utiliza potenciales químicos.
Posteriormente presentan un teorema para reacciones acopladas basado en potenciales químicos, que puede proporcionar velocidades relativas para todas las reacciones acopladas secuencialmente en todas las condiciones, estado estacionario o no. Esto evita la necesidad de constantes de velocidad en muchas aplicaciones.
El uso de potenciales químicos en lugar de constantes de velocidad representa un salto metodológico que, según los autores, podría indicar un gran avance para el modelado predictivo en biología de sistemas.
El nuevo método permitirá complejos, Los sistemas biológicos a gran escala, como el metabolismo, se modelarán con cinética y termodinámica. Esto dará como resultado predicciones más precisas para que el diseño de microorganismos se comporte de la manera deseada, como los que podrían aprovecharse para producir biocombustibles.
Es más, los autores dicen, El nuevo método arrojará información fundamental sobre el acoplamiento termodinámico entre los organismos vivos y su entorno.