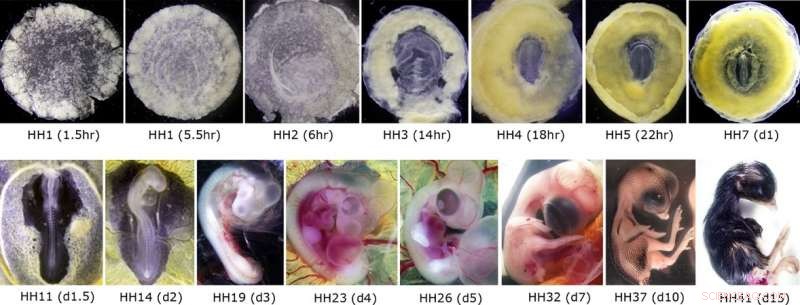
Imágenes representativas de embriones de pollo utilizados para la preparación de ARN y posterior secuenciación CAGE. Los paneles superiores muestran los embriones en el primer día de desarrollo embrionario (21 días en total), equivalente a las primeras cinco semanas de gestación humana (primeras tres semanas de desarrollo post-fertilización humana). Los paneles inferiores muestran los embriones desde el establecimiento del sistema circulatorio hasta aproximadamente una semana antes de la eclosión, similar al comienzo del tercer trimestre en el desarrollo humano. Crédito:Profesor Guojun Sheng
La investigación del desarrollo temprano en embriones humanos es imposible debido a consideraciones éticas, y es técnicamente difícil de realizar en otros mamíferos, haciendo que los recursos para este tipo de investigación sean extremadamente raros. Por ejemplo, el proyecto Functional Annotation of Mammalian Genome (FANTOM) 5 con sede en Japón, establecida en 2000, es un proyecto de colaboración mundial con el objetivo de identificar todos los elementos funcionales en los genomas de mamíferos, pero no tiene datos del sitio de inicio de la transcripción (TSS) basados en el análisis Cap de expresión génica (CAGE) para embriones humanos o para embriones murinos que representan etapas tempranas de desarrollo.
Para sortear esta escasez de información, una colaboración internacional de investigadores de Japón (Universidad de Kumamoto y RIKEN), Rusia (Universidad Federal de Kazán), España (Universidad de Cantabria), y Australia (Universidad de Australia Occidental) ha reunido el primer conjunto de TSS aviares de todo el genoma del mundo. Estos sitios marcan el punto de partida para la transcripción y son extremadamente importantes para la diferenciación celular y el desarrollo del embrión hasta el nacimiento. o, en el caso de una especie aviar, hasta la eclosión. Si bien las aves se ven bastante diferentes a los humanos, las primeras etapas de desarrollo son muy similares, y se ha demostrado que alrededor del 60% de los genes codificadores de proteínas humanas tienen correspondencia biunívoca con los ortólogos de pollo (genes que evolucionaron a partir de un ancestro común).
Usando tecnología CAGE, un método altamente confiable para encontrar TSS y elementos reguladores cis (los bloques de secuencia que regulan la posición y la robustez de la transcripción) en el genoma durante todo el período de desarrollo del pollito, los colaboradores pudieron mapear el 60% de todos los TSS de pollo en desarrollo con el genoma de pollo más reciente, con el otro 40% probablemente siendo promotores alternativos aún no caracterizados o genes de ARN no codificantes. El genoma actualizado se agregó al interactivo de RIKEN, plataforma en línea ZENBU (la palabra japonesa "zenbu" se traduce como "todos" en inglés) bajo el nombre "Chicken-ZENBU" y es de uso gratuito para todos.
Los investigadores también demostraron que su mapeo TSS de todo el genoma podría aplicarse a la tecnología CRISPR-on para activar genes específicos durante el desarrollo activando con éxito el gen T para la proteína Brachyury que codifica en la etapa de desarrollo HH10 (aproximadamente 1,5 días de desarrollo después de la puesta). Este logro fue seguido por varias activaciones de genes más exitosas, mostrando así la efectividad de CRISPR-on cuando se combina con el mapeo de TSS.
"En el proceso de desarrollo de nuestro método de creación de perfiles TSS de desarrollo basado en CAGE, identificamos nuevos factores de transcripción y sus módulos reguladores, así como varios genes nuevos de limpieza, ", dijo el líder del proyecto, el profesor Guojun Sheng del IRCMS de la Universidad de Kumamoto." Esperamos que este trabajo y los datos que agregamos a la base de datos en línea de ZENBU hagan avanzar enormemente la investigación del desarrollo tanto en aves como en mamíferos ".