Un equipo de investigación dirigido por la Universidad de Ciencia y Tecnología de Hong Kong (HKUST) ha desarrollado una plataforma de espectroscopía Raman mejorada de superficie (SERS) óptica plasmónica controlada por pinzas que utiliza el control de encendido y apagado de la luz para sondear varias especies de amilina en mezclas. a nivel de molécula única, revelando las estructuras heterogéneas de las especies de amilina dependientes del pH y los secretos detrás de los mecanismos de agregación de amiloide asociados con la diabetes tipo 2.
Al eliminar el promedio conjunto, las técnicas de una sola molécula disciernen la señal de moléculas individuales para revelar detalles ocultos y revolucionar nuestra comprensión de los sistemas moleculares complejos y heterogéneos. Los enfoques actuales de una sola molécula se limitan a la ultradilución y/o la inmovilización molecular porque el volumen de detección limitado por la difracción no se puede reducir más.
Mientras que ciertas biomoléculas participan en diversas interacciones que se ven significativamente afectadas por las concentraciones. Por ejemplo, como proteína típica intrínsecamente desordenada, el polipéptido amiloide de los islotes humanos (amilina, hIAPP) carece de estructuras secundarias estables pero posee propensión a la agregación gobernada por factores ambientales, como la concentración y el pH, para formar diferentes intermediarios oligoméricos y fibrillas de amiloide en la diabetes tipo II. pacientes.
El mecanismo molecular aún no está claro debido a los desafíos que plantea la detección de especies de amilina raras, transitorias y heterogéneas en una mezcla dinámica, lo que exige el desarrollo de métodos avanzados de una sola molécula.
En un avance reciente, el equipo de investigación dirigido por el Prof. Huang Jinqing, profesor asistente del Departamento de Química de HKUST, ha desarrollado con éxito una novedosa plataforma de molécula única que combina la manipulación plasmónica óptica y la medición SERS para reducir el volumen de detección y elevar la mejora de la señal, lo que permite una eficiencia y caracterización de una sola molécula de alto rendimiento para estudiar especies de amilina dependientes del pH en concentraciones fisiológicas.
Específicamente, el equipo construyó una unión plasmónica entre dos microperlas de sílice recubiertas con nanopartículas de Ag para atrapar una nanopartícula de Ag adicional y formar una nanocavidad dinámica tras la irradiación láser, que podría encapsular una o varias moléculas para caracterizaciones SERS sensibles.
Dado que tanto la captura plasmónica óptica como los fenómenos SERS están confinados espacialmente en la escala nanométrica, supera el límite de difracción óptica para permitir un control de posición preciso, minimizar el volumen de detección e impulsar la mejora SERS simultáneamente.
Además, los dímeros de microperlas de sílice recubiertos con nanopartículas de Ag construidos son más estables que los ensamblajes de nanopartículas de Ag convencionales en soluciones, lo que facilita la observación y localización de la unión plasmónica en microscopios regulares para mejorar la eficiencia y la reproducibilidad. Al cambiar la luz láser entre los estados "encendido" y "apagado", los investigadores pueden controlar la captura plasmónica óptica para modular el ensamblaje y desmontaje de la nanocavidad dinámica para muestreo de alto rendimiento y mediciones SERS simultáneas.
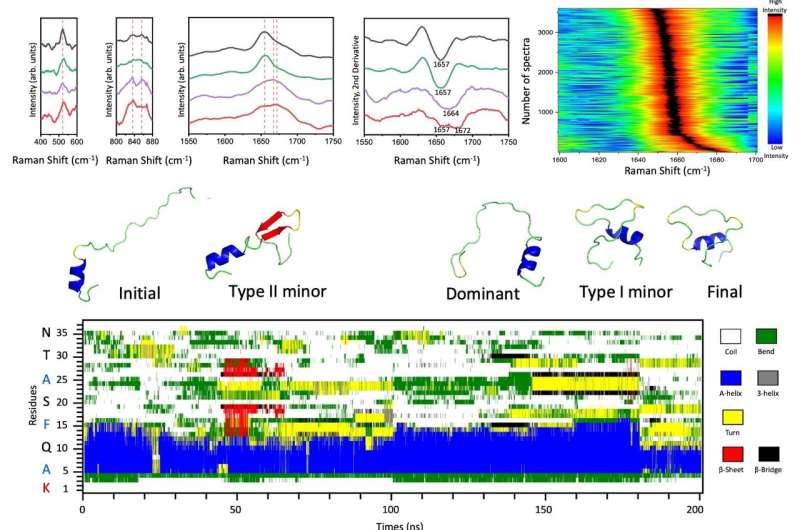
Utilizando esta eficiente plataforma de una sola molécula, el equipo de investigación aprovechó una cantidad estadísticamente significativa de espectros SERS subyacentes a las características estructurales de varias especies de amilina en dos condiciones fisiológicas distintas:los gránulos secretores de las células β pancreáticas a pH 5,5 y los compartimentos extracelulares a pH 7,4, respectivamente.
Se identificaron dos tipos de especies de amilina poco pobladas a partir de sus monómeros predominantes en la etapa temprana de agregación amiloide en pH neutro, que contienen una estructura de giro crítica o una horquilla β corta con terminal C restringido, según lo respaldado por simulaciones de dinámica molecular (MD). .
Un cambio tan leve en el equilibrio entre las diferentes especies de amilina podría impulsar un desarrollo irreversible de amiloide incluso después del ajuste posterior del pH de 7,4 a 5,5. Por lo tanto, la caracterización estructural directa de estas especies de amilina dentro de mezclas heterogéneas indica el impacto del pH en sus interacciones intra e intermoleculares y arroja luz sobre el mecanismo detrás de la agregación de amiloide regulada por el pH para comprender la diabetes tipo 2.
"Presentamos una estrategia fácil de usar que reduce el volumen de detección, mejora la señal molecular y aumenta la eficiencia del recambio", explicó el profesor Huang. "Nuestra plataforma de una sola molécula puede adquirir una gran cantidad de espectros SERS como instantáneas moleculares, comparables a las obtenidas mediante simulaciones MD. Al analizar estadísticamente los detalles estructurales a nivel de una sola molécula, podemos reconstruir las propiedades generales y obtener resultados únicos. información sobre la población y la probabilidad de tipos de moléculas específicas dentro de la mezcla heterogénea. Tiene el potencial de descubrir misterios ocultos en sistemas complejos".
El estudio fue publicado recientemente en Nature Communications. .
Más información: Wenhao Fu et al, Caracterización SERS de una sola molécula controlada por pinzas plasmónicas ópticas eficientes de especies de amilina dependientes del pH en medios acuosos, Nature Communications (2023). DOI:10.1038/s41467-023-42812-3
Información de la revista: Comunicaciones sobre la naturaleza
Proporcionado por la Universidad de Ciencia y Tecnología de Hong Kong