Diseño de motores y montaje experimental. a,b, Esquemas de un pedestal y una plataforma triangular, respectivamente. Los cilindros indican dobles hélices de ADN. c, Ilustración esquemática de los pasos de montaje del motor. d,e, Componentes del brazo del rotor. f, Izquierda, ilustración esquemática de la configuración experimental para observar la dinámica del motor en un microscopio TIRF invertido. El pedestal se fija a través de varios enlaces de biotina-neutravidina a un cubreobjetos de microscopio. Estrella naranja, tintes Cy5. Estrellas azules, posiciones de etiquetado para hebras del generador de imágenes DNA-PAINT. A la derecha, dos electrodos de platino se sumergen en la cámara de líquido desde arriba y se conectan a un generador de funciones que genera una corriente alterna de onda cuadrada para crear una modulación energética de eje fijo que actúa en todos los motores. Crédito:Naturaleza (2022). DOI:10.1038/s41586-022-04910-y
Un equipo de investigación dirigido por la Universidad Técnica de Munich (TUM) logró por primera vez producir un motor eléctrico molecular utilizando el método del origami de ADN. La diminuta máquina hecha de material genético se autoensambla y convierte la energía eléctrica en energía cinética. Los nuevos nanomotores se pueden encender y apagar, y los investigadores pueden controlar la velocidad de rotación y la dirección de rotación.
Ya sea en nuestros automóviles, taladros o molinillos de café automáticos, los motores nos ayudan a realizar trabajos en nuestra vida cotidiana para realizar una amplia variedad de tareas. En una escala mucho más pequeña, los motores moleculares naturales realizan tareas vitales en nuestros cuerpos. Por ejemplo, una proteína motora conocida como ATP sintasa produce la molécula trifosfato de adenosina (ATP), que nuestro cuerpo utiliza para almacenar y transferir energía a corto plazo.
Si bien los motores moleculares naturales son esenciales, ha sido bastante difícil recrear motores a esta escala con propiedades mecánicas más o menos similares a las de los motores moleculares naturales como la ATP sintasa. Ahora, un equipo de investigación ha construido un motor rotativo molecular a nanoescala que funciona utilizando el método de origami de ADN y ha publicado sus resultados en Nature. . El equipo estuvo dirigido por Hendrik Dietz, profesor de Nanotecnología Biomolecular en TUM, Friedrich Simmel, profesor de Física de Sistemas Biológicos Sintéticos en TUM, y Ramin Golestanian, director del Instituto Max Planck de Dinámica y Autoorganización.
Un nanomotor autoensamblable
El nuevo motor molecular consta de ADN, material genético. Los investigadores utilizaron el método de origami de ADN para ensamblar el motor a partir de moléculas de ADN. Este método fue inventado por Paul Rothemund en 2006 y luego fue desarrollado por el equipo de investigación de TUM. Varias hebras largas de ADN sirven como base a las que se unen hebras de ADN adicionales como contrapartes. Las secuencias de ADN se seleccionan de tal manera que las hebras y los pliegues unidos crean las estructuras deseadas.
"Hemos estado avanzando en este método de fabricación durante muchos años y ahora podemos desarrollar objetos muy precisos y complejos, como interruptores moleculares o cuerpos huecos que pueden atrapar virus. Si pones las hebras de ADN con las secuencias correctas en solución, los objetos autoensamblar", dice Dietz.
El nuevo nanomotor fabricado con material de ADN consta de tres componentes:base, plataforma y brazo de rotor. La base tiene una altura aproximada de 40 nanómetros y está fijada a una placa de vidrio en solución mediante enlaces químicos sobre una placa de vidrio. Un brazo de rotor de hasta 500 nanómetros de longitud está montado en la base para que pueda girar. Otro componente es crucial para que el motor funcione según lo previsto:una plataforma que se encuentra entre la base y el brazo del rotor. Esta plataforma contiene obstáculos que influyen en el movimiento del brazo del rotor. Para pasar los obstáculos y girar, el brazo del rotor debe doblarse un poco hacia arriba, como un trinquete.

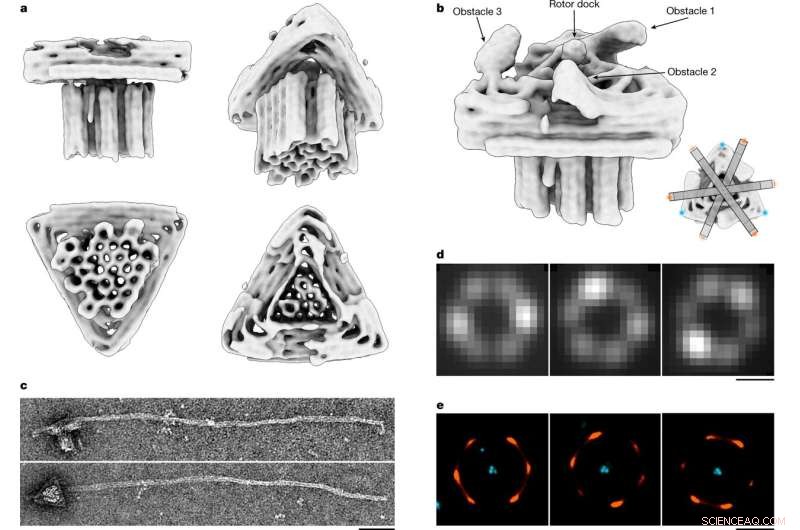
Análisis estructural del motor origami de ADN. a, Diferentes vistas de un mapa de densidad de electrones en 3D del bloque motor determinado por medio de crio-EM de una sola partícula (ver también Datos extendidos Fig. 4 y en el Banco de datos de microscopía electrónica (EMDB) bajo el código EMD-14358). b, detalle del mapa crio-EM del bloque del motor representado en diferentes umbrales de densidad en los que se pueden distinguir los tres obstáculos y el muelle del rotor. Recuadro, esquema que muestra los seis sitios de residencia preferidos del brazo del rotor. c, Imágenes ejemplares de TEM con tinción negativa de una variante de motor con un brazo de rotor largo adjunto. Barra de escala, 50 nm. d, Imágenes ejemplares de fluorescencia de una sola partícula. Barra de escala, 500 nm. Las imágenes muestran la desviación estándar de la intensidad media por píxel calculada en todos los fotogramas de los videos TIRF grabados. e, imágenes de DNA-PAINT que muestran las posiciones de la punta del brazo del rotor en relación con la plataforma del triángulo. Barra de escala, 500 nm. Crédito:Naturaleza (2022). DOI:10.1038/s41586-022-04910-y
Movimiento dirigido a través del voltaje de CA
Sin suministro de energía, los brazos del rotor de los motores se mueven aleatoriamente en una dirección u otra, impulsados por colisiones aleatorias con moléculas del solvente circundante. Sin embargo, tan pronto como se aplica voltaje de CA a través de dos electrodos, los brazos del rotor giran de manera dirigida y continua en una dirección.
"El nuevo motor tiene capacidades mecánicas sin precedentes:puede lograr pares en el rango de 10 piconewton por nanómetro. Y puede generar más energía por segundo que la que se libera cuando se dividen dos moléculas de ATP", explica Ramin Golestanian, quien dirigió el análisis teórico. del mecanismo del motor.
El movimiento dirigido de los motores resulta de una superposición de las fuerzas eléctricas fluctuantes con las fuerzas experimentadas por el brazo del rotor debido a los obstáculos de trinquete. El mecanismo subyacente realiza el llamado "trinquete browniano intermitente". Los investigadores pueden controlar la velocidad y la dirección de la rotación a través de la dirección del campo eléctrico y también a través de la frecuencia y amplitud del voltaje de CA.
"El nuevo motor también podría tener aplicaciones técnicas en el futuro. Si desarrollamos más el motor, posiblemente podríamos usarlo en el futuro para impulsar reacciones químicas definidas por el usuario, inspiradas en cómo la ATP sintasa hace que el ATP sea impulsado por la rotación. Entonces, por ejemplo ", las superficies podrían recubrirse densamente con dichos motores. Luego, agregaría materiales de partida, aplicaría un poco de voltaje de CA y los motores producirían el compuesto químico deseado", dice Dietz. Un motor biológico que consume combustible quiral impulsa la rotación en una dirección alrededor de un enlace covalente único