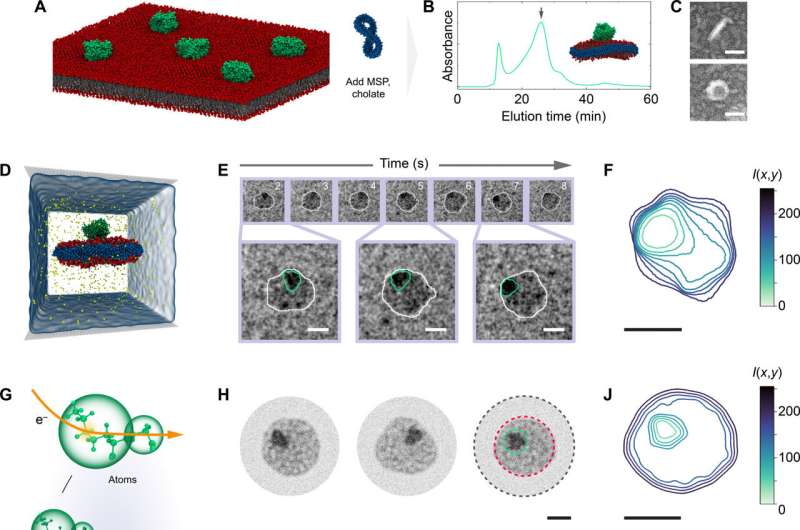
En una primera demostración de "videografía electrónica", los investigadores capturaron una imagen microscópica en movimiento de la delicada danza entre proteínas y lípidos que se encuentran en las membranas celulares. La técnica se puede utilizar para estudiar la dinámica de otras biomoléculas, liberándose de las limitaciones que han limitado la microscopía a imágenes fijas de moléculas fijas, afirman investigadores y colaboradores del Instituto de Tecnología de Georgia de la Universidad de Illinois en Urbana-Champaign.
"Vamos más allá de tomar fotografías únicas, que dan estructura pero no dinámica, a registrar continuamente las moléculas en el agua, su estado original", dijo el líder del estudio Qian Chen, profesor de ciencia e ingeniería de materiales de Illinois. "Realmente podemos ver cómo las proteínas cambian su configuración y, en este caso, cómo toda la estructura autoensamblada proteína-lípido fluctúa con el tiempo".
Los investigadores informaron sobre su técnica y sus hallazgos en la revista Science Advances. .
Las técnicas de microscopía electrónica obtienen imágenes a escala molecular o atómica, produciendo imágenes detalladas a escala nanométrica. Sin embargo, a menudo dependen de muestras que han sido congeladas o fijadas en su lugar, lo que deja a los científicos intentar inferir cómo se mueven e interactúan las moléculas, como intentar mapear la coreografía de una secuencia de baile a partir de un solo fotograma de una película.
"Esta es la primera vez que observamos una proteína a escala individual y no la hemos congelado ni etiquetado", dijo Aditi Das, profesora de Georgia Tech y autora correspondiente del estudio. "Por lo general, tenemos que cristalizar o congelar una proteína, lo que plantea desafíos a la hora de capturar imágenes de alta resolución de proteínas flexibles. Alternativamente, algunas técnicas utilizan una etiqueta molecular que rastreamos, en lugar de observar la proteína en sí. En este estudio estamos viendo la proteína tal como es, comportándose como lo hace en un ambiente líquido y viendo cómo los lípidos y las proteínas interactúan entre sí."
Los investigadores lograron la videografía combinando un novedoso método de microscopía electrónica de transmisión basado en agua con un modelado computacional detallado a nivel atómico. La técnica basada en agua consiste en encapsular gotas de escala nanométrica en grafeno para que puedan resistir el vacío en el que opera el microscopio. Comparar los datos de vídeo resultantes con modelos moleculares, que muestran cómo deberían moverse las cosas según las leyes de la física, ayuda a los investigadores no sólo a interpretar sino también a validar sus datos experimentales.
"Actualmente, esta es realmente la única forma experimental de filmar este tipo de movimiento a lo largo del tiempo", dijo John W. Smith, el primer autor del artículo, quien completó el trabajo mientras era estudiante de posgrado en Illinois. "La vida está en líquido y en movimiento. Estamos intentando llegar a los detalles más finos de esa conexión de forma experimental".
Para el nuevo estudio, la primera demostración publicada de la técnica de videografía electrónica, los investigadores examinaron discos a nanoescala de membranas lipídicas y cómo interactuaban con proteínas que normalmente se encuentran en la superficie o incrustadas en las membranas celulares.
"Las proteínas de membrana están en la interfaz entre las células y entre el interior y el exterior de la célula, controlando lo que entra y sale", dijo Smith. "Son objetivos abrumadoramente objetivos para la medicina; están involucrados en todo tipo de procesos como cómo se contraen nuestros músculos, cómo funcionan nuestros cerebros, el reconocimiento inmunológico; y mantienen unidos las células y los tejidos. Y toda la complejidad de cómo funciona una proteína de membrana proviene de no sólo su propia estructura, sino también cómo experimenta los lípidos que lo rodean."
La videografía electrónica permitió a los investigadores ver no sólo cómo se movía todo el conjunto de lípidos y proteínas, sino también la dinámica de cada componente. Los investigadores descubrieron que había regiones distintas dentro del nanodisco y más fluctuación y más estabilidad de lo esperado.
Si bien a menudo se supone que la influencia del movimiento de una proteína de membrana se limita a las moléculas de lípidos que la rodean directamente, los investigadores observaron fluctuaciones más dramáticas en un rango más amplio, dijo Smith. Las fluctuaciones tomaron la forma de dedos, como limo salpicado en una pared. Sin embargo, incluso después de un movimiento tan dramático, el nanodisco volvería a su configuración normal.
"El hecho de que vimos esos dominios y los vimos recuperarse de esos procesos sugiere que las interacciones entre la proteína y la membrana en realidad tienen un rango mayor de lo que comúnmente se piensa", dijo Smith.
Los investigadores planean utilizar su técnica de videografía electrónica para estudiar otros tipos de proteínas de membrana y otras clases de moléculas y nanomateriales.
"Podríamos estudiar canales iónicos que se abren y cierran para regular el flujo y las interacciones entre células utilizando esta plataforma", afirmó Chen.
Qian Chen también está afiliado al departamento de química, el Instituto Beckman de Ciencia y Tecnología Avanzadas, la Facultad de Medicina Carle Illinois y el Laboratorio de Investigación de Materiales de Illinois.
Más información: John W. Smith et al, Videografía electrónica de un tango de lípidos y proteínas, Science Advances (2024). DOI:10.1126/sciadv.adk0217
Información de la revista: Avances científicos
Proporcionado por la Universidad de Illinois en Urbana-Champaign