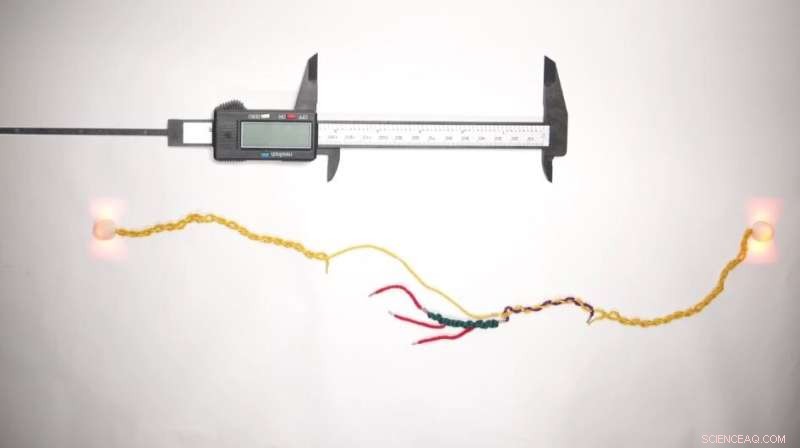
Crédito:Universidad de Harvard
A medida que los científicos han investigado los misterios de la vida a escalas cada vez más pequeñas, han inventado herramientas para ayudarlos a comprender lo que observan. Determinar la identidad de las moléculas de ADN y ARN ahora se ha convertido en un lugar común gracias al desarrollo comercial de las tecnologías de secuenciación de próxima generación, pero aún no ocurre lo mismo con las proteínas, que son actores de importancia crítica en casi todos los procesos biológicos. Las proteínas son mucho más complejas que el ADN y el ARN y, a menudo, se modifican químicamente, lo que hace que el objetivo de identificar fácilmente proteínas individuales dentro de una muestra (proteómica de molécula única) sea difícil de lograr.
Ahora, los científicos que trabajan en la Iniciativa de Robótica Molecular dentro del Instituto Wyss de la Universidad de Harvard, el Instituto Blavatnik de la Facultad de Medicina de Harvard (HMS) y el Hospital de Niños de Boston (BCH) han utilizado el ADN, el material fundamental de la vida misma, para crear lo que puede ser ser la regla más pequeña del mundo para medir proteínas.
Apodado "DNA Nanoswitch Calipers" (DNC), esta tecnología permite a los investigadores realizar mediciones de distancia en péptidos individuales (los componentes básicos de las proteínas) con alta precisión mediante la aplicación de pequeñas cantidades de fuerza. Al realizar rápidamente muchas mediciones de distancia en la misma molécula, DNC crea una "huella digital" única que se puede utilizar para identificarla en experimentos posteriores. El logro se informa en Nature Nanotechnology .
"Cuando intentas entender algo en biología, existen dos métodos principales de investigación:puedes observar a tu sujeto en su estado natural o puedes perturbarlo y ver cómo reacciona. Las observaciones pueden proporcionar mucha información biológica excelente, pero a veces la mejor manera de aprender sobre algo es interactuar físicamente con él", dijo el coautor correspondiente Wesley Wong, Ph.D., miembro asociado de la facultad en el Instituto Wyss y profesor asociado en HMS que también es investigador en BCH. . "Determinar el patrón de aminoácidos dentro de una molécula peptídica mediante la aplicación de fuerza es un nuevo paradigma en la búsqueda científica en curso de técnicas que nos permitan secuenciar proteínas tan fácilmente como actualmente secuenciamos ADN".
Usa la fuerza
DNC se basa en la tecnología subyacente del nanointerruptor de ADN:una sola hebra de ADN con "asas" moleculares unidas a él en múltiples puntos a lo largo de su longitud. Cuando dos de estos mangos se unen entre sí, crean un bucle en la hebra de ADN y la longitud total de la hebra se acorta. Cuando se aplica fuerza para separar los mangos, el hilo vuelve a su longitud original. La diferencia entre la longitud de la hebra en su estado enrollado y no enrollado refleja el tamaño del bucle y, por lo tanto, la distancia entre los mangos.
El equipo de investigación se dio cuenta de que podían llevar los nanointerruptores de ADN un paso más allá:si, en cambio, diseñaran los mangos para que se unan a una biomolécula, los mangos podrían "pellizcar" la molécula entre ellos como las dos puntas de un calibrador, en lugar de unirse a cada uno. otro. Al medir cómo la adición de la molécula objetivo entre los mangos cambió la longitud total del nanointerruptor de ADN en sus estados en bucle y sin bucle, el equipo planteó la hipótesis de que podrían medir efectivamente el tamaño de la molécula.
"De alguna manera, los nanointerruptores de ADN aprovechan uno de los métodos mecánicos más clásicos para medir objetos:simplemente aplique fuerza a algo y vea cómo cambia en respuesta", dijo el coautor principal Darren Yang, investigador postdoctoral en el Instituto Wyss y BCH. "Es un enfoque que realmente no hemos visto que se use en el campo de la proteómica de una sola molécula, porque aplicar fuerza a objetos tan pequeños es increíblemente desafiante. Pero estuvimos a la altura del desafío".
Para hacer realidad su idea de una nueva técnica de medición basada en la fuerza, Yang y sus colegas primero colocaron dos tipos diferentes de manijas en una molécula objetivo:una manija "fuerte" para anclar firmemente la molécula a un extremo del DNC, y varias manijas "débiles" que podrían unirse al otro extremo del DNC. Luego ataron ambos extremos del DNC a dos cuentas "atrapadas ópticamente" suspendidas en rayos láser. Al acercar las perlas, indujeron a uno de los mangos débiles de la molécula objetivo a unirse al DNC, creando un estado en bucle. Cuando luego aumentaron la fuerza al separar más las cuentas, el mango débil finalmente liberó su unión, devolviendo el DNC a su estado más largo y sin bucles.
El equipo primero probó esta técnica en moléculas simples de ADN monocatenario (ssDNA) y confirmó que el cambio en las mediciones de distancia entre el estado en bucle y sin bucle del DNC se correlacionaba directamente con la longitud de la molécula objetivo. Estos cambios de longitud podrían medirse con una precisión de nivel de angstrom (que es diez veces más pequeño que el ancho de una doble hélice de ADN), lo que permite identificar cambios de longitud tan pequeños como los de un solo nucleótido.
Debido a que la molécula objetivo contiene múltiples puntos débiles que pueden unirse al DNC, los ciclos repetidos de unir y romper esos puntos crean una serie de medidas de distancia entre el punto fuerte y los puntos débiles que son únicos para cada molécula medida. Esta "huella digital" se puede utilizar para identificar una molécula conocida dentro de una muestra o para inferir información estructural sobre una molécula desconocida.
Proteínas de sondeo
Habiendo confirmado que DNC podía medir de manera confiable el tamaño de las moléculas de ADN, los investigadores cambiaron el enfoque a su objetivo real:las proteínas. Diseñaron un péptido sintético (una cadena corta de aminoácidos) con una longitud y secuencia conocidas y repitieron el experimento, uniéndolo a un extremo del DNC a través de el mango fuerte y unir y romper repetidamente los enlaces entre sus mangos débiles y el DNC aplicando diferentes cantidades de fuerza. Descubrieron que todas las distancias que midió su herramienta entre los mangos fuerte y débil coincidían con las distancias esperadas según la longitud del DNC y las longitudes de los aminoácidos en el péptido. También obtuvieron resultados similares cuando usaron el DNC para medir un péptido linealizado de origen natural llamado NOXA BH3.
Este proceso también generó huellas dactilares de medición únicas para cada péptido. El equipo creó un modelo informático para predecir cuántas proteínas humanas podrían identificarse de forma única mediante este método y descubrió que más del 75 % de las proteínas en una base de datos de proteínas de uso común podrían identificarse a través de huellas dactilares con una probabilidad de al menos el 90%.
"En realidad, nos sorprendió un poco lo bien que funcionó esta técnica", dijo el coautor principal Prakash Shrestha, Ph.D., becario postdoctoral en el Instituto Wyss y BCH. "Las pinzas ópticas han existido durante décadas y el ciclo del ADN entre un estado en bucle y sin bucle ha existido durante unos 10 años, y no estábamos seguros de poder obtener medidas de resolución suficientemente alta combinando esas ideas. Pero resultó que estas huellas dactilares son muy eficaces para identificar proteínas".
Identificar moléculas de proteínas individuales es una hazaña impresionante en sí misma, pero poder hacerlo para múltiples proteínas simultáneamente es el verdadero santo grial para la proteómica de moléculas individuales. El equipo demostró además que al reemplazar las perlas ópticas con un sistema de pinzas magnéticas, pudieron realizar mediciones en múltiples péptidos diferentes en paralelo, así como determinar las concentraciones relativas de diferentes moléculas.
"La proteómica de una sola molécula sigue siendo en gran medida una quimera debido a los desafíos en el escalado y la resolución. Nuestro trabajo actual muestra que la toma de huellas dactilares de secuencia basada en la fuerza tiene el potencial de hacer realidad este sueño", dijo el coautor William Shih, Ph.D. , miembro principal de la facultad en el Instituto Wyss y profesor en HMS y el Instituto del Cáncer Dana-Farber. "Nuestra máxima ambición es leer eficientemente no solo las secuencias de proteínas, sino también las estructuras de proteínas de una manera de alto rendimiento".
El próximo paso de los científicos hacia ese objetivo es validar sus calibradores para mediciones estructurales de baja fuerza en proteínas plegadas y sus complejos, investigando su uso potencial para biología estructural y proteómica. También están trabajando para aumentar el rendimiento de la tecnología para acelerar aún más el análisis de muestras mixtas.
"Esta investigación integra la biofísica molecular con la nanotecnología de ADN de vanguardia, iniciada aquí en el Instituto Wyss, para permitirnos interactuar y analizar moléculas biológicas de una manera verdaderamente novedosa. Cuando William y Wesley plantearon por primera vez esta idea como un desafío central para la recién formada Molecular Robotics Initiative, realmente parecía ciencia ficción, pero ese es precisamente el tipo de proyecto que queremos emprender en Wyss. Estoy muy orgulloso del equipo por hacer realidad esta tecnología, tiene el potencial de cambiar por completo cómo hacemos ciencia y desarrollamos terapias", dijo el director fundador de Wyss, Don Ingber, M.D., Ph.D., quien también es profesor de biología vascular Judah Folkman en la Escuela de Medicina de Harvard y BCH, y profesor de bioingeniería en la Universidad de Harvard John A. Escuela Paulson de Ingeniería y Ciencias Aplicadas. Democratización del análisis de fuerza de una sola molécula de alto rendimiento