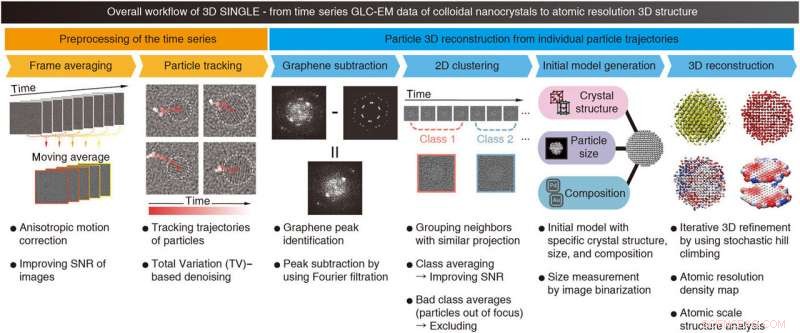
Flujo de trabajo general de SINGLE. Se muestran el flujo de trabajo general y las descripciones de cada paso de SINGLE. SINGLE consta de dos pasos principales:preprocesamiento de la serie temporal (naranja), incluyendo (i) el promedio del marco de la ventana de tiempo con corrección de movimiento anisotrópico y (ii) el seguimiento de la trayectoria de las partículas con el uso de eliminación de ruido basada en la variación total (TV), y reconstrucción 3D de partículas a partir de trayectorias de partículas individuales (azul), incluyendo (i) identificación y sustracción de antecedentes de grafeno, (ii) agrupación en 2D de tiempo restringido con exclusión de imágenes desenfocadas, (iii) generación inicial del modelo, y (iv) reconstrucción 3D y análisis de estructuras a escala atómica. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Los científicos de materiales suelen utilizar microscopía electrónica de transmisión (TEM) en fase de solución para revelar las propiedades fisicoquímicas únicas de las estructuras tridimensionales (3-D) de los nanocristales. En un nuevo informe sobre Avances de la ciencia , Cyril F. Reboul y un equipo de investigación de la Universidad de Monash, Australia, Universidad Nacional de Seúl, Corea del Sur, y el Laboratorio Nacional Lawrence Berkeley de EE. UU., desarrolló un método de reconstrucción 3-D browniano de una sola partícula. Para lograr esto, obtuvieron imágenes de conjuntos de nanocristales coloidales utilizando microscopía electrónica de transmisión de células líquidas de grafeno. El equipo obtuvo imágenes de proyección de nanocristales rotados de manera diferente utilizando un detector de electrones directo para obtener un conjunto de reconstrucciones tridimensionales. En este trabajo, Introdujeron métodos computacionales para reconstruir con éxito nanocristales 3D a resolución atómica y lo lograron rastreando partículas individuales a lo largo del tiempo. mientras resta el fondo de interferencia. El método también podría identificar / rechazar imágenes de baja calidad para facilitar estrategias personalizadas para la alineación 2-D / 3-D que difieran de las de la microscopía biológica crioelectrónica. El equipo puso los desarrollos a disposición a través de un paquete de software de código abierto conocido como SINGLE. El software gratuito está disponible en GitHub.
Usando SINGLE para Cristalografía
Los investigadores han sostenido avances en cristalografía en los últimos 50 años para transformar la comprensión existente de la química y la biología. Sin embargo, algunos objetivos, incluidos los nanocristales solubilizados, siguen siendo intratables con los métodos cristalográficos tradicionales. Por ejemplo, Los nanocristales coloidales contienen decenas a cientos de átomos y mantienen una variedad de aplicaciones en campos multidisciplinarios, incluida la electrónica, catálisis y sensores biológicos. Las versatilidad surgen de la alta sensibilidad de las propiedades de los nanocristales al tamaño, composición química y otras variables durante la síntesis. Típicamente, los científicos utilizan partículas individuales, Reconstrucción 3-D en biología estructural para determinar la estructura de proteínas. La técnica es relativamente nueva para la reconstrucción tridimensional in situ de nanocristales individuales solubilizados. En este trabajo, Reboul y col. desarrollado SINGLE; un método que se basaba en la reconstrucción tridimensional independiente de nanocristales individuales solubilizados, incluido el movimiento browniano. La técnica es el primer avance en estudio para resolver estructuras atómicas tridimensionales de nanocristales directamente de la fase de solución.
Seguimiento de trayectorias individuales de nanocristales. Seguimiento de los resultados de la partícula 1 (A y B) y 2 (C y D) a lo largo de la película (azul a rojo). Se muestran promedios de tiempo representativos de imágenes de partículas sin alinear sin procesar (50 cuadros) (B y D). Barras de escala, 1 nm. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679 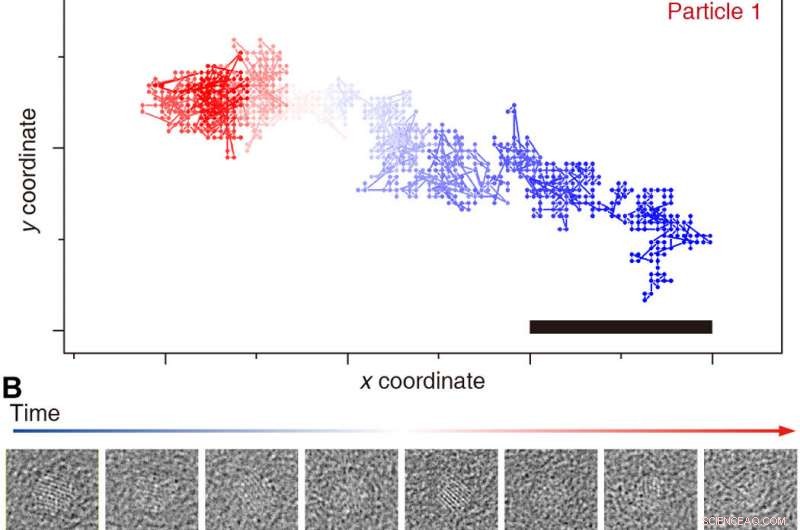
Los científicos introdujeron nuevos métodos de preprocesamiento para mejorar la relación señal-ruido (SNR) para rastrear las trayectorias de las partículas mientras eliminan la señal de fondo inducida por el grafeno. Los métodos computacionales avanzados podrían reconstruir con éxito en 3D a partir de los datos de microscopía electrónica de transmisión de células de grafeno líquido (GLC) in situ. En comparación con las técnicas existentes, el trabajo presentó la aplicabilidad de un método computacional sin precedentes para obtener reconstrucciones 3-D a resolución atómica para nanocristales dispersos en solución. Dividieron el flujo de trabajo ÚNICO en dos pasos principales (1) preprocesamiento y (2) reconstrucción tridimensional de partículas. Los científicos tenían como objetivo proporcionar el mayor rendimiento y eficiencia posibles en cualquier hardware de CPU, desde supercomputadoras hasta estaciones de trabajo o incluso portátiles.
En primer lugar, el equipo promedió la ventana de tiempo en varios fotogramas con movimiento anisotrópico para mejorar la relación señal-ruido, conduciendo a partículas visibles y una señal de grafeno mejorada. Luego, el equipo identificó las posiciones de las partículas manualmente en el promedio de la primera ventana de tiempo. Después de eso, el equipo desarrolló un modelo inicial basado en la estructura cristalográfica esperada, diámetro de partícula y elementos constituyentes y produjeron reconstrucciones 3-D con coordenadas atómicas ajustadas para el análisis de estructuras a escala atómica.
Agrupación en 2D con tiempo limitado. (A) Fracción de cambio angular a lo largo de la serie de tiempo. La línea discontinua roja es la línea de tendencia. Las direcciones de proyección cambian rápidamente en las regiones entre los fotogramas 1500 a 1600 (naranja), marcos 3800 a 3900 (verde), y marcos 5600 a 5700 (azul). Los recuadros son gráficos que representan la diferencia angular en la dirección de proyección en esas regiones. (B) Representación esquemática del promedio de clase 2D restringido en el tiempo. (C) Gráfico que muestra las clases asignadas para fotogramas individuales en la región de 1 a 400. El recuadro es un gráfico que muestra las clases asignadas en todos los fotogramas. (D) Promedios de clase obtenidos con agrupamiento y alineación 2D restringidos en el tiempo. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679 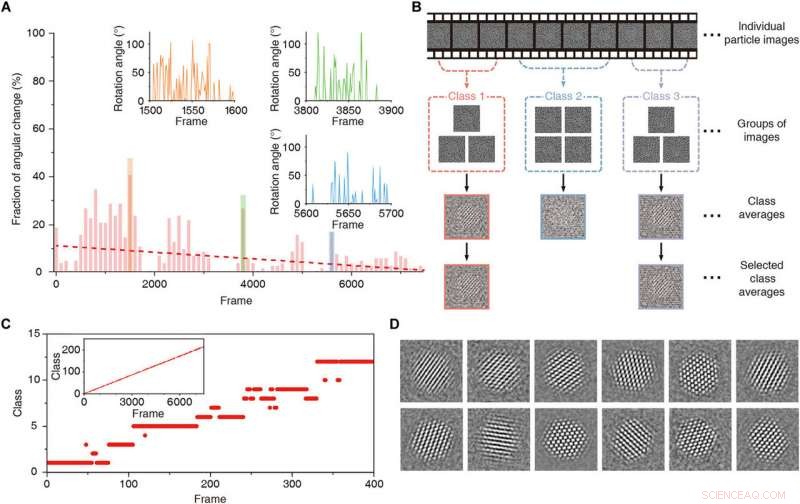
Reboul y col. introdujo un nuevo método de seguimiento que utiliza transformadas rápidas de Fourier y la correlación de fase para identificar un máximo de correlación con precisión de subpíxeles. El equipo eliminó el ruido de la ventana de tiempo de extracción utilizando la eliminación de ruido basada en la variación total (TV) y la eliminación de ruido combinada y el promedio de tiempo para proporcionar un método robusto para rastrear el movimiento de los nanocristales individuales en toda la muestra. El método les permitió discernir la forma general de los nanocristales y / o sus características cristalinas, lo que atestigua la robustez del algoritmo de seguimiento. Usando el método, también recuperaron trayectorias previamente desafiantes para obtener reconstrucciones 3-D y emplearon una trayectoria de partículas sustraídas de fondo en todos los pasos de procesamiento de imágenes para la sustracción de grafeno de la GLC (celda líquida de grafeno). El equipo caracterizó además la naturaleza de las rotaciones de nanocristales en el espacio altamente confinado de la celda de grafeno líquido. El método no fue trivial debido a la naturaleza probabilística del algoritmo de reconstrucción 3D. Por lo tanto, el equipo incorporó un enfoque determinista para mejorar la precisión del clúster, al tiempo que mejora la relación señal-ruido frente a los fotogramas individuales.
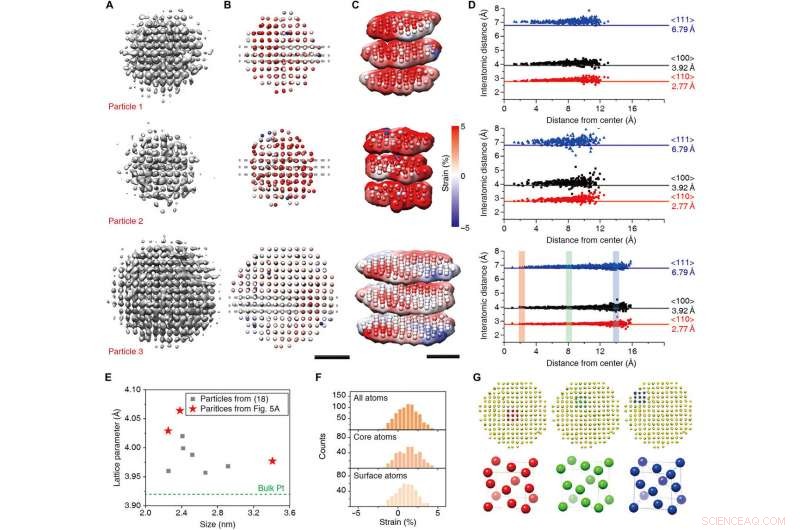
Resultados de reconstrucción 3D y análisis de estructuras a nivel atómico. (A a C) Mapas de densidad 3D (A), mapas de deformaciones radiales a partir de coordenadas atómicas ajustadas (B), y su representación de corte (C). (D) Distancias interatómicas en las direcciones de <110> (rojo), <100> (negro), y <111> (azul) para tres nanocristales. (E) Parámetros de celosía ajustados de nanocristales previamente reportados (18) (cuadrados grises) y nuevos resultados (estrellas rojas). (F) Histograma de la deformación radial de todos los átomos (arriba), átomos del núcleo (medio), átomos de superficie (parte inferior) de la partícula 3. (G) Estructura de celda unitaria del núcleo (rojo), medio (verde), y superficie (azul) de la partícula 3. Barras de escala, 1 nm. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Generando modelos
A continuación, los investigadores desarrollaron un modelo inicial utilizando el conocimiento de que las partículas tienen una disposición de posición atómica aproximadamente cúbica. Simularon las densidades atómicas utilizando factores de dispersión atómica 5-gaussianos. Las proyecciones 2-D de la densidad 3-D simulada representaron el carácter de las proyecciones en el núcleo del nanocristal, para superar problemas relacionados con la simetría traslacional y una señal de fondo interferente. El método de refinamiento tridimensional que se utiliza para la microscopía crioelectrónica biológica no podría aplicarse directamente a los datos de series de tiempo de nanocristales; por lo tanto, Reboul y col. introdujo modificaciones críticas. Utilizaron un esquema de refinamiento de dos etapas para establecer la forma correcta del nanocristal para permitir que los átomos y sus formas impulsen la alineación 3-D. Los investigadores eligieron tres nanocristales de diferentes tamaños que no fueron reconstruidos previamente para la evaluación comparativa. luego, usando mapas atómicos producidos con el método, Reboul y col. obtuvieron detalles estructurales microscópicos a nivel atómico. El trabajo también facilitó mapas atómicos que detallan el análisis de tensión y el análisis de la estructura de la celda unitaria.
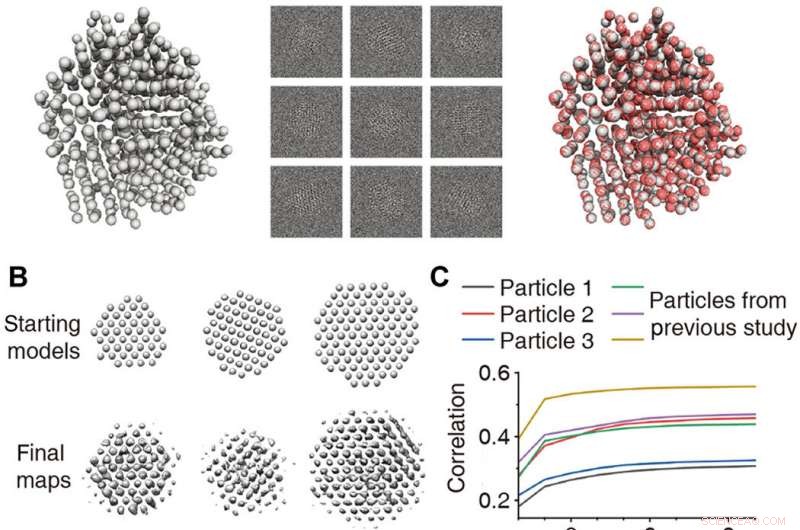
Validación de reconstrucciones 3D. (A) Reconstrucción en 3D de una partícula desordenada simulada con estructura atómica conocida. Izquierda:Modelo de un nanocristal desordenado obtenido por simulación de dinámica molecular. Medio:5000 imágenes simuladas multicorte con ruido añadido para dar una SNR =0,1. Derecha:mapa atómico (rojo) como resultado de la reconstrucción 3D superpuesta con el modelo de verdad del terreno (gris). (B) Modelos 3D iniciales y mapas de densidad 3D finales obtenidos a partir de datos experimentales. (C) Correlación entre las reproyecciones del mapa de densidad 3D refinado y las vistas de partículas experimentales trazadas como una función de iteración para la primera etapa de refinamiento 3D. Las partículas presentadas en este documento (negro, rojo, y color azul) y presentado en un estudio previo (18) (verde, púrpura, y color ocre). (D) Comparación de promedios de clase (indicados como proyección) con reproyecciones para validación de las tres estructuras. (E) Representación atómica dependiente del tiempo de las direcciones de proyección para las tres estructuras:blanco (comienzo) a rosa (medio) a rojo (final). Rojo, amarillo, y las flechas azules indican x, y, y ejes z, respectivamente. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Validando las reconstrucciones 3-D
Los investigadores generaron además un modelo de un nanocristal desordenado utilizando simulaciones de dinámica molecular para comprender la aplicabilidad de SINGLE a nanocristales altamente desordenados. Usando simulaciones de múltiples cortes, aplicaron movimiento de traslación y variaciones de desenfoque aleatorias para representar un movimiento de partículas realista. Luego obtuvieron un mapa de densidad tridimensional del nanocristal desordenado a partir de 500 imágenes simuladas con una relación señal / ruido de 0,1 y un modelo inicial con un orden cristalino perfecto para coincidir excelentemente con las partículas originales. El equipo obtuvo la distribución de las direcciones de proyección de los nanocristales giratorios para validar la calidad de la reconstrucción tridimensional y requerirá más estudios para comprender cómo las estructuras atómicas reales de los nanocristales afectan la dinámica de rotación.
De este modo, Cyril F. Reboul y sus colegas demostraron métodos computacionales en SINGLE para obtener mapas de densidad de nanocristales de resolución atómica. Usando una configuración avanzada de celda líquida, como celdas líquidas de grafeno con nanocámaras ordenadas, el equipo permitió el control del espesor del líquido para ampliar la aplicabilidad de SINGLE para una adquisición de datos eficiente. La suite SINGLE proporcionó una plataforma analítica eficiente, la primera en estudio, para comprender el origen estructural de las propiedades físicas y químicas únicas de los nanocristales en su fase de solución nativa.
© 2021 Science X Network