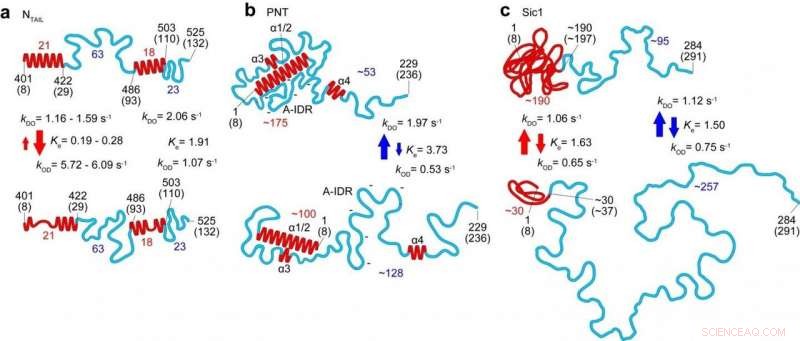
Características estructurales y dinámicas de tres desplazados internos (N COLA , PNT y Sic1) revelados por imágenes HS-AFM Los paneles superior e inferior corresponden a los estados más ordenados y menos ordenados, respectivamente. Los números en rojo y azul representan el número de aminoácidos contenidos en las respectivas regiones plegadas y regiones completamente desordenadas. respectivamente. Las flechas roja y azul indican la naturaleza cinética de los cambios en la altura de las regiones plegadas y la distancia de un extremo a otro de las regiones completamente desordenadas. respectivamente. Crédito:Universidad de Kanazawa
Nuestra comprensión de las proteínas biológicas no siempre se correlaciona con lo comunes o importantes que son. La mitad de todas las proteínas, moléculas que juegan un papel integral en los procesos celulares, están intrínsecamente desordenados, lo que significa que muchas de las técnicas estándar para sondear biomoléculas no funcionan con ellas. Ahora, los investigadores de la Universidad de Kanazawa en Japón han demostrado que su tecnología de microscopía de fuerza atómica de alta velocidad cultivada en casa puede proporcionar información no solo sobre las estructuras de estas proteínas, sino también sobre su dinámica.
Comprender cómo se junta una proteína proporciona pistas valiosas sobre sus funciones. El desarrollo de la cristalografía de proteínas en las décadas de 1930 y 1950 trajo a la vista varias estructuras de proteínas por primera vez, pero gradualmente se hizo evidente que una gran fracción de proteínas carecen de una estructura de un solo conjunto, lo que las hace intratables para la cristalografía de rayos X. Como son demasiado delgados para microscopía electrónica, las únicas alternativas viables para muchas de estas proteínas intrínsecamente desordenadas (IDP) son la resonancia magnética nuclear y la dispersión de rayos X de ángulo pequeño. Los datos recopilados a partir de estas técnicas se promedian en conjuntos y, por lo tanto, no dan una indicación clara de las conformaciones de proteínas individuales o con qué frecuencia ocurren. La microscopía de fuerza atómica, por otro lado, es capaz de obtener imágenes biológicas de resolución a nanoescala a alta velocidad, por lo que puede capturar la dinámica y las estructuras de las proteínas.
En este último trabajo, investigadores de la Universidad de Kanazawa junto con colaboradores en Japón, Francia e Italia aplicaron la técnica al estudio de varios desplazados internos e identificaron parámetros que definen la forma, tamaño y longitud de la cadena de las regiones proteicas, así como una ley de potencia que relaciona el tamaño de la proteína con la longitud de la proteína, y una descripción cuantitativa del efecto de la superficie de la mica sobre las dimensiones de las proteínas. La dinámica de las conformaciones de proteínas capturadas gracias a las capacidades de alta velocidad de la técnica reveló glóbulos que aparecen y desaparecen, y transformaciones entre conformaciones completamente desestructuradas y ligeramente plegadas en segmentos de hasta 160 aminoácidos de longitud.
Los estudios de la nucleoproteína del virus del sarampión en particular ayudaron a identificar no solo la forma y las dimensiones, sino también las características de las transiciones de orden-trastorno en la región responsable del reconocimiento molecular. lo que permite que los virus identifiquen los factores del huésped para que puedan reproducirse. También podrían determinar estructuras a mayor escala de la fosfoproteína del virus que no son accesibles a la resonancia magnética nuclear (que solo puede dar una indicación de las distancias entre los aminoácidos separados por menos de 2 nm). Los investigadores sugieren que la formación de ciertas formas compactas observadas puede explicar la resistencia a la proteólisis, es decir, la degradación de las proteínas.
En su informe del trabajo, los investigadores destacan que, además de ser una herramienta poderosa por derecho propio, "Cuando todas las características moleculares reveladas por HS-AFM se combinan con la estructura local plegada proporcionada por RMN, la información combinada permite una delimitación cuantitativa de los caracteres estructurales y dinámicos de los desplazados internos, de una manera más realista en comparación con las imágenes representadas individualmente, como se demostró para PNT [fosfoproteína del virus del sarampión] ".
Microscopía de fuerza atómica de alta velocidad
La microscopía de fuerza atómica se desarrolló en la década de 1980 y llevó la resolución a escala atómica lograda mediante la microscopía de túnel de barrido (que ganó el Premio Nobel de Física en 1986) a muestras no conductoras. Funciona usando un pequeño voladizo con una punta a nanoescala al final, que siente la superficie como una aguja de disco de vinilo o la golpea. Ya sea ajustando la altura de la punta o la frecuencia de resonancia del golpeteo, las interacciones entre la punta y la superficie proporcionan una señal que se puede utilizar para generar una imagen.
Si bien las imágenes AFM aportaron enormes beneficios a la investigación biológica, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.