
Un resumen gráfico del eje intestino-cerebro (izquierda) y la plataforma in vitro impresa en 3D (derecha). Crédito:Universidad de Maryland
Cualquiera que haya experimentado "mariposas en el estómago" antes de dar una gran presentación no se sorprenderá al saber que existe una conexión física real entre su intestino y su cerebro. Los neurocientíficos y los profesionales médicos llaman a esto el "eje intestino-cerebro" (GBA). Una mejor comprensión de la GBA podría conducir a tratamientos y curas para los trastornos neurológicos del estado de ánimo como la depresión y la ansiedad. así como para una variedad de enfermedades inflamatorias autoinmunes crónicas como el síndrome del intestino irritable (SII) y la artritis reumatoide (AR).
El problema es que hasta ahora las "mariposas" han sido todo lo que los médicos han tenido que trabajar para detectar estos trastornos relacionados con GBA. Incluso hoy en día, estas enfermedades se diagnostican principalmente por los propios informes de los pacientes sobre sus síntomas. Encontrar "biomarcadores" —medidas objetivas de una sustancia en el cuerpo que indica la presencia de un trastorno— podría mejorar drásticamente el diagnóstico y aliviar la enorme carga de los pacientes para identificar correctamente sus propios síntomas.
Los científicos sospechan que el neurotransmisor químico serotonina es el biomarcador de una variedad de trastornos de GBA. La serotonina estimula la acción del sistema nervioso a través del nervio vago, el conector físico entre el cerebro y el colon. Generado en lo profundo del revestimiento del intestino, en última instancia, la serotonina influye en todo, desde el estado de ánimo y las emociones hasta el sueño, digestión y secreción de hormonas. Su producción se ve afectada de alguna manera por el "microbioma" bacteriano presente en este entorno. Los investigadores esperan que la creación de herramientas para analizar la producción y la disfunción de la serotonina en el microbioma intestinal ayude a descubrir los misterios de los trastornos relacionados con GBA.
Con $ 1 millón en fondos de la National Science Foundation, Ingenieros de la Universidad de Maryland (UMD), neurocientíficos, Los microbiólogos y físicos han logrado un progreso significativo en el desarrollo de una plataforma que puede monitorear y modelar el procesamiento en tiempo real de la actividad de la serotonina del microbioma intestinal. Su objetivo es empaquetar algún día la plataforma en una cápsula ingerible capaz de detectar, tratamiento y seguimiento de las enfermedades de GBA.
La convergencia de disciplinas es clave, dice el profesor Reza Ghodssi, investigador principal del proyecto. "Estamos convergiendo en neurociencia, señalización molecular, y micro-nano dispositivos y sistemas. Esto nos permite medir e investigar datos en la interfaz de cada unión de una plataforma GBA simulada:celda a celda, célula a molécula, molécula a nervio, y desarrollar metodologías de ingeniería para analizarlo e interpretarlo ".
El trabajo se basa en la experiencia en dispositivos médicos ingeribles que se ha desarrollado en el Laboratorio de Sensores y Actuadores MEMS de UMD. el Departamento de Bioingeniería de Fischell, y la Iniciativa Cerebro y Comportamiento.
Tres nuevos artículos publicados detallan el progreso en la detección de serotonina, evaluar sus efectos neurológicos, y detectar cambios mínimos en el epitelio intestinal.
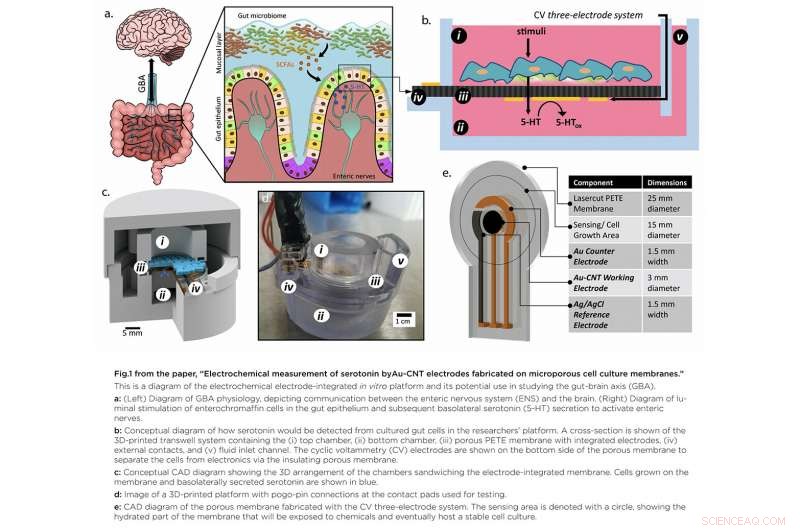
En "Medición electroquímica de serotonina mediante electrodos de Au-CNT fabricados en membranas de cultivo celular poroso, "el equipo desarrolló una plataforma que brinda acceso al sitio específico de producción de serotonina, importante porque la serotonina se secreta desde el fondo de las células. Una innovadora membrana porosa con un sensor de serotonina integrado en el que se puede cultivar un modelo del revestimiento intestinal permitió a los investigadores acceder a los lados superior e inferior del cultivo celular.
El documento fue publicado en línea el 7 de septiembre de 2020 en el Naturaleza diario, Microsistemas y Nanoingeniería. Los autores son Ph.D. en Bioingeniería. estudiante Ashley Chapin, ex investigador postdoctoral de ISR Pradeep Ramiah Rajasekaran, ex alumno David N. Quan (BioE Ph.D.2015), Profesor Liangbing Hu (MSE / MEII), Profesor asociado Jens Herberholz (Psicología / NACS), Profesor William Bentley (BioE / Fischell Institute / IBBR), y el profesor Reza Ghodssi (ECE / ISR).
Crédito:Universidad de Maryland
Usando deposición de metal, diseñaron un sistema de tres electrodos directamente sobre una membrana de cultivo celular porosa suspendida dentro de una carcasa personalizada impresa en 3D. Las células se pueden cultivar en la parte superior de la membrana con el sensor de serotonina orientado hacia la parte inferior para una detección directa. Luego, el equipo mejoró la sensibilidad de la detección de serotonina al aumentar el área de superficie efectiva del electrodo, lanzar gota a gota un pequeño volumen de nanotubos de carbono sobre la superficie del electrodo. Las soluciones preparadas de serotonina se detectaron bien dentro del rango de concentración fisiológica esperado.
El trabajo es el primero en demostrar un método factible para detectar moléculas redox, como la serotonina, directamente sobre un sustrato de cultivo celular poroso y flexible. Otorga un acceso superior a las moléculas liberadas por las células y crea un entorno intestinal modelo controlable sin recurrir a procedimientos invasivos en humanos o animales.
El segundo artículo del equipo, "Un sistema de biomonitoreo híbrido para la comunicación intestino-neurona, "se basa en los hallazgos del primero:los investigadores desarrollaron aún más la plataforma de medición de serotonina para que pudiera evaluar los efectos neurológicos de la serotonina. Al agregar e integrar un modelo de nervio de cangrejo de río disecado con el modelo de revestimiento intestinal, El equipo creó una interfaz intestino-neurona que puede evaluar electrofisiológicamente la respuesta nerviosa a la serotonina detectada electroquímicamente. Este avance permite el estudio de la señalización molecular entre el intestino y las células nerviosas, haciendo posible la monitorización en tiempo real de ambos tejidos GBA por primera vez.
El documento se publicó en línea en junio de 2020. Revista IEEE de sistemas microelectromecánicos . Fue escrito por Chapin, Doctorado en Ingeniería Eléctrica e Informática. estudiante Jinjing Han Doctorado en Neurociencia y Ciencias Cognitivas. estudiante Ta-Wen Ho Herberholz y Ghodssi.
Finalmente, el concepto, El diseño y uso de toda la plataforma de biomonitoreo se describe en un tercer artículo. "Sistemas Transwell integrados con sensor electroquímico impreso en 3-D, "publicado en línea el 5 de octubre de 2020 en el Naturaleza diario Microsistemas y nanoingeniería . El documento fue escrito por Rajasekaran, Chapin, Quan, Herberholz, Bentley and Ghodssi.
This paper delves into the development of the 3-D-printed housing, the maintenance of a healthy lab-on-a-chip gut cell culture, and the evaluation of the two types of sensors integrated on the cell culture membrane. The dual sensors are particularly important because they provide feedback about multiple components of the system—namely, the portions that model the gut lining's permeability (a strong indicator of disease) and its serotonin release (a measure of communication with the nervous system). Alongside the electrochemical sensor—evaluated using a standard redox molecule ferrocene dimethanol—an impedance sensor was used to monitor cell growth and coverage over the membrane. Using both these sensors would allow monitoring of a gut cell culture under various environmental and dietary conditions. It also would enable researchers to evaluate changes to barrier permeability (a strong indicator of disease), and serotonin release (a measure of communication with the nervous system).
"These works represent a big step forward in our understanding of the gut/brain axis, " says Cornell University's John March, Chair of the Department of Biological and Environmental Engineering. "One of the limitations of this field is the inability to perform highly controlled experiments in a 'close to in vivo' system. These papers provide ways around this problem with simple, elegant experiments that are highly accessible. I expect these will be used frequently."
Because the engineering aspects of the platform are well underway, the researchers are working towards culturing multi-tissue interfaces with the help of Jay Pasricha and Subhash Kulkarni at Johns Hopkins University. Eventually multiple platforms will be created, each colonized with a different combination of gut bacteria, to measure the neurophysiological effects of serotonin production in varying microbiome environments.
With this information, Professor Wolfgang Losert (Physics/IPST/IREAP) will lead a machine learning effort to process the sensor data through a computer model that can simulate the outcomes of the different microbiomes. This will provide the clearest picture yet of how a system as complex and individually unique as the gut microbiome affects both gut and brain health. It also may help researchers better understand the connection between nutrition and mental health.
"Understanding biology at the level of whole organisms is a frontier in biology, and essential to forming a basis for precision medicine, " says the University of California, Berkeley's Amy Herr, the John D. &Catherine T. MacArthur Professor of Bioengineering. "By harnessing hallmarks of engineering—integrated, systems-level design—the new research from the Ghodssi-Bentley-Herberholz team presents an integrated approach to elegantly perturb and then probe the electrons and molecules that are key conduits of information flow in whole organisms."