
Aplicación in vitro de ensayo utilizando múltiples tipos de células con simulación y tasa de extracción. (A) Datos de fluorescencia y configuración del ensayo que representan la fluorescencia QD (QSH) sin lavar para pocillos sin lavar que contienen riñón (MES13), hígado (AML12), endotelial (C166), y tipos de células de macrófagos (J774a.1). AU, unidades arbitrarias. (B) Concentraciones de captación de fluorescencia lavada cruda (negra) y calibrada (roja) (círculos, medido) en comparación con los ajustes de simulación (líneas) para el hígado, endotelial macrófagos y tipos de células renales. (C) Resumen de las constantes de velocidad optimizadas por el algoritmo genético para el ajuste del modelo a los conjuntos de datos medidos. (D) Imágenes de microscopía confocal dependientes del tiempo de la captación de QD, mostrando la saturación (flechas de doble cara) para cada tipo de célula en el estudio (el azul y el rojo representan el núcleo celular Hoechst y fluorescencia QSH, respectivamente). La caricatura es una representación visual de la cinética de interacción de la célula QD. Crédito: Avances de la ciencia , doi:10.1126 / sciadv.aax2642
Los bioingenieros pueden diseñar medicamentos inteligentes para terapias basadas en anticuerpos y nanomateriales con el fin de optimizar la eficiencia de los medicamentos para que sean cada vez más eficientes. Ensayos preclínicos en etapa inicial. El fármaco ideal tendrá la máxima eficacia en los sitios de tejido diana para el transporte desde la vasculatura del tejido al entorno celular. Los investigadores pueden usar simulaciones biológicas acopladas a enfoques in vitro para predecir su exposición de manera rápida y eficiente para predecir la biodistribución del fármaco dentro de células individuales de tejido animal vivo sin depender de estudios en animales. En un nuevo estudio ahora publicado en Avances de la ciencia , Edward Price y Andre J. Gesquiere utilizaron con éxito un ensayo in vitro y un modelo de dinámica de fluidos computacional (CFD) para traducir la cinética celular in vitro en simulaciones de cuerpo completo en múltiples especies y tipos de nanomateriales. El trabajo les permitió predecir la distribución de medicamentos dentro de las células de los tejidos individuales y el equipo espera que este trabajo se perfeccione. reducir y reemplazar la experimentación con animales al tiempo que brinda a los científicos una nueva perspectiva sobre el desarrollo de fármacos.
Las nanomedicinas (NM) en forma de anticuerpos y nanomateriales sintéticos pueden complementar la medicina convencional de moléculas pequeñas a través de la selección activa de tejidos. la estabilidad y los plazos de circulación variables, acoplado a biodistribución ajustable. Los equipos de investigación dependen en gran medida de modelos animales para cuantificar las dosis administradas, planteando preguntas sobre ética y aumentos repentinos de tiempo y costo. Desde un punto de vista científico, La destrucción de la arquitectura tisular llevada a cabo durante la homogeneización tisular para la cuantificación puede eliminar el conocimiento crítico del transporte de nanomedicina dentro de las células tisulares y la vasculatura. Un fármaco puede llegar a las células exhibiendo una farmacocinética plasmática óptima para alcanzar la vasculatura tisular. La arquitectura y el diámetro de las nanopartículas permiten el transporte crítico del fármaco a través de las paredes de los microvasos hacia un entorno celular de tejido normal o infectado.
En el presente trabajo, Price y Gesquiere combinaron un enfoque de simulación in vitro / in silico para predecir la biodistribución de NM dentro de especies preclínicas (modelos animales y celulares) a nivel de una sola célula. Tenían en cuenta las diferencias en el tamaño de las nanopartículas, especies animales y diámetros del tamaño de los poros de la vasculatura utilizando un modelo fluidodinámico. El equipo cuantificó el contenido de NM celular en situaciones en las que los resultados eran difíciles de traducir a estudios con animales debido a la falta de perspectivas sistemáticas. También abordaron problemas experimentales existentes en los que las células inducían la degradación de la fluorescencia de las nanopartículas para producir falsos negativos.
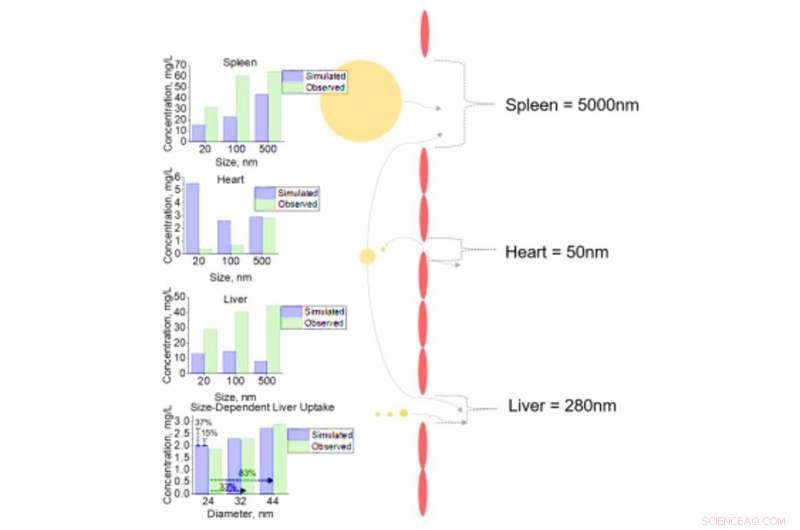
Análisis celular de la captación de NM en diferentes tejidos para NM con diferentes diámetros. Captación celular de NM con diámetros de 20, 100, y 500 nm para el bazo, corazón, e hígado con diámetros de fenestra de 5000, 50, y 280 nm respectivamente. Análisis de sensibilidad de los cambios incrementales en el diámetro NM (24, 32, y 44 nm) con la subsiguiente captación de células hepáticas. Todas las salidas simuladas (predichas) se comparan con los datos observados de los conjuntos de datos celulares de la literatura. Crédito: Avances de la ciencia , doi:10.1126 / sciadv.aax2642
Para validar su trabajo in vitro, el equipo simuló el análisis lisosómico acoplado a la espectroscopia de absorción atómica (AAS), y simulaciones in vivo fundamentadas comparando los resultados con la literatura publicada de datos de animales de cuerpo entero para ratas, ratones y primates no humanos. Los científicos utilizaron puntos cuánticos (QD) como sistema modelo debido a su potencial en la detección de nanopartículas. considerando también sus límites. Price y col. contó las complejidades en una plataforma de NM para extender las capacidades a nanopartículas basadas en anticuerpos y metal / polímero.
Cuando una nanopartícula circula a través del suministro de sangre animal para ingresar al entorno tisular, interactuará con las células de los tejidos a través de la adsorción, desorción procesos de internalización o captación activa. Los científicos expusieron nanopartículas a QD (puntos cuánticos) no tóxicos para detectar de manera fluorescente sus interacciones con las células que comúnmente encuentran los NM como los macrófagos, células endoteliales y células epiteliales, después de la inyección intravenosa. El equipo de investigación cuantificó la concentración de nanopartículas que interactúan con las células in vitro y construyó simulaciones de cinética celular para evaluar la cinética de velocidad y las interacciones entre células NM.
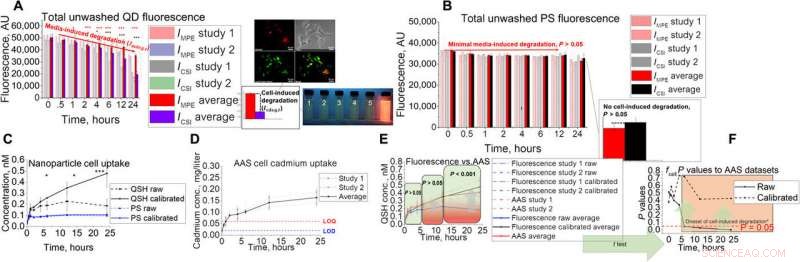
Desarrollo y validación de ensayos in vitro para AAS. (A y B) Configuración de ensayo in vitro con su señal de fluorescencia sin lavar acoplada para (A) QD y (B) PS. Las imágenes muestran la colocalización de Hepa1-6 de QD en compartimentos lisosomales, así como instantáneas de fluorescencia bajo iluminación ultravioleta en condiciones de exposición lisosomal simuladas con pH 2,5, 3,0, 3,5, 4.0, 4.5, y 7,4 (1 a 6, respectivamente). (C a E) Estudios de captación QD utilizando la técnica in vitro con (C) fluorescencia, (D) AAS, (E) combinado, y los resultados estadísticos (F) usando la prueba t de dos colas al comparar la absorción de fluorescencia sin procesar y calibrada con los datos de AAS. Los asteriscos en la figura representan significancia en el * (P <0.05), ** (P <0,01), y niveles *** (P <0,001). Crédito: Avances de la ciencia , doi:10.1126 / sciadv.aax2642
La simulación contenía un (i) medio, (ii) la membrana celular y (iii) los compartimentos del espacio celular interconectados mediante ecuaciones básicas de transferencia de masa y constantes de velocidad de primer orden. Optimizaron el sistema utilizando un algoritmo genético en MATLAB y observaron que la constante de tasa de adsorción de membrana era más alta para los macrófagos y más baja para las células endoteliales y hepáticas. De los tipos de células, el proceso de internalización también fue relativamente más alto para las células del tejido hepático y más bajo para las células endoteliales. Las imágenes confocales respaldaron visualmente estos datos con una rápida captación y saturación de macrófagos dentro de una hora de exposición. mientras que las células de tejido epitelial y endotelial se retrasaron aún más.
El equipo demostró la presencia de factores de degradación celular en ensayos in vitro utilizando simulaciones de cinética celular para ayudar a traducir de manera eficiente los datos in vitro a estudios con animales. La espectroscopia de adsorción atómica (AAS) validó los resultados de la captación de QD mediante ensayos de fluorescencia y destacó la importancia de calcular la degradación inducida por células y medios. Dado que las nanomedicinas (NM) ubicadas dentro de los tejidos pueden acumularse en los intersticios, vasculatura o dentro de una variedad de tipos de células, el equipo tradujo la cinética celular in vitro a simulaciones animales in silico para dar cuenta de esto. Llevaron a cabo dos estudios piloto y evaluaron las tendencias en la absorción de tejido completo en función del tamaño y la absorción cuantificada de NM a nivel de una sola célula. Arquitectónicamente, cada simulación de compartimento de tejido contenía cuatro subcompartimentos para representar el epitelio, células endoteliales y macrófagos.
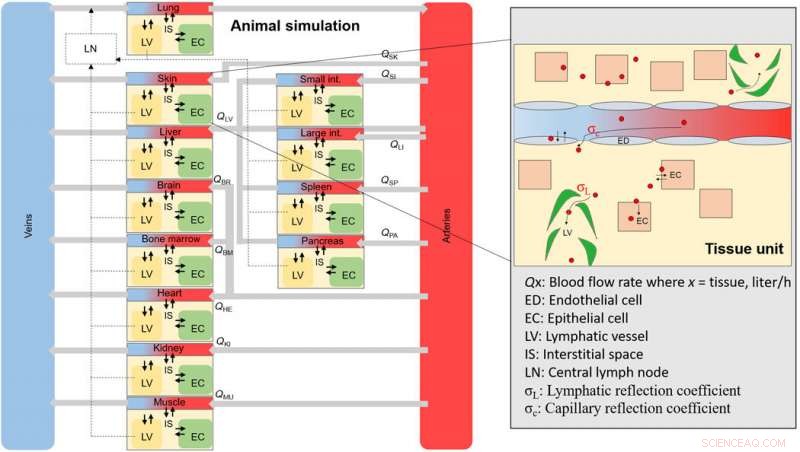
Un esquema general de la simulación de NM animal. Esquema de los compartimentos generales de tejido y sangre que componen el animal completo. Cada compartimento de tejido está interconectado a través de tasas de flujo sanguíneo y contiene subcompartimentos (área ampliada), donde un NM se distribuirá tras la internalización a través de las fenestras endoteliales. Crédito: Avances de la ciencia , doi:10.1126 / sciadv.aax2642
Dado que el hígado y el bazo son objetivos comunes para la acumulación de NM, utilizaron estos tejidos como estudios de caso para comprender y capturar, la sensibilidad y precisión de la simulación. Por ejemplo, La simulación mostró que el aumento de los tamaños de NM excedía el tamaño de los poros del tejido de la mayoría de los tipos de tejido para canalizarse hacia tejidos con tamaños de poro más grandes, como el hígado. En este punto, las células endoteliales del hígado interactuaron con los NM para una rápida unión y saturación de la membrana en 24 horas. Cuando el NM entró en los intersticios del tejido, interactuó rápidamente con las células del tejido epitelial (para el hígado) y macrófago (para el bazo). Los resultados generaron confianza en el poder predictivo de la simulación y su capacidad para predecir el contenido de fármaco de tamaño variable a nivel de una sola célula para animales por primera vez, estrictamente basándose únicamente en datos in vitro.
Luego, los científicos indicaron el poder predictivo y la capacidad de traslación de la simulación comparando con el contenido medido a nivel de tejido para múltiples tipos y especies de NM. incluyendo ratas, ratones y contenido celular (epitelial, endotelial y macrófago). Probaron el rendimiento predictivo en múltiples variables basadas en las pautas y estándares de la Organización Mundial de la Salud del consorcio académico y farmacéutico para el desarrollo y la seguridad de medicamentos. Los promedios pronosticados por el modelo y los promedios observados se correlacionaron linealmente para indicar predicciones razonables del modelo en todas las especies y tipos de NM.
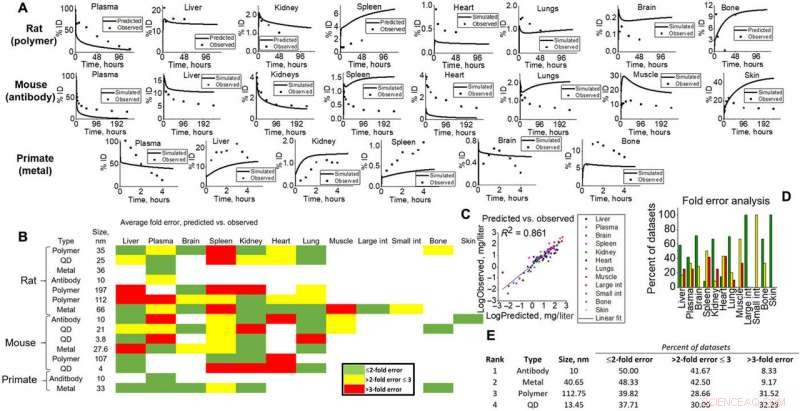
Salidas de simulación para validación de varios estudios con animales. (A) Ejemplos de predicción (línea) a nivel de tejido frente a (puntos) observados para todos los tipos de especies (ratas, ratones, y NHP) para evidencia visual de las capacidades del modelo. (B) Mapa de calor del análisis de error de pliegue calculado para todo el tejido, NUEVO MÉJICO, y tipos de especies en simulación. Verde, amarillo, y el rojo representan <2 veces, <3 veces, y> errores triples. El error de pliegue se calculó de acuerdo con las ecuaciones dadas en Materiales y métodos, donde se compararon los promedios de conjuntos de datos simulados con los observados. (C) Todos los promedios de puntos de datos específicos de los tipos de tejido para las simulaciones y observados se representaron entre sí para producir un ajuste lineal (R2 =0,861). (D) Se muestran errores de pliegue específicos de tejido para visualizar la precisión del modelo para cada tejido en este estudio. (E) Las salidas simuladas por NM se clasificaron (1 a 4) de acuerdo con el análisis de error de veces. Crédito: Avances de la ciencia , doi:10.1126 / sciadv.aax2642
De este modo, Edward Price y Andre J. Gesquiere desarrollaron una plataforma viable para reducir y refinar las pruebas con animales durante el desarrollo de la nanomedicina (NM). Los datos concentrados in vitro acoplados a simulaciones de cinética celular proporcionaron interacciones célula-NM para mostrar un excelente potencial de traducción. Los resultados no requirieron ajustarse a conjuntos de datos de biodistribución animal, ya que todos los parámetros se optimizaron para datos in vitro o se calcularon utilizando un modelo fluidodinámico. El proceso también funcionó en condiciones de degradación de NM en el entorno biológico. Las técnicas combinadas in vitro e in silico ayudarán al futuro diseño de fármacos inteligentes para ayudar a los científicos a formar debates mejor e informados al tiempo que se reducen las pruebas con animales vivos. Price y Gesquiere esperan que este trabajo sirva como un nuevo enfoque para las simulaciones predictivas del transporte de nanomedicina.
© 2020 Science X Network