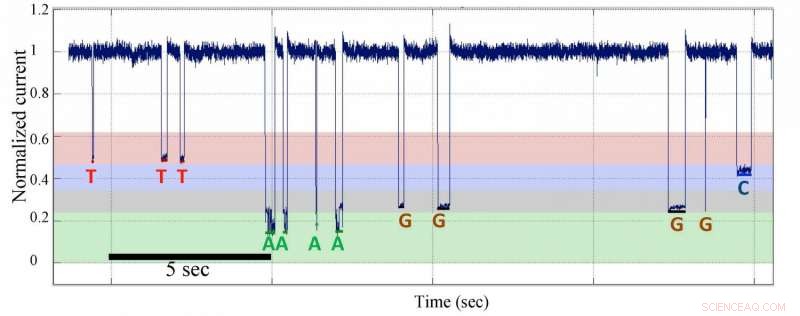
Secuenciación de ADN de nanoporos de una sola molécula mediante la síntesis de datos a partir de una plantilla con secuencias de homopolímeros. Crédito:Jingyue Ju, Ingeniería de Columbia
Investigadores de la Universidad de Columbia, con colegas de Genia Technologies (Roche), La Universidad de Harvard y el Instituto Nacional de Estándares y Tecnología (NIST) informan que lograron la secuenciación electrónica de ADN de una sola molécula en tiempo real con una resolución de base única utilizando una matriz de nanoporos de proteínas.
La secuenciación del ADN es la tecnología clave para las iniciativas de medicina personalizada y de precisión, permitiendo descubrimientos rápidos en la ciencia biomédica. La secuencia completa del genoma de un individuo proporciona marcadores y pautas importantes para el diagnóstico médico, cuidado de la salud, y mantener una vida sana. Hasta la fecha, el costo y la velocidad involucrados en la obtención de secuencias de ADN de alta precisión ha sido un gran desafío. Si bien se han realizado varios avances durante la última década, Los instrumentos de secuenciación de alto rendimiento ampliamente utilizados en la actualidad dependen de la óptica para la detección de cuatro bloques de construcción de ADN:A, C, G y T. Para explorar capacidades de medición alternativas, Se ha desarrollado la secuenciación electrónica de un conjunto de plantillas de ADN para análisis genético. Secuenciación de cadenas de nanoporos, en el que un ADN de una sola hebra se enhebra a través de los poros a nanoescala bajo un voltaje eléctrico aplicado para producir señales electrónicas para la determinación de la secuencia a nivel de una sola molécula, se ha desarrollado recientemente; sin embargo, porque los cuatro nucleótidos son muy similares en sus estructuras químicas, no se pueden distinguir fácilmente con este método. Por lo tanto, los investigadores están persiguiendo activamente la investigación y el desarrollo de una plataforma de secuenciación electrónica de ADN de una sola molécula precisa, ya que tiene el potencial de producir un secuenciador de ADN miniaturizado capaz de descifrar el genoma para facilitar la medicina de precisión personalizada.
Un equipo de investigadores de Columbia Engineering, encabezada por Jingyue Ju (Samuel Ruben-Peter G. Viele Profesor de Ingeniería, Catedrático de Ingeniería Química y Farmacología, Director del Centro de Tecnología Genómica e Ingeniería Biomolecular), con colegas de la Facultad de Medicina de Harvard, dirigido por George Church (profesor de genética); Genia Technologies, dirigido por Stefan Roever (CEO de Genia); y John Kasianowicz, el investigador principal del NIST, han desarrollado un sistema completo para secuenciar el ADN en nanoporos electrónicamente a nivel de molécula única con resolución de base única. Este trabajo, intitulado, "Secuenciación electrónica de ADN de una sola molécula en tiempo real mediante síntesis utilizando nucleótidos marcados con polímeros en una matriz de nanoporos, "se publica en la revista, procedimientos de la Academia Nacional de Ciencias ( PNAS ) Edición temprana.
Previamente, investigadores de los laboratorios de Ju en Columbia y Kasianowicz en NIST informaron sobre el principio general de secuenciación de nanoporos por síntesis (SBS), la viabilidad del diseño y la síntesis de nucleótidos marcados con polímeros como sustratos para la ADN polimerasa, la detección y la diferenciación de las etiquetas de polímero por nanoporo a nivel de molécula única [ Informes científicos 2, 684 (2012) DOI:10.1038 / srep00684]. La corriente PNAS El artículo describe la construcción del sistema SBS de nanoporos completo para producir datos de secuenciación electrónica de una sola molécula con resolución de base única. Esta estrategia de SBS distingue con precisión cuatro bases de ADN al detectar y diferenciar electrónicamente cuatro etiquetas de polímero diferentes unidas al fosfato 5'de los nucleótidos durante su incorporación en una cadena de ADN en crecimiento catalizada por la polimerasa. una enzima sintetizadora de ADN. Los investigadores diseñaron y sintetizaron nuevos nucleótidos marcados en el fosfato terminal con polímeros basados en oligonucleótidos para realizar SBS nanoporo en una plataforma de matriz de nanoporos de proteína α-hemolisina. Las etiquetas de los nucleótidos marcados con polímero, que se verificó que eran sustratos activos para la ADN polimerasa, Producen diferentes niveles de bloqueo de corriente eléctrica. Construyeron una matriz de nanoporos en un chip electrónico con múltiples electrodos; la matriz está compuesta por canales de proteínas que se acoplaron a una ADN polimerasa que se unió a una plantilla de ADN cebada. La adición de distintos nucleótidos etiquetados con polímeros de diseño personalizado a la matriz de nanoporos desencadena la síntesis de ADN. Al bloquear la corriente iónica del canal a diferentes niveles, las distintas etiquetas proporcionan una lectura de la secuencia de la plantilla en tiempo real con resolución de base única.
Como Carl Fuller, autor principal, Investigador científico senior adjunto en el Laboratorio Ju del Departamento de Ingeniería Química en Columbia y Director de Química en Genia, Señala, "La novedad de nuestro enfoque SBS nanopore comienza con el diseño, síntesis, y selección de cuatro nucleótidos etiquetados con polímero diferentes. Usamos una ADN polimerasa unida covalentemente al nanoporo y los nucleótidos marcados para realizar SBS. Durante la replicación del ADN unido a la polimerasa, la etiqueta de cada nucleótido complementario se captura en el poro para producir una señal eléctrica única. Para la determinación de la secuencia se utilizan cuatro etiquetas de polímero distintas que producen firmas distintas que son reconocidas por el detector electrónico en el chip de matriz de nanoporos. Por lo tanto, Las secuencias de ADN se obtienen para muchas moléculas individuales en paralelo y en tiempo real. Las cuatro etiquetas de polímero están diseñadas para ofrecer distinciones mucho mejores entre ellas, en contraste con las pequeñas diferencias entre los cuatro nucleótidos de ADN nativo, superando así el gran desafío al que se enfrentan otros métodos directos de secuenciación de nanoporos ". las etiquetas se pueden optimizar aún más con respecto al tamaño, cargar, y estructura para proporcionar una resolución óptima en el sistema SBS nanopore.
"Este emocionante proyecto reúne a científicos e ingenieros de la academia y la industria con experiencia combinada en ingeniería molecular, nanotecnología, genómica, electrónica y ciencia de datos para producir plataformas de diagnóstico genético rentables con un potencial sin precedentes para la medicina de precisión, ", dice Ju." Estamos muy agradecidos por el generoso apoyo de los NIH que nos permitió hacer un rápido progreso en la investigación y el desarrollo de la tecnología nanopore SBS, y las destacadas contribuciones de todos los miembros de nuestro consorcio de investigación ".
Según Ju, los investigadores ya han ido más allá de lo demostrado en el PNAS estudio donde los datos de secuenciación se obtuvieron en un secuenciador prototipo temprano basado en nanopore SBS. El rendimiento y el rendimiento del secuenciador actual ha progresado más allá de lo que se informó en el PNAS papel. Recientemente se ha logrado la viabilidad de alcanzar longitudes de lectura de más de 1000 bases de ADN. Avanzando, el equipo de investigación colaborativa seguirá optimizando las etiquetas modificando los enlazadores, estructura, y cargar a nivel molecular, y el ajuste fino de la polimerasa y la electrónica para el sistema SBS de nanoporos con el objetivo de secuenciar con precisión un genoma humano completo rápidamente y a bajo costo, lo que permite su uso en diagnósticos médicos de rutina.