
SINGLE utiliza imágenes TEM in situ de nanocristales de platino que giran libremente en una celda de grafeno líquido para determinar las estructuras tridimensionales de nanopartículas coloidales individuales. Crédito:Berkeley Lab
Así como las proteínas son uno de los componentes básicos de la biología, Las nanopartículas pueden servir como bloques de construcción básicos para materiales de próxima generación. De acuerdo con este paralelismo entre biología y nanotecnología, Se ha adaptado una técnica probada para determinar las estructuras tridimensionales de proteínas individuales para determinar las estructuras 3D de nanopartículas individuales en solución.
Un equipo multiinstitucional de investigadores dirigido por el Laboratorio Nacional Lawrence Berkeley (Berkeley Lab) del Departamento de Energía de EE. UU. (DOE), ha desarrollado una nueva técnica denominada "SINGLE" que proporciona las primeras imágenes a escala atómica de nanopartículas coloidales. SOLTERO, que significa Identificación de estructuras 3D de nanopartículas mediante microscopía electrónica de células líquidas de grafeno, se ha utilizado para reconstruir por separado las estructuras 3D de dos nanopartículas de platino individuales en solución.
"Es necesario comprender los detalles estructurales de las nanopartículas coloidales para unir nuestro conocimiento sobre su síntesis, mecanismos de crecimiento, y propiedades físicas para facilitar su aplicación a las energías renovables, catálisis y muchos otros campos, "dice el director de Berkeley Lab y renombrado autoridad en nanociencia Paul Alivisatos, quien dirigió esta investigación. "Mientras que la mayoría de los estudios estructurales de las nanopartículas coloidales se realizan en el vacío después de que se completa el crecimiento de los cristales, nuestro método SINGLE nos permite determinar su estructura 3D en una solución, un paso importante para mejorar el diseño de nanopartículas para catálisis y aplicaciones de investigación energética ".
Alivisatos, quien también ocupa la Cátedra Distinguida de Samsung en Nanociencia y Nanotecnología en la Universidad de California Berkeley, y dirige el Instituto Kavli Energy NanoSciences en Berkeley (Kavli ENSI), es el autor correspondiente de un artículo que detalla esta investigación en la revista Ciencias . El artículo se titula "Estructura 3D de nanocristales individuales en solución por microscopía electrónica". Los coautores principales son Jungwon Park de la Universidad de Harvard, Hans Elmlund de la Universidad Monash de Australia, y Peter Ercius de Berkeley Lab. Otros coautores son Jong Min Yuk, David Limmer, Qian Chen, Kwanpyo Kim, Sang Hoon Han, David Weitz y Alex Zettl.
Las nanopartículas coloidales son grupos de cientos a miles de átomos suspendidos en una solución cuyas propiedades químicas y físicas colectivas están determinadas por el tamaño y la forma de las nanopartículas individuales. Las técnicas de imágenes que se utilizan habitualmente para analizar la estructura 3D de cristales individuales en un material no se pueden aplicar a nanomateriales suspendidos porque las partículas individuales en una solución no son estáticas. La funcionalidad de las proteínas también está determinada por su tamaño y forma, y los científicos que querían obtener imágenes de estructuras de proteínas en 3D se enfrentaron a un problema similar. El problema de la obtención de imágenes de proteínas se resolvió mediante una técnica llamada "microscopía crioelectrónica de una sola partícula, "en el que se registran decenas de miles de imágenes de microscopio electrónico de transmisión 2D (TEM) de copias idénticas de una proteína individual o complejo de proteínas congeladas en orientaciones aleatorias y luego se combinan computacionalmente en reconstrucciones 3D de alta resolución. Alivisatos y sus colegas utilizaron este concepto para crear su técnica ÚNICA.
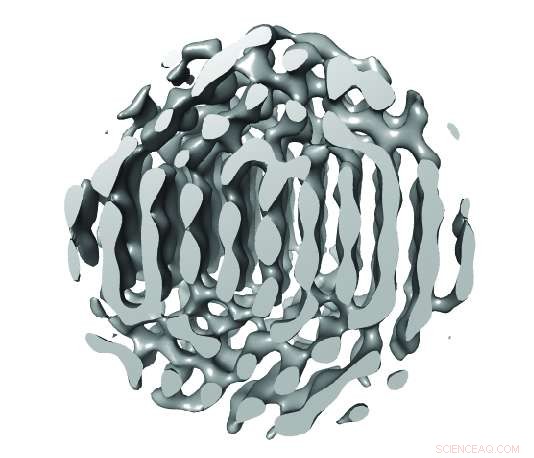
Una losa a través de la reconstrucción tridimensional de la partícula 1 a lo largo del plano vertical con las posiciones atómicas tentativas indicadas. Las repeticiones ABC de {111} planos son visibles. Crédito:Centro de Excelencia ARC en Imagen Molecular Avanzada
"En ciencia de materiales, no podemos asumir que las nanopartículas en una solución son todas idénticas, por lo que necesitábamos desarrollar un enfoque híbrido para reconstruir las estructuras 3D de nanopartículas individuales, "dice el coautor principal del Ciencias papel Peter Ercius, un científico del personal del Centro Nacional de Microscopía Electrónica (NCEM) en Molecular Foundry, una instalación para usuarios de la Oficina de Ciencias del DOE.
"SINGLE representa una combinación de tres avances tecnológicos de las imágenes TEM en la ciencia biológica y de los materiales, Ercius dice. Estos tres avances son el desarrollo de una celda de grafeno líquido que permite la obtención de imágenes TEM de nanopartículas que giran libremente en la solución. detectores de electrones directos que pueden producir películas con milisegundos de resolución de tiempo de fotograma a fotograma de los nanocristales giratorios, y una teoría para la reconstrucción 3D ab initio de una sola partícula ".
La celda de grafeno líquido (GLC) que ayudó a hacer posible este estudio también se desarrolló en Berkeley Lab bajo el liderazgo de Alivisatos. Las imágenes TEM utilizan un haz de electrones en lugar de luz para la iluminación y el aumento, pero solo se pueden usar en un alto vacío porque las moléculas en el aire interrumpen el haz de electrones. Dado que los líquidos se evaporan en alto vacío, las muestras en soluciones deben sellarse herméticamente en recipientes sólidos especiales, llamados celdas, con una ventana de visualización muy delgada antes de obtener imágenes con TEM. En el pasado, Las celdas líquidas presentaban ventanas de visualización basadas en silicio cuyo grosor limitaba la resolución y perturbaba el estado natural de los materiales de muestra. El GLC desarrollado en el laboratorio de Berkeley presenta una ventana de visualización hecha de una hoja de grafeno que tiene solo un átomo de espesor.
"El GLC nos proporciona una cubierta ultrafina de nuestras nanopartículas mientras mantiene las condiciones líquidas en el vacío TEM, "Dice Ercius." Dado que la superficie de grafeno del GLC es inerte, no adsorbe ni perturba el estado natural de nuestras nanopartículas ".
Trabajando en el TEAM I de NCEM, el microscopio electrónico más potente del mundo, Ercio, Alivisatos y sus colegas pudieron obtener imágenes in situ de los movimientos de traslación y rotación de nanopartículas individuales de platino que tenían menos de dos nanómetros de diámetro. Se eligieron nanopartículas de platino debido a su alta fuerza de dispersión de electrones y porque su estructura atómica detallada es importante para la catálisis.
"Nuestros estudios anteriores de GLC de nanocristales de platino mostraron que crecen por agregación, resultando en estructuras complejas que no son posibles de determinar por ningún método desarrollado previamente, ", Dice Ercius." Dado que SINGLE deriva sus estructuras 3D a partir de imágenes de nanopartículas individuales que giran libremente en la solución, permite el análisis de poblaciones heterogéneas de nanopartículas potencialmente desordenadas que se sintetizan en solución, proporcionando así un medio para comprender la estructura y estabilidad de los defectos a nanoescala ".
El siguiente paso para SINGLE es recuperar un mapa de densidad de resolución atómica 3D completo de nanopartículas coloidales utilizando una cámara más avanzada instalada en TEAM I que puede proporcionar 400 cuadros por segundo y una mejor calidad de imagen.
"Planeamos obtener imágenes de defectos en nanopartículas hechas de diferentes materiales, partículas de la cáscara del núcleo, y también aleaciones hechas de dos especies atómicas diferentes, "Dice Ercius.