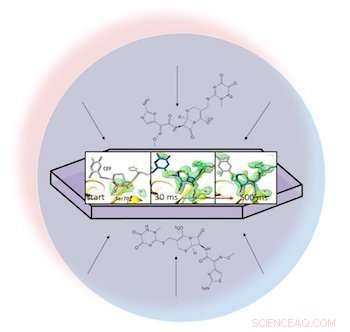
En una gota de líquido (representada por el círculo azul claro), un antibiótico se dispersa a través de una proteína cristalizada, beta-lactamasa, y se une a él. Las instantáneas de rayos X del par unido permitieron a los investigadores crear mapas de la densidad de electrones (verde) y observar un anillo en el antibiótico abrirse (extremo derecho) 30 a 500 milisegundos después de que comenzara la reacción. Crédito:Marius Schmidt / Universidad de Wisconsin Milwaukee
Los científicos de la Universidad de Rice utilizaron un láser de rayos X de pulsación rápida para mostrar cómo las bacterias de la tuberculosis resistentes a los medicamentos desactivan las moléculas de antibióticos destinadas a tratar la enfermedad pulmonar mortal.
El bioquímico de Rice, George Phillips, y el estudiante de posgrado y coautor principal, José Olmos, son parte del Centro BioXFEL, respaldado por la Fundación Nacional de Ciencias, que capturó los innovadores resultados de prueba de principios a través de una técnica llamada cristalografía en serie de mezcla e inyección.
Para hacerlo, se requirió el uso de una nueva herramienta, un láser de rayos X de electrones libres (XFEL) que promete una mejora seria al meticuloso, proceso centenario de caracterización de moléculas mediante espectroscopia de rayos X. El láser se encuentra en el Laboratorio Nacional Acelerador SLAC del Departamento de Energía de la Universidad de Stanford.
Definir estructuras moleculares es clave para comprender cómo funcionan, Phillips dijo. El nuevo descubrimiento demuestra el rápido desarrollo de la capacidad de los científicos para tomar instantáneas de los procesos biológicos dinámicos a medida que ocurren.
Fiel a su nombre, la técnica de mezclar e inyectar alimenta al láser una corriente estrecha de moléculas cristalizadas en una solución. Cuando el láser golpea un cristal con un pulso de 20 femtosegundos (cuadrillonésimo de segundo), borra el cristal, pero no antes de producir un patrón de difracción en un detector que muestra la estructura atómica de la molécula.
En un artículo de acceso abierto en BMC Biology, los investigadores dirigidos por Marius Schmidt, profesor de la Universidad de Wisconsin-Milwaukee, describió la mezcla del antibiótico ceftriaxona con una enzima resistente utilizada por las bacterias, beta-lactamasa, y alimentándolo al láser pulsante. Debido a que podían ajustar el tiempo entre la mezcla y la llegada al láser, capturaron patrones de difracción de las moléculas cristalizadas no solo en orientaciones aleatorias sino también en varias etapas de interacción.
"Si bien se han realizado estudios elegantes para observar los movimientos de las proteínas con cambios inducidos por la luz, Nuestro trabajo ilustra que una clase más grande de proteínas, a saber, enzimas, se puede investigar de manera resuelta en el tiempo en LCLS (Linac Coherent Light Source) y otros XFEL, "Dijo Olmos.
Phillips dijo que el experimento demostró la utilidad de XFEL para capturar patrones de difracción de cristales de una millonésima de metro de diámetro o menos. mucho más pequeño que las técnicas anteriores. "Esto nos enseñará más sobre cómo la naturaleza ha seleccionado y diseñado estas moléculas para que funcionen, ", dijo." No es diferente a ver una bicicleta pedaleando:obtienes más que una imagen estática y una mejor comprensión de cómo funciona.
"Siempre que desee diseñar una proteína o recrear una máquina molecular, saber más sobre cómo funcionan a un nivel fundamental será útil, ya sea descomponiendo la celulosa para biocombustibles o diseñando un nuevo fármaco o mejorando un fármaco existente ".
En sus presentaciones, Phillips compara la capacidad de tomar instantáneas de proteínas en acción con imágenes del siglo XIX de Eadweard Muybridge que capturaron el movimiento de un caballo al galope a mitad de camino. (Por coincidencia, el caballo era propiedad del fundador de Stanford).
Los investigadores esperan que el XFEL que pronto se actualizará en Stanford, una nueva instalación en Europa y otras en proceso en todo el mundo permitirán a los científicos capturar estructuras en minutos en lugar de días y brindarles datos más detallados sobre los procesos químicos.
Phillips tiene grandes esperanzas de que las herramientas mejoradas también ayuden a capturar las estructuras de las moléculas sin tener que cristalizarlas primero.
"Si podemos conseguir que el haz de rayos X y la dispersión de fondo sean lo suficientemente pequeños y el haz de lectura lo suficientemente limpio, entonces en teoría, en lugar de desfilar cristales, podríamos hacer desfilar moléculas individuales en el láser para construir patrones de difracción, " él dijo.
"El láser de Stanford en este momento dispara a 100 hercios (ciclos por segundo), ", Dijo Phillips." El XFEL europeo va a disparar a las 10, 000 hercios. Eso es una gran mejora porque nos da muchas más oportunidades de golpear las moléculas a medida que ingresan ".
Dijo que, en última instancia, el centro espera capturar datos estructurales para reacciones moleculares sobre la marcha.
"Podrían ser dos proteínas que se unen y aprenden a reconocerse entre sí, podría ser la interacción de un virus con un anticuerpo, podría ser la interacción de algún sustrato con una enzima o cualquier cosa que pueda hacer mezclando o con estimulación externa, ", Dijo Phillips." Una vez que puedas hacer eso, El cielo es el límite."