Los anticuerpos (inmunoglobulinas) son proteínas en forma de Y que reconocen y neutralizan patógenos específicos. Su capacidad para atacar moléculas o células específicas los ha convertido en candidatos prometedores para el desarrollo futuro de fármacos. Sin embargo, sus cadenas ligeras (partes del anticuerpo que contribuyen a reconocer y unirse a antígenos específicos) se pliegan mal y se agregan, lo que lleva a la amiloidosis, una afección que provoca complicaciones y disfunción tisular en el cuerpo.
En el contexto del desarrollo de fármacos, la agregación de anticuerpos puede comprometer su capacidad para unirse a antígenos y disminuir su potencial terapéutico. Sin embargo, la falta de información estructural detallada sobre su agregación es uno de los factores que obstaculiza el progreso en este campo.
Como resultado, los esfuerzos en curso tienen como objetivo proporcionar informes detallados sobre las estructuras agregadas y sus mecanismos de formación para avanzar en el desarrollo de fármacos con anticuerpos.
En un estudio publicado en Nature Communications , un equipo de investigadores de Japón, dirigido por Shun Hirota del Instituto de Ciencia y Tecnología de Nara (NAIST), ha proporcionado recientemente nuevos conocimientos sobre las estructuras formadas durante la agregación de anticuerpos mediante el intercambio de dominios 3D (3D-DS), un proceso en el que un específico región de una proteína se intercambia entre dos o más moléculas de la misma proteína.
El proceso 3D-DS se ha observado en varias proteínas, pero no en las cadenas ligeras de anticuerpos hasta el presente estudio.
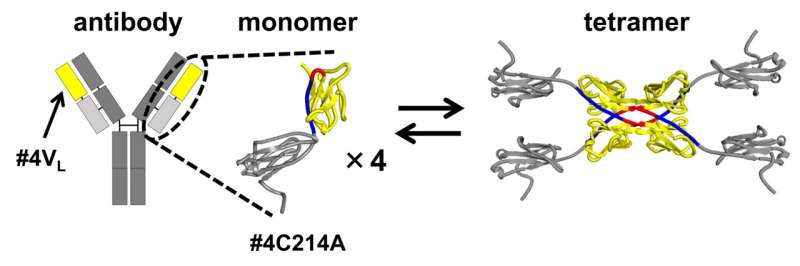
En su investigación, los investigadores utilizaron una versión modificada de la cadena ligera del anticuerpo. En esta forma modificada, un residuo de cisteína (Cys), que normalmente forma un enlace disulfuro con una cisteína de cadena pesada, se reemplazó por alanina (Ala). Esta alteración permitió al equipo aislar y estudiar las estructuras resultantes del 3D-DS en el segmento del anticuerpo que contribuye a la unión al antígeno.
El 3D-DS de la cadena ligera del anticuerpo implica la formación de dímeros (estructuras formadas por dos subunidades idénticas) y tetrámeros (estructuras compuestas por dos dímeros con cuatro subunidades idénticas).
"Nuestro estudio proporciona el primer informe sobre la estructura a nivel atómico del fenómeno 3D-DS en la región variable de la cadena ligera de un anticuerpo", señala Hirota.
La cromatografía de exclusión molecular de la cadena ligera del anticuerpo #4C214A reveló que el anticuerpo existe como monómeros individuales y tetrámeros de cuatro subunidades. Para determinar la región donde se forman los tetrámeros, los investigadores dividieron la cadena ligera del anticuerpo en la región variable (la punta del anticuerpo en forma de Y) y la región constante (la parte media del anticuerpo en forma de Y).
Descubrieron que la región variable #4VL puede cambiar entre estados monoméricos y tetraméricos.
Un análisis más detallado utilizando cristalografía de rayos X y simulaciones termodinámicas reveló que la formación de tetrámeros está impulsada por interacciones hidrofóbicas que ocurren entre dos dímeros 3D-DS.
En comparación con los monómeros, se descubrió que los tetrámeros tenían estructuras de láminas β más rígidas, lo que los hacía menos flexibles. La formación del tetrámero 3D-DS puede ayudar a prevenir la agregación de proteínas al disminuir la flexibilidad, evitando potencialmente la formación de agregados insolubles. Por otro lado, 3D-DS puede promover la agregación de anticuerpos.
Hirota concluye:"Estos hallazgos no sólo aclaran la estructura de dominio intercambiado de la cadena ligera del anticuerpo, sino que también contribuyen a controlar la calidad de los anticuerpos y avanzar en el desarrollo de futuros agentes y fármacos de reconocimiento molecular".
Más información: Conocimientos estructurales y termodinámicos sobre la formación de tetrámeros de cadenas ligeras de anticuerpos mediante el intercambio de dominios 3D, Nature Communications (2023). DOI:10.1038/s41467-023-43443-4
Proporcionado por el Instituto Nara de Ciencia y Tecnología