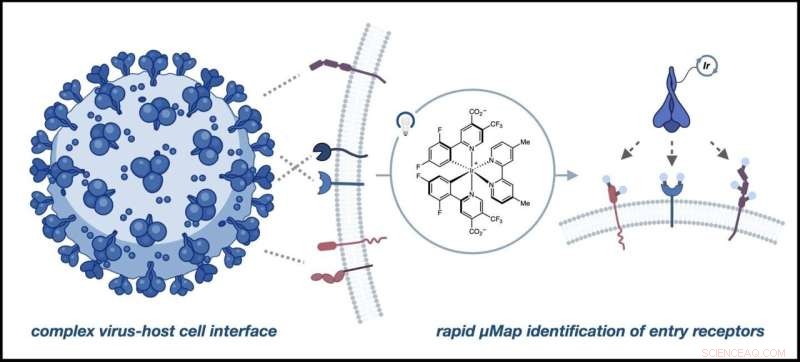
La interfaz entre la célula huésped y el virus puede ser compleja e involucrar muchas proteínas, pero µMap permite la interrogación directa de interacciones funcionalmente importantes. Crédito:Saori Suzuki et al, Journal of the American Chemical Society (2022). DOI:10.1021/jacs.2c06806
Uno de los factores esenciales que el virus COVID-19 necesita para ingresar a un huésped es un receptor en una célula humana, un lugar donde la proteína espiga universalmente reconocida puede adherirse a la superficie celular, perforarla, vomitar su contenido infeccioso y replicarse.
Sin un receptor, no hay replicación. Sin replicación, no hay infección.
Investigadores del Departamento de Química y del Departamento de Biología Molecular de la Universidad de Princeton han utilizado una tecnología de mapeo celular llamada µMap, presentada hace solo dos años por el Laboratorio MacMillan, para descubrir ocho puntos de entrada de interés previamente desconocidos para la proteína espiga.
Los investigadores encontraron que cuatro de ellos son funcionalmente importantes para la entrada viral.
La investigación se publicó a principios de este mes en el Journal of the American Chemical Society (JACS). . Podría expandir el conjunto de herramientas utilizadas para combatir el virus, particularmente a medida que muta y evoluciona para evadir las vacunas.
El proyecto de colaboración se inició en el punto álgido de la incertidumbre pandémica hace dos años bajo la dirección de Alexander Ploss, un destacado virólogo y profesor de biología molecular, y David MacMillan, profesor universitario distinguido James S. McDonnell y premio Nobel de química.
Los científicos saben desde que apareció el virus SARS-CoV-1 en 2003 que su principal receptor de entrada viral era una enzima llamada enzima convertidora de angiotensina 2, o ACE2. Esta enzima se confirmó en 2020 como el mismo receptor del SARS-CoV-2, el virus que causa la COVID-19.
Pero el proyecto de Princeton comenzó con la suposición de que ACE2 no era la única historia.
"Sabíamos que hay ciertas moléculas huésped de las que este virus depende absolutamente para ingresar a las células pulmonares y causar la infección, y una de estas moléculas se llama ACE2", dijo Ploss. "Así que básicamente dijimos, está bien, veamos si hay más por ahí. Buscamos aglutinantes inmediatos.
"Pero como se puede imaginar, el proceso de entrada es complejo. El virus se adhiere a algo y luego aún tiene que atravesar la membrana celular para ingresar a una célula y, de esta manera, puede interactuar con otros factores del huésped. No lo sé. No quiero decir que todo esté dictado por la entrada viral. Obviamente, hay una serie de procesos igualmente esenciales dentro de la célula después de que el virus ha entrado que pueden influir en la gravedad de la enfermedad.
"Pero obviamente es el primer paso clave. Si el virus no puede entrar, se acabó el juego".
Steve Knutson, coautor del artículo e investigador posdoctoral en MacMillan Lab, agregó:"Si bien el descubrimiento de ACE2 como el principal receptor fue un gran hito, ciertamente no cuenta toda la historia de la patología COVID. La biología puede ser intrínsecamente promiscua, y supusimos correctamente que la proteína del pico del SARS-CoV-2 interactúa con múltiples proteínas de la célula huésped para entrar".
Agregó que investigaciones como esta son una investigación "perfecta" para la tecnología µMap.
La espiga como antena µMap
Micromap (µMap) es una tecnología de etiquetado de proximidad que identifica "vecinos" de proteínas y enzimas en la superficie de una célula. Utiliza un fotocatalizador, una molécula que, cuando se activa con la luz, provoca una reacción química, para marcar estas relaciones espaciales mediante la generación de un marcador que etiqueta a los vecinos moleculares.
En este trabajo, los investigadores utilizaron la propia proteína espiga como marcador o "antena" para etiquetar todos los sitios receptores en las inmediaciones de ACE2.
"Alex tenía este gran instinto de que hay otras cosas además de ACE2 que podrían permitirle pensar en la infectividad", dijo MacMillan. "Entonces, lo que hicimos fue colocar este fotocatalizador, y lo llamamos antenas, en la proteína de pico, de modo que cada vez que se une a cosas en la célula cerca de ACE2, esta pequeña antena absorbe la energía fotónica, la luz.
"Pero no puede dar esa energía a largas distancias. Solo puede darla a lo que está cerca. La molécula que flota libremente tiene que encontrarla básicamente dentro de dos nanómetros", agregó MacMillan. "Así que sabemos qué hay al lado. Sabemos qué interactúa con él".
Después de que la tecnología identificara ocho nuevos receptores que interactuaban con la proteína espiga, los científicos los caracterizaron usando una pseudopartícula de virus. (Una pseudopartícula imita la entrada viral pero no lleva el material genético para propagar el virus). Luego aislaron cuatro factores de entrada dignos de una mayor investigación.
"El sistema de pseudopartículas nos permite desacoplar la captación viral y estudiar el proceso de entrada de todo lo que se encuentra aguas abajo del ciclo infeccioso", dijo Ploss. "Si está buscando el impacto de ciertos factores del huésped en la entrada, quiere ver que puede estudiarlo independientemente de la replicación. Así que aquí, básicamente, estamos introduciendo un gen informador en la célula y luego podemos cuantificar la eficiencia de la entrada. ha tenido lugar."
MacMillan dijo que se necesitaba más trabajo para determinar en última instancia la función de los receptores, pero los investigadores se preguntan si podrían dar una pista sobre la gravedad de la enfermedad.
"No podemos decir que los ocho factores estén relacionados con la entrada del SARS-CoV-2", dijo Saori Suzuki, investigadora asociada en Ploss Lab. "Cuatro de los ocho factores sobresalieron en la evaluación virológica. Necesitamos evaluar más y evaluar con mayor precisión.
"En el siguiente paso, debemos evaluar cómo estos factores respaldan a ACE2 para la entrada del virus y si las variantes virales emergentes utilizan el mismo conjunto de factores". Respuesta inmunitaria natural activada por proteína de pico de coronavirus, células musculares cardíacas dañadas