El método de visualización TRAP 'pesca' en busca de proteínas sintéticas de entre una biblioteca de billones de aquellas que pueden apuntar al SARS-CoV-2. El enfoque pudo identificar proteínas que se pueden usar para realizar pruebas del virus y tratar potencialmente a personas infectadas con COVID-19. Crédito:Hiroshi Murakami
Un equipo de investigación dirigido por científicos de la Universidad de Nagoya en Japón ha desarrollado un enfoque que puede encontrar rápidamente proteínas sintéticas que se unen específicamente a objetivos importantes. como componentes del virus SARS-CoV-2. El método fue publicado en la revista Avances de la ciencia y podría usarse para desarrollar kits de prueba o para encontrar tratamientos.
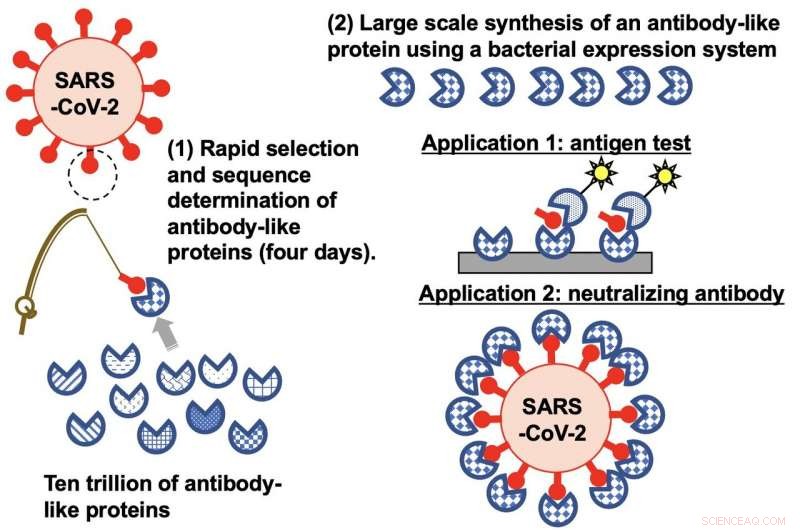
"Desarrollamos una técnica de laboratorio para la selección rápida de proteínas sintéticas que se unen fuertemente al SARS-CoV-2, ", dice el ingeniero biomolecular de la Universidad de Nagoya, Hiroshi Murakami." Las proteínas sintéticas de alta afinidad se pueden utilizar para desarrollar pruebas de antígenos sensibles para el SARS-CoV-2 y para su uso futuro como anticuerpos neutralizantes en pacientes infectados ".
Murakami y sus colegas habían desarrollado previamente una prueba de laboratorio de selección de proteínas llamada pantalla TRAP, que significa "transcripción-traducción acoplada con asociación de enlazador de puromicina". Su enfoque omite dos pasos que requieren mucho tiempo en otra técnica comúnmente utilizada para buscar en bibliotecas de proteínas sintéticas. Pero sus investigaciones indicaron que había un problema con el enlazador de puromicina.
En el estudio actual, el equipo mejoró su técnica modificando el enlazador de puromicina. Por último, pudieron usar su pantalla TRAP para identificar nueve proteínas sintéticas que se unen a la proteína de pico en la membrana externa del SARS-CoV-2. El enfoque tomó solo cuatro días en comparación con las semanas que tomaría usar la tecnología de visualización de ARN mensajero de uso común.
La presentación de TRAP implica el uso de una gran cantidad de plantillas de ADN que codifican y sintetizan billones de proteínas que llevan secuencias de péptidos aleatorias. Las proteínas sintéticas se unen al ADN con la ayuda del enlazador de puromicina modificado y luego se exponen a una proteína diana. Cuando se lava toda la muestra, sólo quedan las proteínas sintéticas que se unen a la diana. Luego, estos se colocan de nuevo en la pantalla de TRAP para rondas adicionales hasta que solo quede una pequeña cantidad de proteínas sintéticas de unión a la diana muy específicas.
Los investigadores investigaron las nueve proteínas sintéticas que se unieron al SARS-CoV-2. Algunos pudieron detectar específicamente el SARS-CoV-2 en hisopos nasales de pacientes con COVID-19, lo que indica que podrían usarse en kits de prueba. Uno también se adhiere al virus para evitar que se una a los receptores que utiliza para acceder a las células humanas. Esto sugiere que esta proteína podría usarse como estrategia de tratamiento.
"Nuestra alta velocidad, La visualización mejorada de TRAP podría ser útil para implementar respuestas rápidas a subespecies de SARS-CoV-2 y a otros posibles virus nuevos que causen futuras pandemias. "dice Murakami.
Este estudio, "Proteínas similares a anticuerpos que capturan y neutralizan el SARS-CoV-2, "se publicó en línea en Avances de la ciencia el 18 de septiembre 2020.