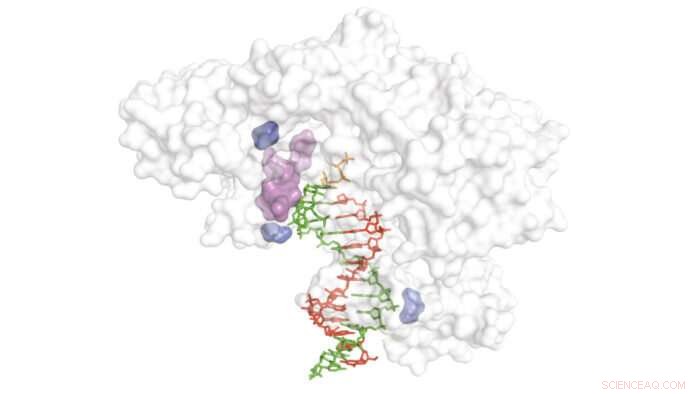
Diagrama que muestra la estructura de la transcriptasa inversa (blanco) con la cadena de ADNc naciente (rojo), Plantilla XNA (verde) y regiones críticas para la transcripción inversa de plantillas XNA (azul y violeta). Crédito:Laboratorio de Biología Molecular del MRC
Además de ser necesario para algunas de las técnicas básicas de la biología molecular, Las enzimas de la transcriptasa inversa (RT) han desempeñado un papel clave en la genética sintética al permitir la síntesis, replicación, y evolución de ácidos xenonucleicos (XNA). Sin embargo, para la mayoría de las químicas de XNA, no hay enzimas RT disponibles o las enzimas existentes tienen baja actividad. El grupo de Philipp Holliger, en la División PNAC de la LMB, han desarrollado un nuevo método de evolución dirigida para mejorar la actividad de RT para cualquier química de ácido nucleico y han descubierto un nuevo grupo de enzimas RT óptimas.
Los XNA son polímeros genéticos como el ADN o el ARN, pero con anillos de azúcar alterados, bases, o columna vertebral. A pesar de estas diferentes químicas, todavía pueden almacenar y transmitir información genética y pueden realizar funciones enzimáticas como las enzimas de ARN, también conocidas como ribozimas. También pueden funcionar como aptámeros y unirse a proteínas con alta especificidad y afinidad. tal como lo hacen los anticuerpos. Estas funciones y las variadas propiedades resultantes de sus diferentes químicas significan que los XNA podrían tener una amplia gama de aplicaciones en biotecnología y medicina. Sin embargo, La generación de aptámeros de XNA y XNAzimas se ha visto limitada por la falta de enzimas RT de alta fidelidad.
¿Qué es la transcripción inversa?
El primer paso del llamado dogma central de la biología molecular es la transcripción del ADN para producir ARN. El ARN también se puede transcribir de forma inversa para producir ADN y las enzimas RT sintéticas permiten el acceso a los XNA.
Al permitir que los científicos conviertan ARN en ADN, Las enzimas RT permiten a los científicos estudiar más fácilmente qué genes se están transcribiendo dentro de las células, y, por tanto, qué genes están "activados", "a través de técnicas básicas como RT-PCR y RNAseq. Junto con las aplicaciones en investigación, esta capacidad también se utiliza para pruebas médicas, como para probar la presencia de ARN viral, incluso en las pruebas COVID-19.
Para abordar la falta de enzimas RT de alta fidelidad, Gillian Houlihan y otros del grupo de Philipp desarrollaron una nueva técnica de evolución dirigida que condujo al descubrimiento de un nuevo grupo de enzimas RT óptimas que pueden decodificar información genética de manera más precisa y eficiente. En tono rimbombante, este nuevo método es compatible con cualquier química de ácidos nucleicos y su descubrimiento incluye nuevas enzimas RT para químicas XNA para las que no existía previamente ninguna enzima RT. Entre las nuevas enzimas RT se encuentran las primeras enzimas capaces de realizar una revisión activa durante la transcripción inversa XNA, mejorando la precisión.
Las enzimas RT de ARN de alta fidelidad tendrán aplicaciones inmediatas en investigación y biotecnología, ya que proporcionarán una precisión de secuenciación mejorada al analizar ARN celulares o virales. La actividad mejorada de XNA RT probablemente ayudará al desarrollo de nuevos aptámeros de XNA que podrían ser útiles en el diagnóstico y la terapéutica de una amplia gama de enfermedades. Como ejemplo específico, este trabajo incluye la primera enzima RT para la química XNA utilizada en el oligo fármaco antisentido nusinersen que cuenta con la aprobación de la FDA y la EMA para el tratamiento de la atrofia muscular espinal. El descubrimiento de esta enzima RT abre la posibilidad de cuantificar el nivel y la vida media de este fármaco en los pacientes. lo que podría ayudar al tratamiento.