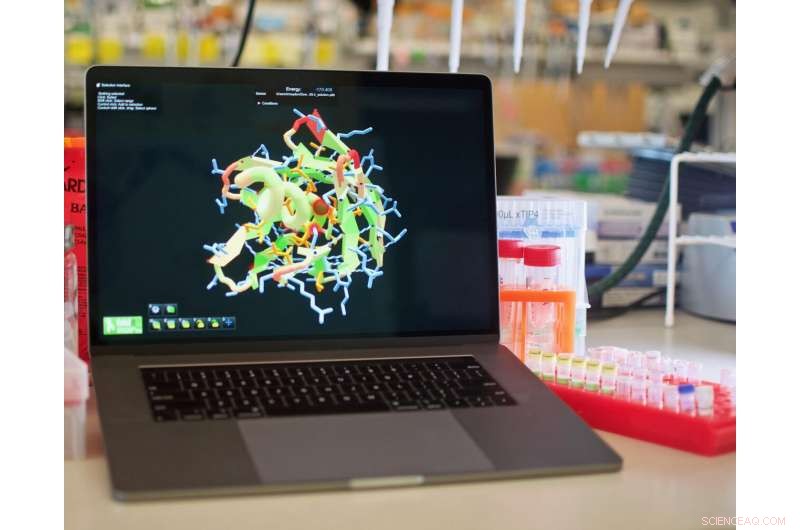
Al jugar al videojuego Foldit, Los entusiastas de los rompecabezas ahora pueden diseñar proteínas que nunca antes existieron, y ayuda en la creación de nuevas vacunas y tratamientos para el cáncer y otras enfermedades graves. Crédito:Instituto de Diseño de Proteínas / UW Medicine
Un equipo de investigadores codificó su conocimiento especializado en el juego de computadora Foldit para permitir a los científicos ciudadanos diseñar con éxito proteínas sintéticas por primera vez.
Los resultados iniciales de esta colaboración aparecen en la edición del 5 de junio de Naturaleza . El Instituto de Diseño de Proteínas de la Facultad de Medicina de la Universidad de Washington lideró el esfuerzo multiinstitucional.
"Hay más proteínas posibles que átomos en el universo. Es emocionante pensar que ahora cualquiera puede ayudar a explorar este vasto espacio de posibilidades, "dijo el coautor principal David Baker, profesor de bioquímica en la Facultad de Medicina de la Universidad de Washington y director del Instituto de Diseño de Proteínas.
"La diversidad de moléculas que se les ocurrió a estos jugadores es asombrosa, "dijo el autor principal Brian Koepnick, investigador postdoctoral en el Instituto de Diseño de Proteínas. "Estas nuevas proteínas no son de ninguna manera inferiores a las cosas que podría hacer un científico con nivel de doctorado".
Foldit se creó en 2008 como una forma de "gamificar" la investigación de proteínas. Las proteínas son biomoléculas esenciales que se encuentran dentro de cada célula de cada organismo. Sus intrincadas estructuras tridimensionales dan lugar a sus diversas funciones, que incluyen la digestión, cicatrización de la herida, autoinmunidad y mucho más.
A través del juego, Los jugadores de Foldit han ayudado a determinar la estructura de una proteína relacionada con el VIH y han mejorado la actividad de enzimas útiles. Hasta ahora, sin embargo, Los jugadores de Foldit solo podían interactuar con proteínas que ya existían. No había forma de diseñar nuevos.
"Diseñar proteínas completamente nuevas que no existían en la naturaleza ha sido nuestro objetivo con Foldit durante mucho tiempo, "dijo el coautor principal Seth Cooper, profesor asistente en el Khoury College of Computer Sciences en Northeastern University. "Este nuevo conjunto de resultados muestra que es posible".
Para convertir Foldit en una plataforma para el diseño de proteínas, los investigadores codificaron el conocimiento bioquímico en el juego. De esa manera, Las moléculas de diseño que obtuvieron buenos resultados en Foldit tendrían más probabilidades de plegarse como se esperaba en el mundo real.
"No les dimos [a los jugadores de Foldit] conferencias ni les dijimos que leyeran nada. En cambio, ajustamos el código que ha ejecutado el juego durante muchos años, "dijo el coautor principal Firas Khatib, profesor asistente de informática en la Universidad de Massachusetts Dartmouth.
Los científicos probaron 146 proteínas diseñadas por jugadores de Foldit en el laboratorio. Se encontró que 56 eran estables. Este hallazgo sugirió que los jugadores habían producido algunas proteínas realistas. Los investigadores recopilaron suficientes datos sobre cuatro de estas nuevas moléculas para demostrar que los diseños adoptaron las estructuras previstas.
"Nunca hubiera creído que llegarían a ser tan buenos, pero los jugadores de Foldit nunca dejan de sorprendernos ", dijo Khatib.
Elaborar nuevas proteínas es un poco como intentar atar nudos nunca antes vistos con una cuerda que es un millón de veces más delgada que un cabello humano. Hasta la fecha, solo un pequeño grupo de expertos con un conocimiento íntimo de la forma en que las biomoléculas se retuercen y giran se molestó con esta tarea extremadamente compleja. La mayoría usa algoritmos de diseño molecular automatizados, y la mayoría de los algoritmos de diseño fallan con mucha más frecuencia de lo que tienen éxito.
"Siempre intentamos mejorar los algoritmos, pero el elemento humano es clave, "dijo Khatib." De hecho, a través del diseño Foldit, los jugadores incluso han descubierto fallas en la función energética de Rosetta, nuestro método de vanguardia para el diseño de proteínas ".
El diseño de proteínas es una disciplina científica emergente. En los últimos cinco años, los expertos del Institute for Protein Design y sus colegas han creado proteínas que estimulan el sistema inmunológico para combatir el cáncer y otras que actúan como potentes candidatas a vacunas. En abril, el Institute for Protein Design recibió un compromiso de $ 45 millones en financiamiento a través de The Audacious Project, una colaboración filantrópica organizada por TED, diseñar vacunas a base de proteínas, medicamentos y materiales.
¿Podrían los jugadores crear la próxima droga de gran éxito?
"Los jugadores de Foldit son una nueva incorporación al arsenal de investigación, "dijo Khatib." No son una bala de plata, pero son un recurso asombroso ".
Cualquiera que esté interesado en ayudar a resolver acertijos científicos puede obtener más información en fold.it/portal/