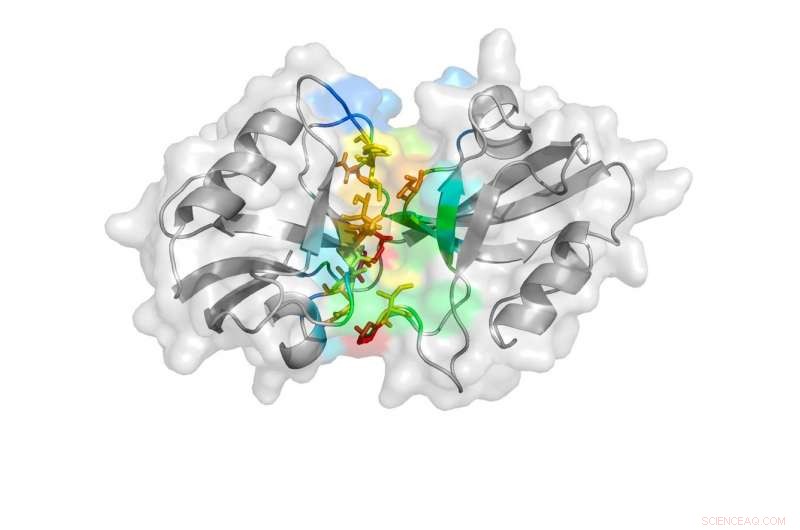
Una representación de dibujos animados revelada por el estudio muestra el estado cerrado de los dominios PDZ. Crédito: Comunicaciones de la naturaleza , doi:10.1038 / s41467-018-06133-0
En una extensión de la investigación publicada hace un mes en Métodos de la naturaleza , un nuevo enfoque híbrido realizado por investigadores del departamento de física y astronomía de la Universidad de Clemson y de la Universidad de Stony Brook ha revelado una estructura tridimensional de un fragmento de proteína que podría servir como objetivo farmacológico en el tratamiento de pacientes con accidente cerebrovascular.
La proteína llamada "proteína de densidad postsináptica de 95 kDa (PSD-95)" se coloca en las neuronas del cerebro que reciben mensajes químicos (neurotransmisores) de las neuronas adyacentes. Al reclutar receptores y otras proteínas auxiliares, PSD-95 trabaja para mantener la integridad de las conexiones neuronales a lo largo del tiempo, facilitando así la comunicación neuronal, Aprendizaje y Memoria.
PSD-95 consta de cinco partes, o dominios, que cada uno juega un papel diferente en la función general de la proteína. Dos de estos dominios, llamados PDZ-1 y PDZ-2, se ha demostrado que influyen en los síntomas asociados con el accidente cerebrovascular isquémico, como parálisis o problemas del habla.
"Una de las ideas que se ha postulado en la literatura es crear un fármaco multivalente que se dirija a ambos dominios PDZ porque son de naturaleza muy similar. Si puede bloquear los dominios PDZ para que no se unan a proteínas o enzimas particulares, puede reducir los efectos debilitantes de un accidente cerebrovascular, "dijo Hugo Sanabria, autor principal del estudio.
El reto, sin embargo, es que es casi imposible diseñar un inhibidor de fármacos sin conocer primero la estructura exacta de los dominios PDZ de PSD-95. Sería como conducir por todo el país sin tener un mapa de los Estados Unidos.
"Las funciones biológicas de las biomoléculas están determinadas por sus estructuras, por lo que necesitamos información detallada estructural y dinámica de PDZ-1 y -2 para ayudar a comprender mejor sus roles funcionales y ayudar en el diseño de nuevos inhibidores, "dijo Feng Ding, Colega de Sanabria aquí en Clemson.
Existe un puñado de enfoques para representar la estructura de biomoléculas. Pero en el caso de PSD-95, cada enfoque:espectroscopia de RMN, La cristalografía de rayos X y la transferencia de energía por resonancia de Förster (FRET) proporcionaron un modelo estructural diferente. Colaborador de los investigadores en la Universidad de Stony Brook, el profesor asociado Mark Bowen en el departamento de fisiología y biofísica, estableció una asociación con Sanabria en este proyecto después de que descubrió uno de los modelos estructurales inconsistentes del fragmento PSD-95.
El laboratorio de Sanabria abordó esta discrepancia modelando primero el fragmento PSD-95 usando FRET, un enfoque que identifica posibles configuraciones de biomoléculas. Bajo este método, Sanabria adjuntó dos moléculas sensibles a la luz, llamados cromóforos, en dos posiciones diferentes en el fragmento PSD-95. Luego descubrió la distancia entre los cromóforos visualizando el fragmento bajo un microscopio. Esto se repitió varias veces desde diferentes puntos de unión.
"Para el aspecto de modelado, FRET te da distancias entre cromóforos, pero eso no es suficiente para llenar todas las restricciones geométricas de la molécula, así que tenemos que confiar en otra cosa, alguna otra metodología. Ahí es donde entra en juego el profesor Ding, "Dijo Sanabria.
Ding dirige un laboratorio de biofísica computacional en la Universidad de Clemson, donde usa software de computadora para medir cómo se ven las biomoléculas. moverse y funcionar. Su enfoque del modelado utiliza una simulación por computadora conocida como dinámica molecular discreta (DMD) que mapea el paisaje de una biomolécula, predecir las trayectorias de las proteínas a medida que se pliegan e interactúan con otras moléculas. La siguiente simulación se puede reproducir como una película, ayudando a los investigadores a visualizar los comportamientos de las proteínas a lo largo del tiempo.
"Si haces simulaciones moleculares tradicionales, por lo general, tomará muestras de una región muy pequeña del espacio, particularmente para moléculas más grandes, por lo que no tendrá una buena descripción general de cómo se verá toda la molécula, incluso en condiciones fisiológicas, "Dijo Sanabria." La dinámica molecular discreta es una forma mucho más rápida y menos costosa desde el punto de vista computacional de muestrear con precisión y rapidez el espacio conformacional de las proteínas ".
Para hacerlo, Sanabria obtuvo por primera vez un conjunto de distancias midiendo PSD-95 con FRET. En ese experimento, Sanabria tenía 10 muestras del fragmento PSD-95, cada una de las cuales presentaba distancias diferentes y se observaron tres formas o conformaciones comunes de PSD-95. Todavía, sin una simulación DMD, los investigadores no tenían forma de saber qué distancia correspondía a qué conformación del fragmento. Entonces ingresan cada distancia posible contra cada forma posible y dejan que la simulación haga el resto.
"Una vez que hicimos la primera simulación, vimos que había tres estados principales que estaban tomando PDZ-1 y -2. Uno mostró un contacto muy estrecho entre los dos, uno mostró un conjunto de contacto intermedio y el otro no tuvo contacto alguno, "Dijo Ding.
Luego, los investigadores volvieron a ejecutar una simulación DMD sin considerar las distancias FRET para confirmar que los tres estados observados existen en la naturaleza y no son simplemente una casualidad impuesta por las distancias FRET. Investigaron aún más las estructuras al observar la forma en que los aminoácidos individuales, que constituyen los dominios PDZ, vínculo entre sí. De estos análisis, Timbre, Bowen y Sanabria pudieron confirmar que los dominios PDZ adoptan dos de los tres estados observados en la simulación DMD:el que tiene algún contacto y el que no tiene ningún contacto.
"Ahora, Tenemos dos objetivos potenciales para diseñar nuevos medicamentos que serán más eficientes que los que están disponibles actualmente. ", Dijo Sanabria." Las perspectivas para los pacientes con accidente cerebrovascular son prometedoras ".
Sin dinámica molecular discreta, que puede capturar cambios conformacionales que ocurren en la escala de tiempo de microsegundos, estos dos estados se habrían perdido como en estudios anteriores.
"La mayoría de las personas que realizan modelos estructurales guiados por FRET están trabajando con una molécula rígida, como el ADN. Si tienes una molécula rígida, es fácil de modelar:solo tiene que capturar un estado. Puedes asignar las distancias FRET y realmente no hay problema, "Dijo Sanabria." En este caso, superamos este enfoque de muchas maneras ".
En estudios futuros, el equipo está buscando analizar el potencial del fragmento PSD-95 para autoinhibirse basándose en la propia estructura del fragmento.