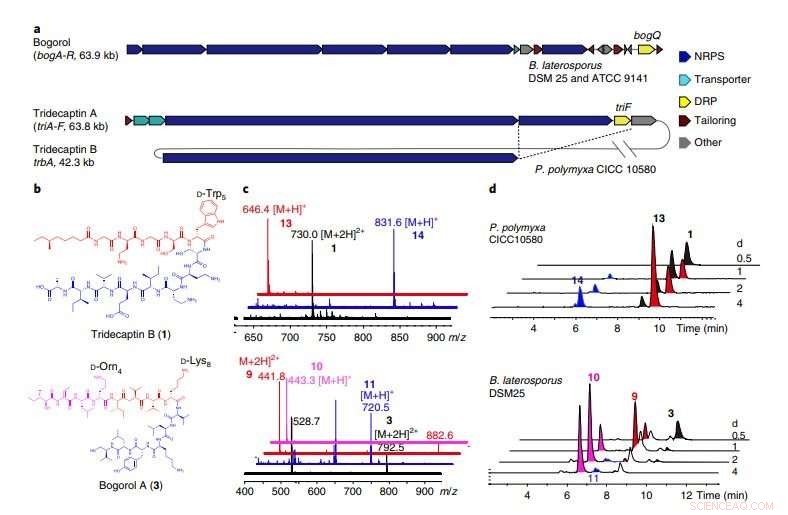
a, Las BGC de bogorol de B. laterosporus DSM 25 y ATCC 9141, así como tridecaptina A y tridecaptina B de P. polymyxa CICC 10580. BogQ de la cepa DSM 25 y la de la cepa ATCC 9141 comparten una identidad de secuencia de aminoácidos del 85 por ciento. Las líneas de puntos que se cruzan indican genes compartidos por dos grupos de genes dentro del mismo hospedador (Fig. 10 complementaria). B, Estructuras de DNRPs tridecaptina B y bogorol A, con motivos de reconocimiento DRP resaltados. C, Superposición apilada de los espectros de masas (ionización por electropulverización) de los compuestos originales (negro), Fragmentos C-terminales (azul), y fragmentos N-terminales (rojo). Los datos son representativos de dos experimentos independientes. Cima, tridecaptina B; fondo, bogorol A. d, Análisis del curso del tiempo de los compuestos correspondientes producidos por P. polymyxa CICC 10580 (arriba) y B. laterosporus DSM 25 (abajo) en diferentes tiempos de fermentación (representativos de tres experimentos independientes). Crédito:División de Ciencias de la Vida, HKUST
Antibióticos peptídicos no ribosomales, incluyendo polimixina, vancomicina, y teixobactina, la mayoría de los cuales contienen D-aminoácidos, son muy eficaces contra bacterias multirresistentes. Sin embargo, El uso excesivo de antibióticos sin tener en cuenta el riesgo de aparición de resistencias ha llevado inexorablemente a la aparición generalizada de bacterias resistentes. Elucidar los mecanismos poco conocidos de resistencia a los antibióticos peptídicos es fundamental cuando se implementan antibióticos peptídicos y mejoraría de manera efectiva la eficiencia.
En un estudio reciente, un grupo de científicos de la Universidad de Ciencia y Tecnología de Hong Kong revela tanto la distribución generalizada como el potencial de resistencia de amplio espectro de las peptidasas D-estereoespecíficas, proporcionando un posible indicador temprano de la resistencia a los antibióticos a los antibióticos peptídicos no ribosomales.
Sus hallazgos fueron publicados en la revista Biología química de la naturaleza el 26 de febrero, 2018.
"Aplicamos un enfoque a 5, 585 genomas bacterianos completos que abarcan todo el dominio de las bacterias, "dijo Pei-Yuan Qian, catedrático de la División de Ciencias de la Vida, HKUST, y autor principal del artículo. "Con posteriores análisis químicos y enzimáticos, demostramos un mecanismo de resistencia hacia antibióticos peptídicos no ribosomales que se basa en la escisión hidrolítica por peptidasas D-estereoespecíficas ".
El equipo identificó una familia de peptidasas de resistencia D-estereoespecíficas (DRP) que están ampliamente distribuidas filogenéticamente en la naturaleza. Se descubrió que el hallazgo de PRM está involucrado en la lucha contra los antibióticos ampliamente distribuidos que contienen D-aa para la supervivencia de su huésped. que fue validado experimentalmente mediante una combinación de edición de genes basada en CRISPR / Cas9, análisis químicos y enzimáticos.
"Dado el potencial de los PRM para la resistencia de amplio espectro y su potencial para atacar antibióticos clínicamente importantes que contienen D-aa, Es probable que estos genes de resistencia ampliamente distribuidos sean particularmente peligrosos si se transfieren a patógenos oportunistas. ", dijo el profesor Qian." El hallazgo de PRD en la naturaleza constituye solo la punta del iceberg, lo que conducirá a una intensa investigación sobre el uso y desarrollo de antibióticos peptídicos para combatir la resistencia a los antibióticos ".
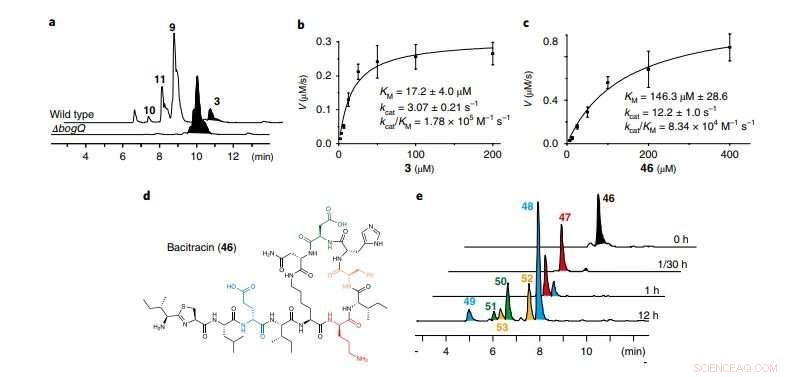
a, Rastros de LC-MS que comparan B. laterosporus ATCC 9141 de tipo salvaje y el mutante ΔbogQ (representativo de tres experimentos independientes). B, C, Análisis cinéticos de la hidrólisis catalizada por BogQ de bogorol A (b; 3) y bacitracina (c; 46) v, velocidad de reacción. Los datos son la media ± ± s.d .; n =? 3 experimentos independientes. D, Estructura del antibiótico bacitracina DNRP; los colores resaltan los sitios de escisión de BogQ. mi, Rastros de LC-MS de ensayos in vitro de BogQ (2,0 µM) contra 46 (200 µM; representativo de dos experimentos independientes). Los productos de escisión en el transcurso del tiempo (47-53) de 46 se marcan usando el mismo código de color que sus sitios de escisión d-aa en d. Para patrones de escisión enzimática, ver la Fig. 22 complementaria. Crédito:División de Ciencias de la Vida, HKUST