Gráficamente abstracto. Crédito:Celular (2022). DOI:10.1016/j.cell.2022.08.003
Estudios clave en la última década han demostrado que el microbioma intestinal, la colección de cientos de especies bacterianas que viven en el sistema digestivo humano, influye en el desarrollo neuronal, la respuesta a las inmunoterapias contra el cáncer y otros aspectos de la salud. Pero estas comunidades son complejas y sin formas sistemáticas de estudiar los constituyentes, las células y moléculas exactas vinculadas con ciertas enfermedades siguen siendo un misterio.
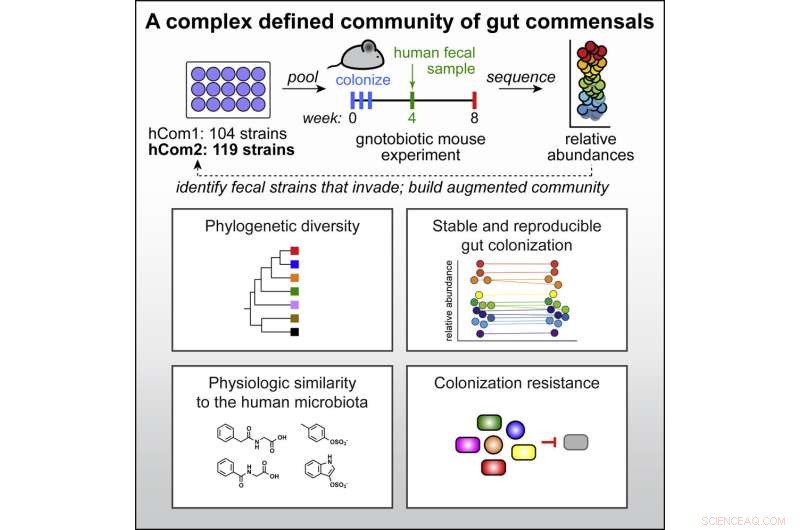
Investigadores de la Universidad de Stanford han construido el microbioma sintético más complejo y mejor definido, creando una comunidad de más de 100 especies bacterianas que se trasplantaron con éxito a ratones. La capacidad de agregar, eliminar y editar especies individuales permitirá a los científicos comprender mejor los vínculos entre el microbioma y la salud y, finalmente, desarrollar terapias de microbioma de primera clase.
Se han realizado muchos estudios clave de microbioma utilizando trasplantes fecales, que introducen el microbioma natural completo de un organismo a otro. Si bien los científicos rutinariamente silencian un gen o eliminan una proteína de una célula específica o incluso de un ratón completo, no existe tal conjunto de herramientas para eliminar o modificar una especie entre los cientos en una muestra fecal dada.
"Mucho de lo que sabemos sobre biología, no lo sabríamos si no fuera por la capacidad de manipular sistemas biológicos complejos por partes", dijo Michael Fischbach, becario del Instituto en Sarafan ChEM-H y autor correspondiente del estudio, publicado en Celda el 6 de septiembre.
Fischbach, que es profesor asociado de bioingeniería y de microbiología e inmunología, y otros vieron una solución:construir un microbioma desde cero cultivándolo individualmente y luego mezclando sus bacterias constituyentes.
Construyendo el arca
Cada célula del microbioma ocupa un nicho funcional específico, realizando reacciones que descomponen y acumulan moléculas. Para construir un microbioma, el equipo tuvo que asegurarse de que la mezcla final no solo fuera estable, manteniendo un equilibrio sin que ninguna especie dominara al resto, sino también funcional, realizando todas las acciones de un microbioma natural completo. Seleccionar especies para incluir en su comunidad sintética también fue difícil dada la variación natural entre individuos; dos personas seleccionadas al azar comparten menos de la mitad de sus genes microbianos.
Los investigadores decidieron construir su colonia a partir de las bacterias más prevalentes y recurrieron al Proyecto Microbioma Humano (HMP), una iniciativa de los Institutos Nacionales de Salud para secuenciar los genomas microbianos completos de más de 300 adultos.
"Estábamos buscando el Arca de Noé de especies bacterianas en el intestino humano, tratando de encontrar las que casi siempre estaban allí en cualquier individuo", dijo Fischbach.
Seleccionaron más de 100 cepas bacterianas que estaban presentes en al menos el 20% de los individuos HMP. Agregar algunas especies necesarias para algunos estudios posteriores los llevó a 104 especies, que cultivaron en poblaciones individuales y luego mezclaron en un cultivo combinado para hacer lo que ellos llaman comunidad humana, o hCom1.
Aunque estaba satisfecho de que las cepas pudieran coexistir en el laboratorio, la verdadera prueba era si su nueva colonia echaría raíces en el intestino. Introdujeron hCom1 en ratones que están cuidadosamente diseñados para no tener bacterias presentes. hCom1 fue notablemente estable, con el 98 % de las especies constituyentes colonizando el intestino de estos ratones libres de gérmenes, y los niveles de abundancia relativa de cada especie permanecieron constantes durante dos meses.
Invasión extranjera
Para hacer que su colonia fuera más completa, los investigadores querían asegurarse de que todas las funciones vitales del microbioma fueran realizadas por una o más especies. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge
To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases. Ecological principles at play in gut microbiome