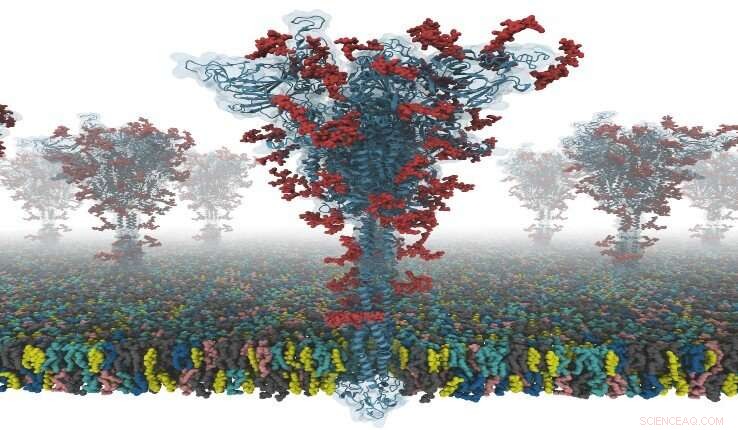
Un modelo de una proteína S. Crédito:Dr. Yeolkyo Choi / Lehigh
El virus SARS coronavirus 2 (SARS-CoV-2) es la causa conocida de la enfermedad por coronavirus 2019 (COVID-19). La proteína "pico" o S facilita la entrada del virus en las células huésped.
Ahora, un grupo de investigadores de la Universidad Nacional de Seúl en Corea del Sur, Universidad de Cambridge en Reino Unido, y la Universidad de Lehigh en EE. UU., han trabajado juntos para producir los primeros modelos de átomos de código abierto de una proteína S de longitud completa. Los investigadores dicen que esto es de particular importancia porque la proteína S juega un papel central en la entrada del virus en las células. convirtiéndolo en un objetivo principal para el desarrollo de vacunas y fármacos antivirales.
Los detalles se pueden encontrar en un documento, "Desarrollo de un modelo de proteína espiga de SARS-CoV-2 de longitud completa completamente glicosilada en una membrana viral", publicado recientemente en línea en La Revista de Química Física B .
Este video de demostración ilustra cómo construir este sistema de membrana a partir de sus modelos de proteína SARS-CoV-2 S. El programa de construcción de modelos es de acceso abierto y se puede encontrar desde la página de inicio de CHARMM-GUI haciendo clic en el enlace Archivo COVID-19, o haciendo clic en el enlace de archivo en el encabezado, luego, el enlace de proteínas COVID-19 en la barra lateral izquierda.
Desarrollado por Wonpil Im, profesor del Departamento de Ciencias Biológicas y Bioingeniería de la Universidad de Lehigh, CHARMM-GUI (GUI =interfaz gráfica de usuario) es un programa que simula sistemas biomoleculares complejos de forma sencilla, con precisión y rapidez. Im lo describe como un "microscopio computacional" que permite a los científicos comprender las interacciones a nivel molecular que no se pueden observar de otra manera. Puede encontrar más información sobre CHARMM-GUI en este video.
"Nuestros modelos son los primeros modelos de proteína de pico (S) de SARS-CoV-2 de longitud completa y completamente glicosilada que están disponibles para otros científicos, ", dice Im." Tuve la suerte de colaborar con el Dr. Chaok Seok de la Universidad Nacional de Seúl en Corea y el Dr. Tristan Croll de la Universidad de Cambridge en el Reino Unido. Nuestro equipo pasó días y noches para construir estos modelos con mucho cuidado a partir del conocido crio- Porciones de estructura EM. El modelado fue muy desafiante porque había muchas regiones en las que el modelado simple no proporcionaba modelos de alta calidad ".
Los científicos pueden utilizar los modelos para realizar investigaciones de simulación innovadoras y novedosas para la prevención y el tratamiento de COVID-19, según Im.
La estructura de la proteína S se determinó con crio-EM con el RBD arriba (ID de PDB:6VSB), y con el RBD abajo (ID PDB:6VXX). Pero, este modelo tiene muchos residuos faltantes. Entonces, primero modelaron los residuos de aminoácidos faltantes, y luego otros dominios faltantes. Además, modelaron todos los glicanos (o carbohidratos) potenciales unidos a la proteína S. Estos glicanos previenen el reconocimiento de anticuerpos, lo que dificulta el desarrollo de una vacuna. También construyeron un sistema de membrana viral de una proteína S para la simulación de dinámica molecular.