Crédito:Universidad de Pensilvania
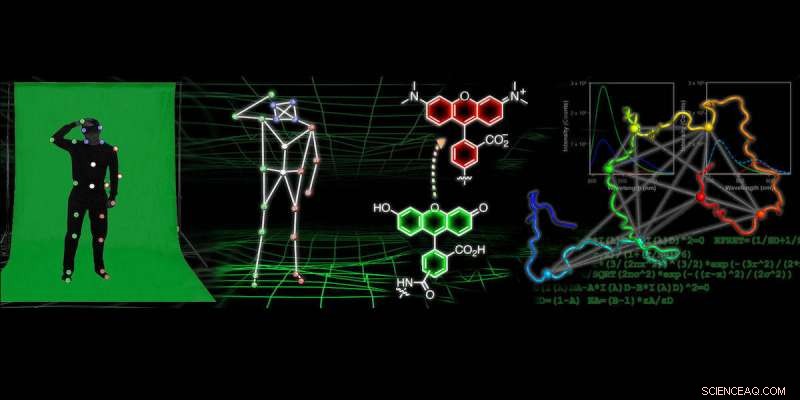
En muchas películas animadas modernas, el truco para lograr movimientos realistas para personajes y objetos individuales radica en la tecnología de captura de movimiento. Este proceso a menudo involucra a alguien que usa un traje de seguimiento cubierto con bolas de colores mientras una cámara captura la posición de esas bolas de colores, que luego se usa para representar cómo se mueve la persona.
Investigadores de la Universidad de Pennsylvania están desarrollando una tecnología similar para obtener "películas" de resolución atómica que rastrean cómo las proteínas se pliegan y cambian de forma. Para generar estas películas, los científicos etiquetan la proteína con sondas en muchas posiciones y observan el movimiento de esas etiquetas. Los datos de fluorescencia sobre las posiciones relativas de las sondas se pueden utilizar para construir modelos computacionales de la estructura de la proteína en detalle atómico. Esta investigación podría conducir a mejoras en los medicamentos utilizados para tratar enfermedades neurodegenerativas, así como nuevos métodos de obtención de imágenes que podrían conducir a su detección más temprana.
La investigación fue un esfuerzo multidisciplinario dirigido por E. James Petersson. El artículo fue publicado en Revista biofísica .
"Una de las grandes preguntas fundamentales en bioquímica es cómo las proteínas se pliegan en una forma determinada, "dijo Petersson, "y esto está dictado por la secuencia de aminoácidos en la proteína. La información en todas las interacciones de las cadenas laterales de aminoácidos de alguna manera conduce a que se pliegue en una forma adecuada".
En escenarios saludables, Petersson dijo:que la forma adecuada permite que la proteína tenga diferentes funciones, como transportar oxígeno en la sangre o convertirse en "máquinas moleculares" que finalmente conducen a movimientos musculares, como los necesarios para caminar y correr.
Pero en ciertos estados de enfermedad, particularmente en enfermedades neurodegenerativas como el Alzheimer y el Parkinson, las proteínas se pliegan mal en una forma poco saludable, lo que puede hacer que múltiples copias de las proteínas se agreguen en "marañas parecidas a espaguetis o fibrillas largas". Estas fibrillas, Petersson dijo:son tóxicos para las neuronas, que subyace a la enfermedad de Alzheimer y Parkinson.
"Dado que todos estos implican el plegamiento de proteínas en una forma determinada, entonces lo que nos gustaría hacer es rastrear los cambios en la forma de las proteínas, ", dijo." Hay varias técnicas diferentes que se pueden utilizar para hacer esto, pero nos gusta la fluorescencia porque puede adquirir datos de fluorescencia lo suficientemente rápido como para ver cómo se pliegan las proteínas en tiempo real. En última instancia, nos gustaría intentar observar el plegamiento de las proteínas en las células ".
Para obtener información sobre la forma de las proteínas mediante sondas fluorescentes, Los investigadores utilizan una técnica llamada transferencia de energía por resonancia de fluorescencia, lo que les obliga a medir muchas distancias entre diferentes puntos de la proteína y luego usar esa información para comprender su forma, similar a la tecnología de captura de movimiento.
En este papel, los investigadores realizaron alrededor de 30 mediciones de diferentes distancias dentro de la proteína alfa-sinucleína en diferentes estados en los que está cambiando de forma. Luego utilizaron esa colección de mediciones de distancia en combinación con modelos computacionales complejos para obtener estructuras de resolución atómica de la forma de la proteína.
Ferrie recibió una beca de la Fundación de la Enfermedad de Parkinson para pasar el verano en el laboratorio de David Baker en la Universidad de Washington, donde Rosetta, uno de los programas más utilizados para modelar proteínas, fue desarrollado.
"Rosetta está diseñada para modelar proteínas estables bien plegadas, Petersson dijo:"proteínas no desordenadas que pueden cambiar de forma, así que Jack tuvo que reescribir mucho el código él mismo para poder modelar estas proteínas rebeldes ".
Ferrie y Yoon hicieron una serie de mediciones experimentales que podrían usarse para dirigir el plegamiento de proteínas para que sea consistente con las mediciones experimentales, lo que le permitió modelar la forma de las proteínas.
Dado que se trataba de un nuevo enfoque, los investigadores querían demostrar que las estructuras que surgían de los modelos computacionales eran consistentes con la realidad. Para hacer esto, llevaron a cabo tres tipos de experimentos para hacer coincidir los datos reales con los modelos que surgen de estos experimentos de fluorescencia.
Colaboraron con el grupo de Rhoades para validar las estructuras modeladas utilizando mediciones de fluorescencia de una sola molécula realizadas por Pan. Trabajando en el grupo de Fakhraii, Lin, utilizó un tipo diferente de técnica llamada microscopía de fuerza atómica para obtener imágenes de la proteína y validar los modelos. Chris Dobson, profesor de química en la Universidad de Cambridge, compartió datos de resonancia magnética nuclear sobre la alfa-sinucleína que proporcionaron una confirmación adicional de la precisión de los modelos informáticos.
El artículo representa una de las bibliotecas más grandes de proteínas marcadas con fluoróforos sintéticos hasta la fecha. Según Petersson, los investigadores necesitaban realizar muchas mediciones de distancia en diferentes regiones de la proteína para tener suficientes datos para generar modelos computacionales. Ferrie y Haney tuvieron que idear un enfoque simplificado para conectar diferentes conjuntos de sondas que funcionarían en diferentes rangos de distancia.
Los investigadores ahora están trabajando para aplicar esta técnica para modelar la estructura de la proteína en las formas agregadas que son tóxicas para las neuronas y modelar su respuesta a los medicamentos que harían que cambie de forma. previniendo esta agregación.
"La capacidad de observar una proteína a medida que cambia de forma, "dijo Petersson, "y sacar estructuras de eso es un objetivo científico básico realmente importante en el que hemos estado trabajando durante 10 años. Ha habido algunos avances muy impresionantes en la obtención de estructuras de proteínas en enfermedades neurodegenerativas, pero la técnica de fluorescencia tiene el potencial de hacer eso en células vivas, que ninguna otra técnica tiene la capacidad de hacer ".
Según Petersson, proteínas que adoptan múltiples formas, apilando diferentes copias entre sí y agregando, como lo hacen en las enfermedades neurodegenerativas, son un importante problema de bioquímica básica que otras técnicas de biología estructural realmente no pueden abordar. A better understanding of how that works and what those shapes are has the potential to make an impact on diseases such as Parkinson's and to give researchers an opportunity to figure out how drug or diagnostic models interact with the protein.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."