Un equipo de científicos dirigido por la Universidad de Oxford ha logrado un avance significativo en la detección de modificaciones en las estructuras de las proteínas. El método, publicado en Nature Nanotechnology , emplea una innovadora tecnología de nanoporos para identificar variaciones estructurales a nivel de una sola molécula, incluso en lo profundo de largas cadenas de proteínas.
Las células humanas contienen aproximadamente 20.000 genes que codifican proteínas. Sin embargo, el número real de proteínas observadas en las células es mucho mayor:se conocen más de 1.000.000 de estructuras diferentes. Estas variantes se generan mediante un proceso conocido como modificación postraduccional (PTM), que ocurre después de que una proteína se ha transcrito del ADN.
PTM introduce cambios estructurales como la adición de grupos químicos o cadenas de carbohidratos a los aminoácidos individuales que forman las proteínas. Esto da como resultado cientos de posibles variaciones para la misma cadena proteica.
Estas variantes desempeñan funciones fundamentales en la biología, al permitir una regulación precisa de procesos biológicos complejos dentro de las células individuales. Mapear esta variación descubriría una gran cantidad de información valiosa que podría revolucionar nuestra comprensión de las funciones celulares. Pero hasta la fecha, la capacidad de producir inventarios completos de proteínas sigue siendo un objetivo difícil de alcanzar.
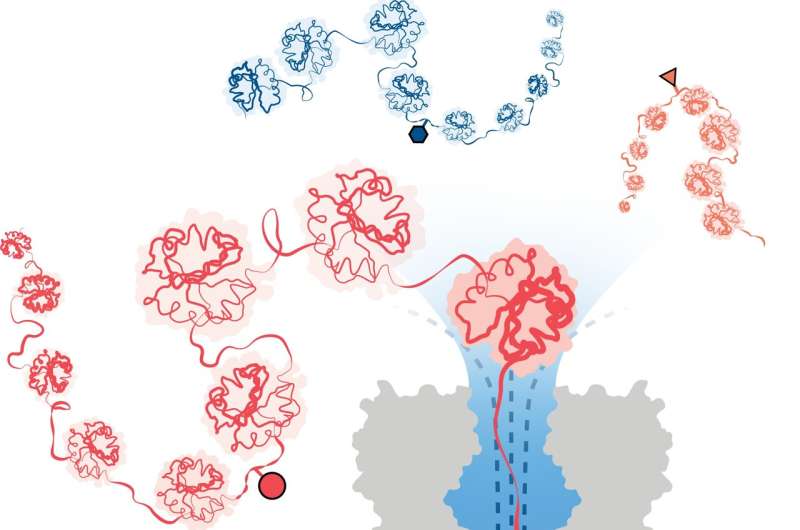
Para superar esto, un equipo dirigido por investigadores del Departamento de Química de la Universidad de Oxford ha desarrollado con éxito un método para el análisis de proteínas basado en tecnología de secuenciación de ADN/ARN con nanoporos. En este enfoque, un flujo direccional de agua captura y despliega proteínas 3D en cadenas lineales que se alimentan a través de poros diminutos, lo suficientemente anchos para que pase una sola molécula de aminoácido.
Las variaciones estructurales se identifican midiendo los cambios en una corriente eléctrica aplicada a través del nanoporo. Diferentes moléculas causan diferentes interrupciones en la corriente, dándoles una firma única.
El equipo demostró con éxito la eficacia del método para detectar tres modificaciones diferentes de PTM (fosforilación, glutatión y glicosilación) a nivel de una sola molécula para cadenas de proteínas de más de 1200 residuos de longitud. Estos incluyeron modificaciones profundas dentro de la secuencia de la proteína. Es importante destacar que el método no requiere el uso de etiquetas, enzimas o reactivos adicionales.
Según el equipo de investigación, el nuevo método de caracterización de proteínas podría integrarse fácilmente en los dispositivos portátiles de secuenciación de nanoporos existentes para permitir a los investigadores crear rápidamente inventarios de proteínas de células y tejidos individuales. Esto podría facilitar el diagnóstico en el lugar de atención, permitiendo la detección personalizada de variantes de proteínas específicas asociadas con enfermedades como el cáncer y los trastornos neurodegenerativos.
El profesor Yujia Qing (Departamento de Química de la Universidad de Oxford), autor colaborador del estudio, dijo:"Este método simple pero poderoso abre numerosas posibilidades. Inicialmente, permite el examen de proteínas individuales, como las involucradas en enfermedades específicas. A largo plazo, el método tiene el potencial de crear inventarios ampliados de variantes de proteínas dentro de las células, desbloqueando conocimientos más profundos sobre los procesos celulares y los mecanismos de las enfermedades".
El profesor Hagan Bayley (Departamento de Química de la Universidad de Oxford), autor colaborador y cofundador de Oxford Nanopore Technologies, añadió:"La capacidad de señalar e identificar modificaciones postraduccionales y otras variaciones de proteínas a nivel de una sola molécula es inmensamente prometedora". para mejorar nuestra comprensión de las funciones celulares y las interacciones moleculares. También puede abrir nuevas vías para la medicina, el diagnóstico y las intervenciones terapéuticas personalizadas".
Este trabajo se llevó a cabo en colaboración con el grupo de investigación del mecanobiólogo Sergi García-Maynes en el King's College de Londres y el Instituto Francis Crick.
Más información: Pablo Martin-Baniandres et al, Detección de nanoporos sin enzimas de modificaciones postraduccionales dentro de polipéptidos largos, Nature Nanotechnology (2023). DOI:10.1038/s41565-023-01462-8
Información de la revista: Nanotecnología de la naturaleza
Proporcionado por la Universidad de Oxford