Un equipo de químicos, microbiólogos y físicos de la Universidad de Cambridge en el Reino Unido ha desarrollado una forma de utilizar nanoporos de estado sólido y códigos de barras de ADN multiplexados para identificar proteínas mal plegadas, como las implicadas en trastornos neurodegenerativos, en muestras de sangre. En su estudio, publicado en el Journal of the American Chemical Society , el grupo utilizó técnicas de códigos de barras de ADN multiplexado para superar los problemas con las técnicas de filtrado de nanoporos para aislar oligómeros dañinos.
Investigaciones anteriores han demostrado que la presencia de oligómeros dañinos en el cerebro puede provocar un plegamiento incorrecto de proteínas asociadas con enfermedades neurodegenerativas como el Parkinson y el Alzheimer. Los investigadores médicos han estado buscando una forma de detectarlos en la sangre para diagnosticar enfermedades neurodegenerativas y realizar un seguimiento de su progresión una vez confirmada.
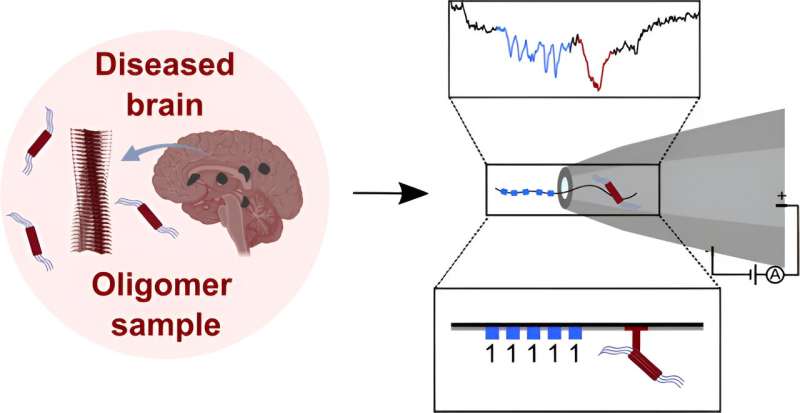
Desafortunadamente, encontrarlos en mezclas complejas como la sangre ha demostrado ser una tarea desalentadora. Un enfoque ha resultado prometedor:el uso de sensores de nanoporos, pero hasta la fecha no pueden rastrear los oligómeros objetivo mientras aceleran a través de sensores de nanoporos de estado sólido personalizables. En este nuevo esfuerzo, el equipo de investigación superó este problema mediante el uso de nanoestructuras de ADN personalizables.
En su trabajo, el equipo de investigación unió proteínas a sus nanoestructuras de ADN hechas a medida como un medio para crear una especie de "código de barras" que podría usarse para identificar moléculas utilizando sensores de nanoporos de estado sólido. Para usar su código de barras, colocaron un fragmento de ADN que había sido etiquetado con una sustancia química que se une solo a los oligómeros objetivo, lo que resultó en un pico adicional, lo que permitió aislar el objetivo y su caracterización completa.
Luego, el equipo probó su técnica utilizando oligómeros de α-sinucleína, el tipo de proteínas involucradas en el plegamiento en la enfermedad de Parkinson, y pudo aislarlos según lo deseado en condiciones de laboratorio. También midieron la tasa de formación de oligómeros. Luego probaron la técnica en un entorno más cercano a las condiciones biológicas y descubrieron que el rendimiento no se veía afectado. Sugieren que su técnica ofrece la promesa de un futuro proceso de detección para pacientes con riesgo de desarrollar trastornos neurodegenerativos.
Más información: Sarah E. Sandler et al, Caracterización digital multiplexada de oligómeros de proteínas mal plegadas mediante nanoporos de estado sólido, Revista de la Sociedad Química Estadounidense (2023). DOI:10.1021/jacs.3c09335
Información de la revista: Revista de la Sociedad Química Estadounidense
© 2023 Red Ciencia X