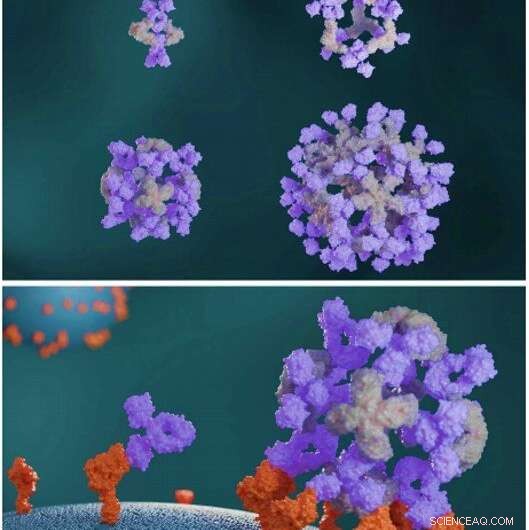
Las proteínas diseñadas ensamblan anticuerpos en grandes arquitecturas simétricas. Las proteínas de agrupamiento de anticuerpos diseñadas (gris claro) ensamblan los anticuerpos (violeta) en diversas arquitecturas de nanocajas (arriba). Las nanocajas de anticuerpos mejoran la señalización celular en comparación con los anticuerpos libres (abajo). Crédito:Ciencia, doi:10.1126 / science.abd9994
En un nuevo informe ahora publicado en Ciencias , Robby Divine y un equipo de investigación interdisciplinario en el departamento de bioquímica, medicina regenerativa, y vacunas y enfermedades infecciosas en la Universidad de Washington, EE. UU. y la Facultad de Medicina, en la Universidad de Ciencias Médicas de Teherán, Irán llevó a cabo diseños computacionales de nanocajas para ensamblar anticuerpos en arquitecturas precisas. Durante la construcción, un componente estructural formó una fusión de anticuerpo o ligando Fc y el segundo diseño formó un homooligómero de unión a anticuerpo para impulsar el ensamblaje de nanocajas con diferentes valencias y simetría. El equipo planteó la hipótesis de cómo este proceso también puede aumentar la neutralización de un pseudovirus; síndrome respiratorio agudo severo coronavirus-2 (SARS-COV-2) a través de anticuerpos monoclonales α-SARS-COV-2 y proteínas de fusión Fc-enzima convertidora de angiotensina 2 (ACE-2).
Anticuerpos en la investigación médica
Los anticuerpos que se unen específicamente a objetivos de interés juegan un papel central en la investigación biomédica y la medicina. Los investigadores pueden generar grupos de anticuerpos uniendo genéticamente fragmentos de anticuerpos para mejorar la señalización. Actualmente es difícil formar conjuntos de anticuerpos con una variedad de arquitecturas y valencias precisas. En este trabajo, Divine et al. proteínas diseñadas computacionalmente que ensamblan anticuerpos en arquitecturas precisas con diferentes valencias y simetrías. El equipo planteó la hipótesis de que tales diseños podrían conducir de manera robusta anticuerpos arbitrarios a nanocajas homogéneas y estructuralmente bien definidas para efectos pronunciados en la señalización celular. Los investigadores diseñaron proteínas para impulsar el ensamblaje de anticuerpos arbitrarios en ensamblajes simétricos con estructuras bien definidas. Para esto, fusionaron rígidamente tres tipos de unidades de "bloques de construcción" que contienen dominios de unión a Fc de anticuerpos, conectores helicoidales repetidos y módulos cíclicos formadores de oligómeros. En su arquitectura, la unidad de unión a Fc se posicionó con el dímero de anticuerpo C2, el homo-oligómero cíclico formó el segundo eje de simetría cíclica en la nanojaula, y el conector de repetición helicoidal unía el anticuerpo y los ejes de simetría homooligoméricos cíclicos en la orientación correcta para formar las nanocajas de anticuerpos denominadas AbC.
Diseño de nanocaja de anticuerpos (AbC). (A) Se especifica la geometría poliédrica. En el sentido de las agujas del reloj desde la parte superior izquierda:icosaédrico, diedro octaédrico, y se muestran geometrías tetraédricas. (B) Un modelo de anticuerpo Fc de hIgG1 se alinea con uno de los ejes C2 (en este caso, se muestra un diedro D2). (C) Los aglutinantes de anticuerpo Fc se fusionan con conectores repetidos helicoidales que luego se fusionan con la subunidad monomérica de oligómeros cíclicos helicoidales. Se muestrean todas las combinaciones de bloques de construcción y uniones de bloques de construcción (recuadro inferior sombreado en gris; los números entre paréntesis se refieren a la cantidad de bloques de construcción disponibles). (D) Se verifican las fusiones tripartitas para asegurar la alineación exitosa de los ejes de simetría C2 Fc con el de la arquitectura poliédrica (en el caso de la simetría D2 que se muestra aquí, los ejes C2 deben cruzarse en un ángulo de 90 °). (E) Las fusiones que pasan los criterios geométricos avanzan con el rediseño de la cadena lateral, dónde, por ejemplo, Los aminoácidos están optimizados para garantizar que los residuos del empaquetamiento del núcleo sean apolares y empaquetados estrechamente y que los residuos expuestos al solvente sean polares. (F) Los oligómeros que forman AbC diseñados se expresan bacterianamente, purificado y ensamblado con anticuerpo Fc o IgG. Crédito:Ciencia, doi:10.1126 / science.abd9994 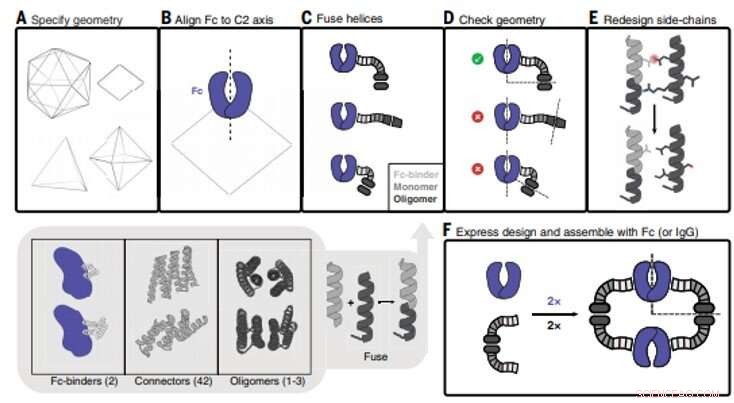
Para formar diseños de jaulas de anticuerpos (AbC), Divine et al. moléculas de unión de dominio constante de anticuerpo rígidamente fusionadas a oligómeros cíclicos usando dominios espaciadores helicoidales. Facilitaron el proceso a través de dominios espaciadores helicoidales para que los ejes de simetría del anticuerpo dimérico y el oligómero cíclico pudieran estar en orientaciones que generaran diversas arquitecturas diédricas o poliédricas. Los científicos optimizaron las regiones de unión entre los bloques de construcción conectados para plegarse a las estructuras diseñadas. El enfoque de fusión se basó en grandes conjuntos de bloques de construcción con muchos sitios de fusión posibles por bloque de construcción como criterios geométricos estrictos para formar la arquitectura simétrica deseada. El equipo utilizó un método computacional para el diseño de jaulas de anticuerpos para generar diédrico, tetraédrico AbC octaédricos e icosaédricos y utilizó una convención de nomenclatura para describir las arquitecturas finales de nanocajas. Divine et al. Luego expresaron genes sintéticos que codifican los diseños en cultivos bacterianos de Escherichia coli. Por ejemplo, los diseños exitosos incluyeron decaedro D-2 (tres diseños), T-32 (un diseño) y 152 icosaédricos (dos diseños) que contienen dos, seis, 12 o 30 anticuerpos, respectivamente. Caracterizaron los Fc AbC mediante microscopía electrónica y dispersión de rayos X de ángulo pequeño. Las nanojaulas reconstruidas estaban en estrecha concordancia con los modelos de diseño computacional. Para evaluar la estabilidad de las nanojaulas, Divine et al. utilizaron lecturas de dispersión de luz dinámica para obtener una estabilidad alentadora que permita la caracterización de su impacto biológico a continuación.
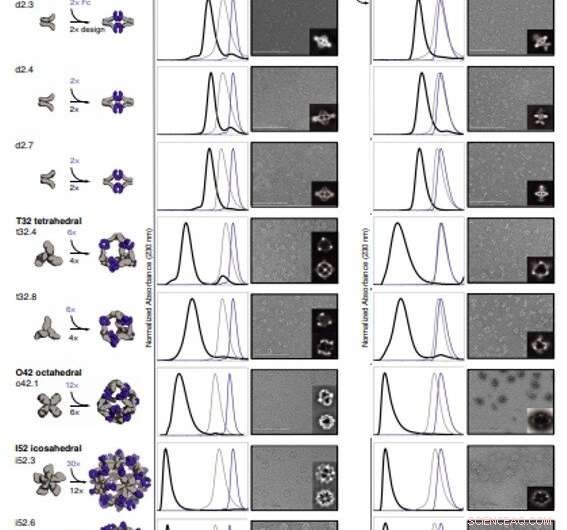
Caracterización estructural de AbCs. (A) Modelos de diseño, con anticuerpo Fc (violeta) y oligómeros formadores de AbC diseñados (gris). (B) Superposición de trazas SEC representativas de ensamblaje formadas por diseño de mezcla y Fc (negro) con las de los componentes individuales en gris (diseño) o violeta (Fc). (C) Imágenes EM con promedios de clase 2D sin referencia en recuadro; todos los datos son de NS-EM, a excepción de los diseños o42.1 e i52.3 (cryo-EM). (D y E) Micrografías representativas de SEC (D) y NS-EM con promedios de clase 2D sin referencia (E) de las mismas jaulas de anticuerpos diseñadas ensambladas con IgG1 humana completa (con las 2 regiones Fab intactas). En todos los casos EM mostrados en (C) y (E), Los ensamblajes se purificaron primero a través de SEC, y las fracciones correspondientes al pico más a la izquierda se combinaron y se utilizaron para la formación de imágenes para eliminar cualquier diseño o componente de Ig en exceso. Barras de escala, 200 nm. Crédito:Ciencia, doi:10.1126 / science.abd9994
Efectos sobre la señalización celular
Los AbC diseñados proporcionaron una plataforma general para comprender el efecto de la valencia y la geometría del compromiso del receptor en la activación de la vía de señalización. La amplia gama de anticuerpos de unión al receptor y ligandos naturales formados con el método AbC desarrollado en este trabajo permitió un sondeo rápido y sistemático del efecto de la geometría y la valencia de la asociación de la subunidad del receptor en la señalización celular para casi cualquier vía. Para explorar la practicidad de este enfoque, Divine et al. ensamblaron anticuerpos o fusiones de ligando Fc dirigidos a una variedad de vías de señalización en jaulas de anticuerpos (AbC) y estudiaron su impacto en la señalización. Por ejemplo, los abc, formado con una apoptosis (muerte celular) inducida por un anticuerpo dirigido al receptor de muerte de líneas de células tumorales que hasta ahora no habían sido afectadas por el anticuerpo soluble o el ligando nativo. Además, el ensamblaje de Fc-fusiones o anticuerpos en AbC, permitió mejorar la señalización de la vía de la angiopoyetina, Señalización de CD-40 y proliferación de células T. La formación de AbC permitió además la neutralización de un pseudovirus in vitro, como el síndrome respiratorio agudo severo coronavirus 2.
Reconstrucciones 3D de AbCs formados con Fc. Los modelos de diseño computacional (representación de dibujos animados) de cada AbC se ajustan a la densidad 3D determinada experimentalmente de EM. Cada nanojaula se ve a lo largo de un eje de simetría desocupado (izquierda) y se gira para mirar hacia abajo uno de los ejes de simetría C2 ocupados por el Fc (derecha). Las reconstrucciones tridimensionales de o42.1 e i52.3 son del análisis crio-EM, todos los demás son de NS-EM. Crédito:Ciencia, doi:10.1126 / science.abd9994 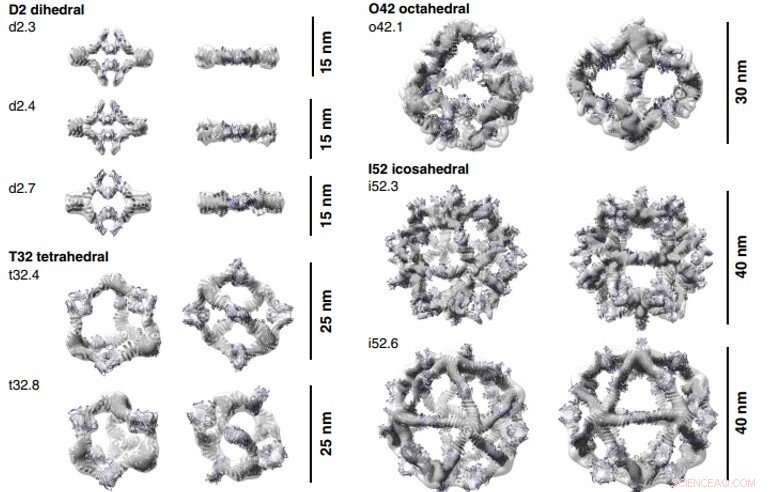
El método detallado en este trabajo, fue más allá de los esfuerzos previos de diseño computacional para crear nanomateriales de proteínas que integran forma y función. Por lo tanto, los AbC utilizaron anticuerpos como componentes estructurales y funcionales para lograr una amplia gama de geometrías y orientaciones. Esta estrategia es aplicable para diseñar vacunas con nanocajas ensambladas con glicoproteínas virales utilizando componentes que terminan en dominios de unión a glicoproteínas para maximizar la proximidad de los sitios activos. De este modo, Robby Divine y sus colegas diseñaron múltiples proteínas formadoras de jaulas de anticuerpos para agrupar con precisión cualquier anticuerpo de unión a proteínas en nanojaulas a través de valencia y geometría controladas. El equipo usó dos, seis y 12 o 30 anticuerpos dentro de los AbC simplemente mezclando el anticuerpo con la proteína diseñada correspondiente sin modificaciones covalentes adicionales. Los científicos agregaron anticuerpos que se unen al receptor o neutralizan el virus en los ABC para mejorar su actividad biológica en diversos sistemas celulares. El equipo espera que este resultado para el ensamblaje rápido de anticuerpos dentro de nanocajas ordenadas sin modificaciones covalentes tenga amplias aplicaciones en la investigación y la medicina.

Los AbC activan las vías de señalización de la apoptosis y la angiogénesis. (A y B) La caspasa-3/7 es activada por los AbC formados con el anticuerpo a-DR5 (A), pero no el anticuerpo libre, en células de cáncer renal RCC4 (B). (C y D) a-DR5 AbCs (C), pero no controles Fc AbC (D), reducir la viabilidad celular 4 días después del tratamiento. Los (E) a-DR5 AbC reducen la viabilidad 6 días después del tratamiento. (F y G) o42.1 a-DR5 AbC mejoran la escisión de PARP, un marcador de señalización apoptótica; (G) es una cuantificación de (F) en relación con el control de PBS. (H) El dominio F de la angiopoyetina-1 se fusionó con Fc (A1F-Fc) y se ensambló en AbC octaédricos (o42.1) e icosaédricos (i52.3). (I) Las transferencias Western representativas muestran que los AbC de A1F-Fc, pero no controles, aumente las señales pAKT y pERK1 / 2. (J) Cuantificación de (I):la cuantificación de pAKT se normaliza a la señalización de o42.1 A1F-Fc (sin señal de pAKT en el control de PBS); pERK1 / 2 se normaliza a PBS. (K) Los AbC de A1F-Fc aumentan la estabilidad vascular después de 72 horas. (Izquierda) Cuantificación de la estabilidad vascular en comparación con PBS. (Right) Representative images; scale bars, 100 mm. All error bars represent means ± SEM; means were compared using analysis of variance and Dunnett post-hoc tests (tables S8 and S9). *P ≤ 0.05; **P ≤ 0.01; ***P ≤ 0.001; ****P ≤ 0.0001 Credit:Science, doi:10.1126/science.abd9994
© 2021 Science X Network