Imagen de microscopio de fuerza atómica en color de estructuras de árboles aleatorias autoensambladas en la superficie de matrices de mosaicos de ADN. Cada árbol tiene un solo bucle como "raíz". Crédito:Caltech / Grigory Tikhomirov, Philip Petersen y Lulu Qian.
Muchos sistemas autoorganizados en la naturaleza explotan una sofisticada combinación de procesos deterministas y aleatorios. No hay dos árboles exactamente iguales porque el crecimiento es aleatorio, pero una secuoya se puede distinguir fácilmente de una jacaranda ya que las dos especies siguen diferentes programas genéticos. El valor de la aleatoriedad en los organismos biológicos no se comprende completamente, pero se ha planteado la hipótesis de que permite tamaños de genoma más pequeños, porque no se deben codificar todos los detalles. La aleatoriedad también proporciona la variación subyacente a la evolución adaptativa.
En contraste con la biología, la ingeniería rara vez se aprovecha del poder de la aleatoriedad para fabricar estructuras complejas. Ahora, un grupo de científicos de Caltech ha demostrado que la aleatoriedad en el autoensamblaje molecular se puede combinar con reglas deterministas para producir nanoestructuras complejas a partir del ADN.
La obra, hecho en el laboratorio de la profesora asistente de bioingeniería Lulu Qian, aparece en la edición del 28 de noviembre de la revista Nanotecnología de la naturaleza .
Los seres vivos usan el ADN para almacenar información genética, pero el ADN también se puede utilizar como un bloque de construcción químico robusto para la ingeniería molecular. Las cuatro moléculas complementarias que componen el ADN, llamados nucleótidos, se unen solo de maneras específicas:A se unen con T, y los de G se unen con los de C. En 2006, Paul Rothemund (BS '94), profesor investigador de bioingeniería, ciencias de la computación y matemáticas, y computación y sistemas neuronales en Caltech, inventó una técnica llamada origami de ADN que aprovecha la coincidencia entre cadenas largas de nucleótidos de ADN, doblándolos en todo, desde obras de arte a nanoescala hasta dispositivos de administración de fármacos. Las estructuras autoensambladas formadas a través del origami de ADN pueden ser funcionales por sí mismas o pueden usarse como plantillas para organizar otras moléculas funcionales, como los nanotubos de carbono, proteínas, nanopartículas de metal, y tintes orgánicos, con una capacidad de programación y una precisión espacial sin precedentes.
Usando origami de ADN como un bloque de construcción, los investigadores han creado nanoestructuras de ADN más grandes, como matrices periódicas de mosaicos de origami. Sin embargo, porque el componente básico se repite en todas partes, la complejidad de los patrones que se pueden formar en estas estructuras más grandes es bastante limitada. Los procesos de ensamblaje completamente deterministas, que controlan el diseño de cada baldosa individual y su posición distinta en la matriz, pueden dar lugar a patrones complejos, pero estos procesos no se escalan bien. En cambio, si solo están involucrados procesos aleatorios y las características globales de la matriz no están controladas por reglas de diseño, es imposible crear patrones complejos con las propiedades deseadas sin generar simultáneamente una gran fracción de moléculas no deseadas que se desperdician. Hasta el trabajo de Qian y sus colegas, La combinación de procesos deterministas con procesos aleatorios nunca se había explorado sistemáticamente para crear nanoestructuras de ADN complejas.

Izquierda, Las baldosas Truchet tienen dos arcos que son rotacionalmente asimétricos. Derecha, juegos de mesa populares inspirados en las fichas de Truchet. Crédito:Cortesía de L. Qian
"Estábamos buscando principios de autoensamblaje molecular que abarquen tanto aspectos deterministas como aleatorios, ", dice Qian." Desarrollamos un conjunto simple de reglas que permiten que los mosaicos de ADN se unan al azar, pero solo en patrones controlados específicos ".
El enfoque implica diseñar patrones en mosaicos individuales, modular las proporciones de diferentes mosaicos, y determinar qué baldosas se pueden unir durante el autoensamblaje. Esto conduce a características emergentes a gran escala con propiedades estadísticas ajustables, un fenómeno que los autores denominan "trastorno programable".
"Las estructuras que podemos construir tienen aspectos aleatorios programables, "dice Grigory Tikhomirov, un erudito postdoctoral senior en biología e ingeniería biológica, y autor principal del artículo. "Por ejemplo, podemos hacer estructuras que tengan líneas que tomen caminos aparentemente aleatorios, pero podemos asegurarnos de que nunca se crucen y eventualmente se cierren en bucles ".
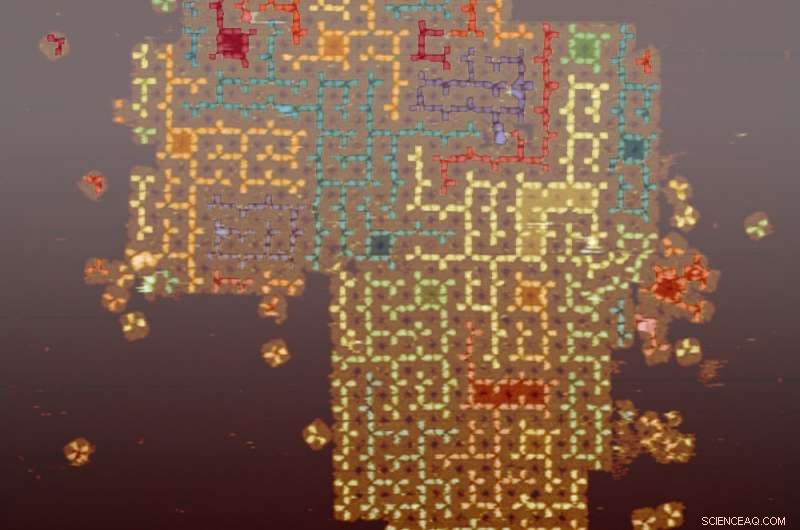
Además de los bucles, el equipo eligió otros dos ejemplos, laberintos y árboles, para demostrar que muchas propiedades no triviales de estas estructuras pueden controlarse mediante reglas locales simples. Encontraron estos ejemplos interesantes porque loop, laberinto, y las estructuras de los árboles existen ampliamente en la naturaleza en múltiples escalas. Por ejemplo, los pulmones son estructuras de árboles en una escala de milímetro a centímetro, y las dendritas neuronales son estructuras de árboles en una escala de micrómetro a milímetro. Las propiedades controladas que mostraron incluyen las reglas de ramificación, las direcciones de crecimiento, la proximidad entre redes adyacentes, y la distribución del tamaño.
El grupo se inspiró primero en los azulejos clásicos de Truchet, que son baldosas cuadradas con dos arcos de ADN simétricos en diagonal en la superficie. Hay dos orientaciones rotacionalmente asimétricas del patrón de arco. Permitiendo una elección aleatoria de las dos orientaciones de mosaico en cada ubicación en la matriz, el patrón continuará a través de los azulejos vecinos, ya sea convirtiéndose en bucles de varios tamaños o saliendo de un borde de la matriz.

Bucle autoensamblado, laberinto, y estructuras de árboles en la superficie de las matrices de mosaicos de ADN. Fila superior, laberintos aleatorios con uniones de tres y cuatro vías de distancias variables entre uniones adyacentes versus uniones de tres vías de una distancia fija entre uniones adyacentes. Fila del medio, árboles aleatorios (cada árbol tiene un solo bucle como "raíz") con ramas más largas de diferentes longitudes versus ramas más cortas de longitudes fijas. Fila inferior, bucles aleatorios con longitudes ajustables y número de cruces. Crédito:L. Qian
Para crear matrices de Truchet a escala molecular, el equipo utilizó la técnica de origami de ADN para doblar el ADN en mosaicos cuadrados y luego diseñó las interacciones entre estos mosaicos para alentarlos a autoensamblarse en grandes matrices bidimensionales.
"Debido a que todas las moléculas chocan entre sí mientras flotan en un tubo de ensayo durante el proceso de autoensamblaje, las interacciones deben ser lo suficientemente débiles para permitir que los mosaicos se reorganicen y eviten quedar atrapados en configuraciones no deseadas, "dice Philip Petersen, estudiante de posgrado en el laboratorio de Qian y coautor del artículo. "Por otra parte, las interacciones deben ser lo suficientemente específicas para que las interacciones deseadas sean siempre mucho más preferidas a las no deseadas, interacciones espurias ".
Surgen diferentes tipos de patrones globales cuando los mosaicos se marcan con diferentes patrones locales. Por ejemplo, si cada mosaico orientado aleatoriamente lleva una "T" en lugar de dos arcos, el patrón global es un laberinto con ramas y bucles en lugar de solo bucles. Si las reglas de autoensamblaje restringen la posible orientación relativa de las baldosas "T" vecinas, es posible asegurarse de que, además de una única "raíz, "las ramas de los laberintos nunca se cierran en bucles, produciendo árboles. Para explorar la generalidad completa de estos principios, El equipo de Qian desarrolló un lenguaje de programación para objetos de origami de ADN aleatorios.
"Con este lenguaje de programación, el proceso de diseño comienza con una descripción de alto nivel de los mosaicos y las matrices, que se puede traducir automáticamente a diagramas de matriz abstractos y simulaciones numéricas, luego pasa al diseño de mosaicos de origami de ADN que incluye cómo los mosaicos interactúan entre sí en sus bordes. Finalmente, diseñamos secuencias de ADN, "Dice Qian." Con estas secuencias de ADN, Es sencillo para los investigadores ordenar las hebras de ADN, mezclarlos en un tubo de ensayo, esperar a que las moléculas se autoensamblen en las estructuras diseñadas durante la noche, y obtener imágenes de las estructuras utilizando un microscopio de fuerza atómica ".
El método de desorden programable del grupo tiene diversas aplicaciones futuras. Por ejemplo, podría usarse para construir entornos de prueba complejos para robots moleculares cada vez más sofisticados:máquinas a nanoescala basadas en ADN que pueden moverse sobre una superficie, recoger o dejar proteínas u otros tipos de moléculas como cargas, y tomar decisiones sobre navegación y acciones.
"Las aplicaciones potenciales son mucho más amplias, ", Agrega Qian. Desde la década de 1990, Se han utilizado cadenas unidimensionales aleatorias de polímeros para revolucionar la síntesis química y de materiales. entrega de medicamentos, y química de ácidos nucleicos mediante la creación de vastas bibliotecas combinatorias de moléculas candidatas y luego seleccionando o desarrollando las mejores en el laboratorio. “Nuestro trabajo extiende el mismo principio a redes bidimensionales de moléculas y ahora crea nuevas oportunidades para fabricar dispositivos moleculares más complejos organizados por nanoestructuras de ADN, " ella dice.
El artículo se titula "Desorden programable en teselaciones de ADN aleatorias".