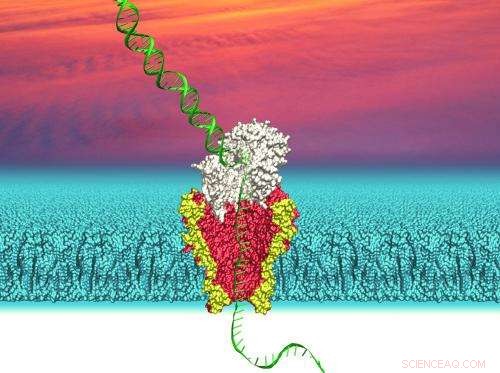
Ilustración de un nanoporo derivado de un canal de membrana bacteriana modificado genéticamente que se utiliza para secuenciar el ADN. Crédito:Ian Derrington
(Phys.org) —Una tecnología de bajo costo puede hacer posible leer largas secuencias de ADN mucho más rápidamente que las técnicas actuales.
La investigación avanza una tecnología, llamada secuenciación de ADN de nanoporos. Si se perfecciona, algún día podría usarse para crear dispositivos portátiles capaces de identificar rápidamente secuencias de ADN a partir de muestras de tejido y el medio ambiente. dijeron los investigadores de la Universidad de Washington que desarrollaron y probaron el enfoque.
"Una de las razones por las que la gente está tan entusiasmada con la secuenciación de ADN de nanoporos es que la tecnología podría usarse para crear dispositivos similares a los 'tricorders' para detectar patógenos o diagnosticar trastornos genéticos rápidamente y en el lugar". "dijo Andrew Laszlo, autor principal y estudiante de posgrado en el laboratorio de Jen Gundlach, un profesor de física de la Universidad de Washington que dirigió el proyecto.
El artículo "Decodificación de lecturas largas de secuenciación de nanoporos de ADN natural" describe la nueva técnica. Aparece el 25 de junio en la edición avanzada en línea de la revista. Biotecnología de la naturaleza .
La mayoría de las tecnologías de secuenciación de genes actuales requieren trabajar con fragmentos cortos de ADN, típicamente de 50 a 100 nucleótidos de longitud. Estos deben ser procesados por grandes secuenciadores en un laboratorio. El engorroso proceso puede tardar días o semanas en completarse.
La tecnología Nanopore aprovecha lo pequeño, estructuras en forma de túnel que se encuentran en las membranas bacterianas. En naturaleza, tales poros permiten que las bacterias controlen el flujo de nutrientes a través de sus membranas.
Un investigador de la Universidad de Washington utilizó el nanoporo Mycobacterium smegmatis porin A (MspA). Este poro bacteriano ha sido alterado genéticamente para que la parte más estrecha del canal tenga un diámetro de aproximadamente un nanómetro, o la mil millonésima parte de un metro. Esto es lo suficientemente grande como para que pase una sola hebra de ADN. El nanoporo modificado se inserta luego en una membrana que separa dos soluciones salinas para crear un canal que conecta las dos soluciones.
Para leer una secuencia de ADN con este sistema, Se aplica un pequeño voltaje a través de la membrana para hacer que los iones de la solución salina fluyan a través del nanoporo. El flujo de iones crea una corriente medible. Si se agrega una hebra de ADN a la solución en un lado de la membrana y luego entra en un poro, las voluminosas moléculas de ADN impedirán el flujo del ion mucho más pequeño y, por lo tanto, alterarán la corriente. Cuánto cambia la corriente depende de qué nucleótidos:las moléculas individuales de adenina, guanina la citosina y la timina que forman la cadena de ADN se encuentran dentro del poro. La detección de cambios en la corriente puede revelar qué nucleótidos pasan a través del canal del nanoporo en un momento dado.
Dado que la técnica se propuso por primera vez en la década de 1990, Los investigadores esperaban que la secuenciación de ADN de nanoporos ofreciera un alternativa rápida a la secuenciación genética actual. Pero sus intentos se han visto frustrados por varios desafíos. Es difícil identificar cada nucleótido uno por uno a medida que atraviesan el nanoporo. En lugar de, los investigadores tienen que trabajar con cambios en la corriente asociados con cuatro nucleótidos a la vez. Además, algunos nucleótidos pueden pasarse por alto o leerse más de una vez. Como consecuencia, La tecnología actual de secuenciación de nanoporos produce una lectura imprecisa de una secuencia de ADN.
Los investigadores de la UW describen cómo evitaron estos problemas. Los investigadores primero identificaron las firmas electrónicas de todas las combinaciones de nucleótidos posibles con los cuatro nucleótidos que componen el ADN, un total de 256 combinaciones en total (4 x 4 x 4 x 4).
Luego crearon algoritmos informáticos para hacer coincidir los cambios actuales generados cuando un segmento de ADN pasa a través del poro con los cambios actuales esperados de las secuencias de ADN de genes y genomas conocidos almacenados en una base de datos informática. Una coincidencia mostraría que la secuencia del ADN que pasa a través del poro es idéntica o cercana a la secuencia de ADN almacenada en la base de datos. Todo el proceso tomaría de minutos a algunas horas, en lugar de semanas.
Para probar este enfoque, los investigadores utilizaron su sistema de nanoporos para leer la secuencia del bacteriófago Phi X 174, un virus que infecta bacterias y que se usa comúnmente para evaluar nuevas tecnologías de secuenciación del genoma. Descubrieron que el enfoque leía de manera confiable las secuencias de ADN del bacteriófago y podía leer secuencias de hasta 4, 500 nucleótidos.
"Esta es la primera vez que alguien ha demostrado que los nanoporos se pueden utilizar para generar firmas interpretables correspondientes a secuencias de ADN muy largas de genomas del mundo real". "dijo el coautor Jay Shendure, un profesor asociado de ciencias del genoma de la UW cuyo laboratorio desarrolla aplicaciones de tecnologías de secuenciación del genoma. "Es un gran paso adelante".
Debido a que la técnica se basa en hacer coincidir las lecturas con las bases de datos de genes y genomas previamente secuenciados, todavía no se puede utilizar para secuenciar un gen o genoma recién descubierto, los investigadores dijeron, pero con algunos refinamientos, ellos agregaron, debería ser posible mejorar el desempeño en esta área. Para acelerar la investigación sobre esta nueva tecnología, los científicos están haciendo sus métodos, datos y algoritmos informáticos totalmente disponibles para todos.
"A pesar de los obstáculos restantes, nuestra demostración de que un dispositivo de bajo costo puede leer de manera confiable las secuencias de ADN natural y puede interpretar segmentos de ADN de hasta 4, 500 nucleótidos de longitud representa un avance importante en la secuenciación de ADN de nanoporos, "Dijo Gundlach.