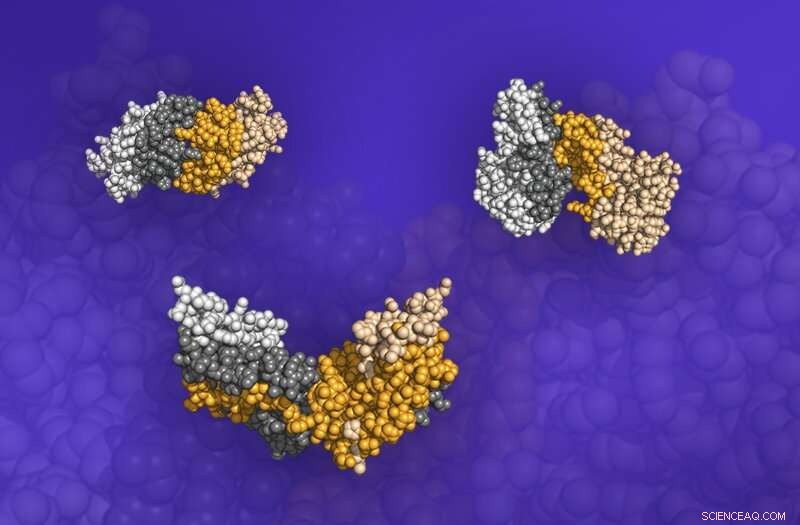
Tres dímeros estructuras proteicas que constan de dos proteínas unidas, de la base de datos de Dockground. Las interfaces en las que se encuentran las proteínas se muestran como regiones oscuras. Crédito:ORNL
¿Puede algo tan simple como la forma determinar completamente si las proteínas se unirán o no? Los científicos están encargando supercomputadoras para averiguarlo.
Un equipo dirigido por Sharon Glotzer, profesor distinguido y presidente del departamento de ingeniería química de la Universidad de Michigan (UM), usó la supercomputadora Summit de 200 petaflop en el Laboratorio Nacional Oak Ridge (ORNL) del Departamento de Energía de EE. UU. (DOE) para modelar interacciones de bloqueo y llave entre proteínas para estudiar sus comportamientos de unión. Los resultados, publicado en Materia blanda, reveló que algunas proteínas lo hacen, De hecho, enlazar basándose solo en la forma.
"Hemos demostrado que algo tan simple como la forma es capaz de predecir interacciones de proteínas que a veces son realmente complejas, "dijo Jens Glaser, científico computacional en el grupo de Computación Avanzada para Química y Materiales en Oak Ridge Leadership Computing Facility (OLCF). "Esta primera demostración nos ha llevado a creer que la forma ha sido un ingrediente poco apreciado en muchos procesos de ensamblaje de proteínas".
Los resultados podrían tener numerosas aplicaciones en la investigación biológica. Por ejemplo, el enfoque podría usarse para detectar medicamentos en busca de enfermedades o proporcionar a los científicos información sobre cómo usar proteínas como bloques de construcción para diseñar nuevos materiales biológicos.
"Este interesante estudio demuestra el poder de la complementariedad de formas en la predicción de interfaces proteína-proteína, "dijo la Dra. Stephanie McElhinny, Gerente de programa en el Laboratorio de Investigación del Ejército del Comando de Desarrollo de Capacidades de Combate del Ejército de EE. UU. refiriéndose a la relación espacial favorable entre dos proteínas de forma compatible. "Los modelos computacionales que predicen con precisión estas interfaces respaldarán el diseño futuro de materiales avanzados basados en proteínas con propiedades activas y receptivas, como los plásticos a base de proteínas que recogen la luz y que podrían funcionar como una hoja artificial para la generación de energía ".
Las supercomputadoras revelan que la forma es clave en algunas proteínas
Para que las proteínas se unan con éxito entre sí, uno de ellos actúa como ligando, una molécula que se adhiere a una proteína objetivo, y uno de ellos actúa como receptor, la molécula que recibe el ligando. Este proceso implica complejas interacciones químicas, en el que las moléculas comparten enlaces y cambian sus configuraciones al unirse.
El equipo de Glotzer quería ver si podían predecir esta unión molecular basándose solo en la forma, ignorando las interacciones entre proteínas. De una base de datos de más de 6, 000 pares de proteínas, el equipo probó 46 pares que se sabe que se unen entre sí y simuló su ensamblaje en Summit. El equipo realizó las simulaciones en el marco del programa INCITE (Innovative and Novel Computational Impact on Theory and Experiment).
Como varias pelotas de tenis lanzadas a un solo objetivo, las simulaciones modelaron múltiples ligandos lanzados a un solo, receptor objetivo fijo. De los 46 pares probados, encontraron 6 pares que funcionaron bien, más del 50 por ciento de las veces se ensamblaron con éxito basándose únicamente en sus formas complementarias.
"Observamos las interfaces donde las proteínas se unían para ver qué tan similares eran a sus interfaces de la vida real, y luego determinamos el límite para ver cuántos pares eran buenos predictores de las interfaces reales, "dijo Fengyi Gao, Doctor. candidato en la UM. "Descubrimos que el 13 por ciento de estos pares de proteínas podían unirse basándose únicamente en la forma".
Luego, el equipo construyó un modelo de aprendizaje automático que podría determinar qué proteínas pueden ensamblarse únicamente en función de la forma. La combinación de su modelo inicial con estas herramientas de aprendizaje automático les ayudará a comprender qué información se necesita para los pares de proteínas que no se pueden ensamblar basándose únicamente en la complementariedad de la forma.
Ejecutando proteínas en paralelo
Para modelar múltiples procesos de unión reversibles de 46 pares de proteínas bajo diferentes parámetros, necesitaban dos días de tiempo computacional y más de 3, 000 GPU, una cantidad que solo una supercomputadora como la Cumbre de OLCF podría proporcionar. La OLCF es una instalación para usuarios de la Oficina de Ciencias del DOE en ORNL.
Como parte del código computacional HOOMD-blue que se utilizó para ejecutar las simulaciones, Glaser, quien anteriormente fue investigador científico asistente en el grupo de Glotzer en la UM, desarrolló un algoritmo que simulaba las proteínas en presencia de muchas partículas pequeñas. Pero Glaser encontró una manera de modelar solo el movimiento de las proteínas en las que el equipo estaba interesado, evitando cálculos innecesarios y costosos para las moléculas de solvente que los rodean.
"Ejecuté el código en paralelo para que muchos parámetros diferentes, iteraciones del mismo sistema, y diferentes proteínas podrían distribuirse a través de las GPU, ", Dijo Glaser." Esto nos permitió hacer uso fácilmente de las capacidades de computación paralela de Summit ".
Usando Summit, el equipo capturó seis pares de proteínas que se unían basándose únicamente en la complementariedad de la forma, y uno de ellos logró la unión más del 94 por ciento de las veces.
"Fue bastante sorprendente para nosotros que un modelo tan simplificado pudiera seleccionar correctamente solo esa pose que asumen entre los muchos cientos o más poses que compiten, ", Dijo Glaser." Esperábamos que se necesitaría mucho más para reproducir la pose de unión real para estos pares de proteínas ".
Los modelos pueden ayudar en la detección de drogas
El equipo planea estudiar más proteínas que también puedan unirse en función de la forma o formar estructuras de orden incluso superior. El estudio actual del equipo exploró solo dímeros de proteínas, que constan de dos proteínas unidas entre sí, pero el equipo quiere conocer la limitación de cómo las formas de proteínas pueden evolucionar para formar estructuras de proteínas jerárquicas.
"Antes de hacer este estudio, De hecho, no esperaba que las proteínas pudieran formar dímeros basándose solo en la forma, "dijo Fengyi Gao, Doctor. candidato en la UM. "Pero ahora, hemos descubierto que esto funciona, y podemos estudiar estructuras más complejas o incluso combinar esto con otros enfoques, como el aprendizaje automático, para ver qué funciones necesitamos para habilitar el enlace correcto ".
El equipo espera poder predecir eventualmente la unión de las interfaces proteína-proteína en grupos de proteínas o estructuras de cristalización de proteínas.
"Creemos que podemos adaptar este enfoque a algo como la detección de drogas en el futuro, "Dijo Gao." Además de eso, esperamos que este modelo basado en formas pueda servir como base para estudiar el ensamblaje de proteínas en general ".