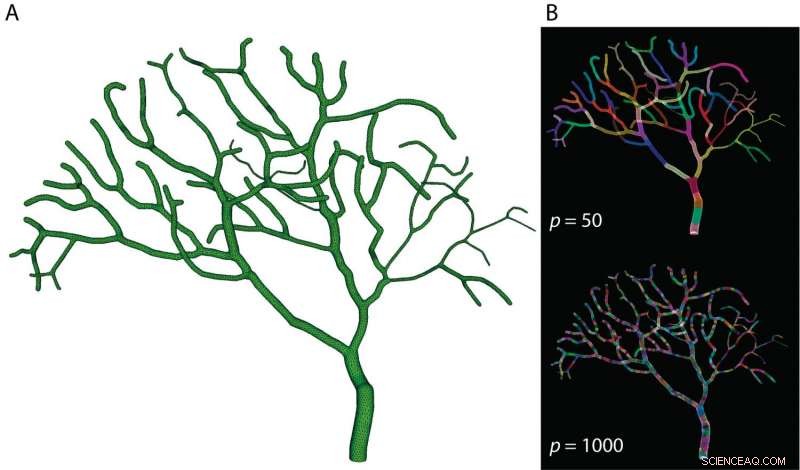
Al dividir el modelo de celda de Purkinje parcial (A) en 50 secciones (B-top) o 1000 secciones (B-bottom) y ejecutar cálculos de cada sección en paralelo en una supercomputadora, Los investigadores de OIST redujeron drásticamente el tiempo de simulación del modelo. Crédito:OIST
A diferencia de los neurocientíficos experimentales que se ocupan de las neuronas de la vida real, Los neurocientíficos computacionales utilizan simulaciones de modelos para investigar cómo funciona el cerebro. Si bien muchos neurocientíficos computacionales utilizan modelos matemáticos simplificados de neuronas, Los investigadores de la Unidad de Neurociencia Computacional de la Universidad de Graduados del Instituto de Ciencia y Tecnología de Okinawa (OIST) desarrollan software que modela neuronas al detalle de interacciones moleculares con el objetivo de obtener nuevos conocimientos sobre la función neuronal. Las aplicaciones del software tenían un alcance limitado hasta ahora debido a la intensa potencia computacional requerida para modelos neuronales tan detallados. pero recientemente el Dr. Weiliang Chen, Dr. Iain Hepburn, y el profesor Erik De Schutter publicaron dos artículos relacionados en los que describen la precisión y escalabilidad de su nuevo software computacional de alta velocidad, "PASOS Paralelos". Los hallazgos combinados sugieren que Parallel STEPS podría usarse para revelar nuevos conocimientos sobre cómo funcionan y se comunican las neuronas individuales entre sí.
El primer papel publicado en La Revista de Física Química en agosto de 2016, se centra en garantizar que la precisión de Parallel STEPS sea comparable a la de los métodos convencionales. En enfoques convencionales, Los cálculos asociados con las reacciones químicas neuronales y la difusión de moléculas se calculan secuencialmente en una unidad de procesamiento computacional o "núcleo". Sin embargo, El Dr. Iain Hepburn y sus colegas introdujeron un nuevo enfoque para realizar cálculos de reacción y difusión en paralelo que luego se pueden distribuir en múltiples núcleos de computadora, manteniendo la precisión de la simulación en un alto grado. La clave fue desarrollar un algoritmo original separado en dos partes:una que computara los eventos de reacciones químicas y la otra los eventos de difusión.
"Probamos una variedad de simulaciones de modelos, desde modelos de difusión simples hasta modelos biológicos realistas, y descubrimos que podíamos lograr un rendimiento mejorado utilizando un enfoque paralelo con una mínima pérdida de precisión. Esto demostró la idoneidad potencial del método a una escala mayor, "dice el Dr. Hepburn.
En un artículo relacionado publicado en Fronteras en neuroinformática este febrero El Dr. Weiliang Chen presentó los detalles de implementación de Parallel STEPS e investigó su desempeño y aplicaciones potenciales. Al dividir un modelo parcial de una célula de Purkinje, una de las neuronas más grandes del cerebro, en 50 a 1000 secciones y simular eventos de reacción y difusión para cada sección en paralelo en la supercomputadora Sango en OIST, El Dr. Chen y sus colegas vieron un aumento dramático en las velocidades de cálculo. Probaron este enfoque tanto en modelos simples como en modelos más complicados de estallidos de calcio en células de Purkinje y demostraron que la simulación paralela podría acelerar los cálculos en más de varios cientos de veces que los métodos convencionales.
"Juntos, Nuestros hallazgos muestran que la implementación de STEPS en paralelo logra mejoras significativas en el rendimiento, y buena escalabilidad, "dice el Dr. Chen." Modelos similares que antes requerían meses de simulación ahora se pueden completar en horas o minutos, lo que significa que podemos desarrollar y simular modelos más complejos, y aprender más sobre el cerebro en menos tiempo ".
Dr. Hepburn y Dr. Chen de la Unidad de Neurociencia Computacional de OIST, dirigido por el profesor Erik De Schutter, están colaborando activamente con Human Brain Project, una iniciativa mundial basada en École Polytechnique Fédérale de Lausanne (EPFL) en Suiza, para desarrollar una versión más robusta de Parallel STEPS que incorpore simulación de campo eléctrico de membranas celulares.
Hasta ahora, STEPS solo es capaz de modelar de manera realista partes de neuronas, pero con el apoyo de Parallel STEPS, la Unidad de Neurociencia Computacional espera desarrollar un modelo a gran escala de una neurona completa y, posteriormente, las interacciones entre neuronas en una red. Colaborando con el equipo de EPFL y haciendo uso de la supercomputadora IBM 'Blue Gene / Q' ubicada allí, su objetivo es lograr estos objetivos en un futuro próximo.
"Gracias a las supercomputadoras modernas podemos estudiar los eventos moleculares dentro de las neuronas de una manera mucho más transparente que antes, ", dice el profesor De Schutter." Nuestra investigación abre vías interesantes en la neurociencia computacional que vincula la bioquímica con la electrofisiología por primera vez ".