Las "superbacterias" resistentes a los antibióticos que pueden frustrar los esfuerzos para matarlas son una crisis de salud pública urgente y, según los Centros para el Control de Enfermedades, cada año ocurren más de 2,8 millones de infecciones resistentes a los antibióticos. Investigadores de todo el mundo están luchando para afrontar el desafío.
Un equipo colaborativo de investigadores dirigido por la Universidad de Massachusetts Amherst y que incluye científicos de la compañía biofarmacéutica Microbiotix anunció recientemente que habían aprendido con éxito cómo sabotear una pieza clave de la maquinaria que los patógenos utilizan para infectar sus células huésped y han desarrollado una prueba para identificar los medicamentos de próxima generación para atacar esta maquinaria celular vulnerable y lograr avances reales en la salud pública.
La estrategia típica en el tratamiento de infecciones microbianas es atacar al patógeno con un antibiótico, que actúa ingresando a la célula dañina y matándola. Esto no es tan fácil como parece, porque cualquier antibiótico nuevo debe ser soluble en agua, para que pueda viajar fácilmente a través del torrente sanguíneo, y oleoso, para cruzar la primera línea de defensa de la célula patógena, la membrana celular. El agua y el aceite, por supuesto, no se mezclan, y es difícil diseñar un fármaco que tenga suficientes características de ambas para ser eficaz.
La dificultad tampoco termina ahí, porque las células patógenas han desarrollado algo llamado "bomba de eflujo", que puede reconocer los antibióticos y luego excretarlos de forma segura fuera de la célula, donde no pueden causar ningún daño. Si el antibiótico no puede superar la bomba de expulsión y matar la célula, entonces el patógeno "recuerda" cómo se ve ese antibiótico específico y desarrolla bombas de expulsión adicionales para manejarlo de manera eficiente; de hecho, se vuelve resistente a ese antibiótico en particular.
Un camino a seguir es encontrar un nuevo antibiótico, o combinaciones de ellos, y tratar de ir un paso por delante de las superbacterias.
"O podemos cambiar nuestra estrategia", dice Alejandro Heuck, profesor asociado de bioquímica y biología molecular en la UMass Amherst y autor principal del artículo. "Soy químico y siempre me ha interesado mucho comprender cómo interactúan las moléculas químicas con los organismos vivos. En particular, he centrado mi investigación en las moléculas que hacen posible la comunicación entre un patógeno y la célula huésped que quiere invadir."
Heuck y sus colegas han estado particularmente interesados en un sistema de comunicación llamado sistema de secreción tipo 3, que, hasta ahora, parece ser una adaptación evolutiva exclusiva de los microbios patógenos.
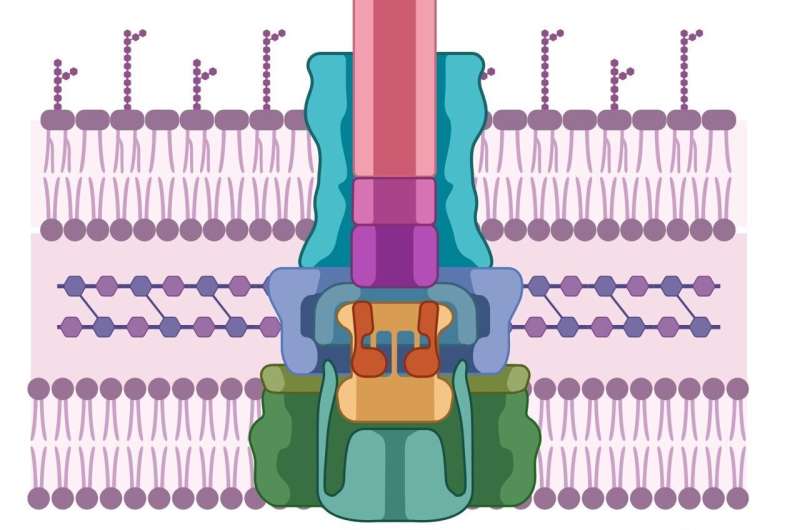
Al igual que la célula patógena, las células huésped también tienen paredes celulares gruesas y difíciles de penetrar. Para romperlos, los patógenos han desarrollado una máquina similar a una jeringa que primero secreta dos proteínas, conocidas como PopD y PopB. Ni PopD ni PopB individualmente pueden atravesar la pared celular, pero las dos proteínas juntas pueden crear un "translocón", el equivalente celular de un túnel a través de la membrana celular. Una vez que se establece el túnel, la célula patógena puede inyectar otras proteínas que realizan el trabajo de infectar al huésped.
Todo este proceso se denomina sistema de secreción tipo 3 y nada de esto funciona sin PopB y PopD. "Si no intentamos matar el patógeno", dice Heuck, "entonces no hay posibilidad de que desarrolle resistencia. Simplemente estamos saboteando su máquina. El patógeno todavía está vivo; simplemente es ineficaz y el huésped tiene tiempo". utilizar sus defensas naturales para deshacerse del patógeno."
La pregunta entonces es ¿cómo encontrar la molécula que pueda bloquear el ensamblaje del translocón?
A veces, las soluciones llegan a los científicos en esos "momentos de iluminación" cuando de repente todo tiene sentido. En este caso, fue más bien un momento relámpago.
Heuck y sus colegas se dieron cuenta de que una clase de enzimas llamadas luciferasas, similares a las que hacen que las luciérnagas brillen por la noche, podrían usarse como rastreadores. Dividieron la enzima en dos mitades. La mitad se destinó a las proteínas PopD/PopB y la otra mitad se diseñó en una célula huésped.
Estas proteínas y huéspedes diseñados pueden inundarse con diferentes compuestos químicos. Si la célula huésped se ilumina repentinamente, eso significa que PopD/PopB rompió con éxito la pared celular, reuniendo las dos mitades de la luciferasa y haciendo que brillaran. ¿Pero si las celdas permanecen oscuras? "Entonces sabemos qué moléculas rompen el translocón", afirma Heuck.
Heuck se apresura a señalar que la investigación de su equipo no sólo tiene aplicaciones obvias en el mundo de los productos farmacéuticos y la salud pública, sino que también mejora nuestra comprensión de cómo exactamente los microbios infectan las células sanas. "Queríamos estudiar cómo funcionaban los patógenos", afirma, "y de repente descubrimos que nuestros hallazgos pueden ayudar a resolver un problema de salud pública".
Esta investigación se publica en la revista ACS Infectious Diseases .
Más información: Hanling Guo et al, ensayo basado en células para determinar el ensamblaje del translocón del sistema de secreción tipo 3 en Pseudomonas aeruginosa utilizando luciferasa dividida, Enfermedades infecciosas del SCA (2023). DOI:10.1021/acsinfecdis.3c00482
Información de la revista: Enfermedades infecciosas SCA
Proporcionado por la Universidad de Massachusetts Amherst