Un equipo de investigadores ha demostrado recientemente la utilidad de emplear un sistema de detección previamente establecido para investigar las interacciones entre moléculas pequeñas y el ARN. Su estudio se publica en Communications Chemistry .
El equipo estuvo dirigido por el profesor Hirohide Saito (Departamento de Fronteras de las Ciencias de la Vida, CiRA), el Dr. Kaoru R. Komatsu (ex estudiante de doctorado en CiRA), el profesor asociado Kazumitsu Onizuka y el profesor Fumi Nagatsugi (Instituto de Investigación Multidisciplinaria). de Materiales Avanzados, Universidad de Tohoku).
Desde las recientes vacunas de ARNm del SARS-CoV-2 para combatir la pandemia de COVID-19 hasta el risdiplam, un modificador de empalme de ARN aprobado por la Administración de Alimentos y Medicamentos de EE. UU. para la atrofia muscular espinal, la palabra "ARN" ha entrado en el lenguaje común ya que representa ambos. nuevas clases de agentes terapéuticos y objetivos farmacológicos.
Sin embargo, nuestra comprensión de cómo diversas secuencias de ARN y, a su vez, estructuras dictan las interacciones con moléculas pequeñas, como compuestos farmacológicos, o biomoléculas grandes, como proteínas, sigue siendo incompleta.
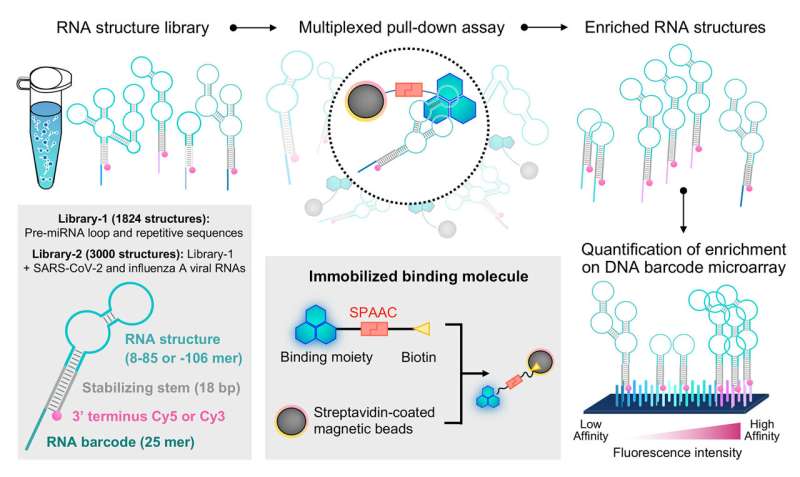
En un estudio anterior, el profesor Saito y su equipo de investigación diseñaron un sistema llamado perfilado de elementos de ARN plegado con biblioteca de estructuras, o BOSQUE, para examinar los detalles moleculares de cómo el ARN interactúa con proteínas de unión a ARN conocidas.
Para este nuevo estudio, a través de una colaboración con investigadores de la Universidad de Tohoku, el equipo de investigación conjunto ilustró cómo se puede utilizar FOREST para analizar interacciones moleculares pequeñas con el ARN.
El equipo de investigación validó primero la aplicabilidad del enfoque FOREST a moléculas pequeñas examinando cómo interactúa una biblioteca de estructuras de ARN con interactores de ARN moleculares pequeños conocidos:G-clamp y derivados de naranja de tiazina (TO).
Los investigadores aislaron estructuras de ARN unidas a G-clamp de una biblioteca que comprende más de 1.800 secuencias de ARN derivadas de pre-miARN humanos y otras secuencias repetitivas y de control. Debido a que estas estructuras de ARN están vinculadas a un tinte fluorescente y a un código de barras de ARN, podrían decodificarse y cuantificarse fácilmente mediante una micromatriz de ADN con secuencias complementarias a los códigos de barras de ARN para proporcionar un análisis cuantitativo de cómo las estructuras de ARN interactúan con la pequeña molécula específica de interés.
Como se esperaba, identificaron la unión preferencial a secuencias de ARN monocatenario y bicatenario (ssRNA y dsRNA, respectivamente) que contienen guanosina (G) mediante G-clamp. De la biblioteca de estructuras de ARN, el equipo de investigación eligió secuencias que mostraban una unión de afinidad alta, intermedia o baja a G-clamp para su validación mediante un experimento independiente basado en fluorescencia que mide directamente las constantes de disociación aparente de las interacciones individuales.
Sorprendentemente, observaron una buena correlación entre la afinidad de unión relativa estimada mediante el enfoque FOREST y las constantes de disociación aparente determinadas por el ensayo de unión basado en fluorescencia, lo que indica la gran solidez de este método para cuantificar las interacciones entre moléculas pequeñas y ARN.
Además, al mutar una estructura de bucle de ARN específica con múltiples guanosinas en diferentes ubicaciones, descubrieron que G-clamp no interactúa con todas las guanosinas en el bucle por igual, pero que el contexto estructural adicional puede influir en la interacción.
Por el contrario, los derivados de TO se utilizan comúnmente como sondas para ensayos de desplazamiento de indicador fluorescente (FID). A continuación, los investigadores mezclaron TO y TO-3 por separado con una biblioteca de estructuras de ARN ampliada que contenía secuencias adicionales derivadas de los ARN del virus del SARS-CoV-2 y la influenza A para caracterizar mejor los derivados de TO para las mediciones de ARN.
Como se esperaba, mientras que no hubo correlación entre los perfiles de unión de las estructuras de ARN que interactúan con los derivados G-clamp y TO, TO y TO-3 compartieron perfiles de unión similares con algunas distinciones menores.
Más comparaciones entre TO-N3 , TO-N3 -2 y A-3-N3 Reveló que la posición del enlazador tiene una influencia modesta sobre los perfiles de unión de ARN. Además, basándose en estos perfiles de unión, los investigadores discernieron algunas preferencias posicionales de base y bucle que tienen los derivados de TO cuando interactúan con estructuras de ARN.
El equipo de investigación amplió además su análisis de los derivados de TO comparando las afinidades de unión relativas determinadas por FOREST con las constantes de disociación aparente medidas para los indicadores nucleicos fluorescentes disponibles comercialmente, TO-PRO-1 y TO-PRO-3, mediante el ensayo de unión basado en fluorescencia. .
A través de este análisis, revelaron que si bien TO-N3 -2 puede representar con mayor precisión el perfil de unión de TO-PRO1 en comparación con TO-3-N3 , ambos TO-N3 -2 y A-3-N3 simula TO-PRO-3 casi igual de bien, proporcionando así información estructural crucial para mejorar los emparejamientos de ARN objetivo e indicadores fluorescentes para ensayos FID.
Utilizando los perfiles de unión determinados para los derivados de TO, el equipo de investigación seleccionó combinaciones de indicadores fluorescentes (TO-PRO-1 o TO-PRO-3) y secuencias de pre-miARN que previamente se había demostrado que estaban desreguladas en tumores con afinidades de unión intermedias para los ensayos de FID.
Como prueba de concepto, los investigadores examinaron una biblioteca química disponible comercialmente con 118 compuestos para identificar pequeñas moléculas capaces de interactuar con pre-miARN asociados a enfermedades. A través de este esfuerzo, identificaron baicaleína (Bai), miricetina (Myr), cloruro de queleritrina (Che) y AS 602801 (AS) como compuestos candidatos. Mientras que se sabe que Myr y Che unen ADN y ARN, esta fue la primera demostración de AS como interactuante de ácidos nucleicos.
En particular, los investigadores observaron resultados diferentes cuando se utilizó TO-PRO-1 o TO-PRO-3 como indicador fluorescente, lo que sugiere que se deben emplear indicadores distintos para evitar identificaciones falsas positivas y negativas. Un examen más detallado de AS confirmó la unión a varios pre-miARN humanos de interés, pero los investigadores también notaron que el compuesto exhibe fuertes propiedades de iluminación cuando interactúa con el ARN.
El examen estructural del compuesto sugiere que contiene una fracción química probablemente responsable de las propiedades de iluminación, lo que lo convierte en un compuesto de interés para un mayor desarrollo en un nuevo interactor de ARN y una sonda fluorescente.
En este estudio, el esfuerzo conjunto de investigación ilustra una vez más la aplicabilidad de la metodología FOREST, no sólo para inspeccionar las interacciones ARN-proteína sino también para investigar los detalles finos de las interacciones entre el ARN y las moléculas pequeñas.
Dado el enorme potencial del ARN como nuevo enfoque terapéutico en la medicina de próxima generación, la capacidad de caracterizar sistémicamente las interacciones entre moléculas pequeñas y ARN a gran escala tendrá enormes impactos en la investigación básica del ARN y la traducción de ese conocimiento en terapias.
Más información: Ryosuke Nagasawa et al, Análisis a gran escala de interacciones entre moléculas pequeñas y ARN utilizando bibliotecas de estructuras de ARN multiplexadas, Química de las comunicaciones (2024). DOI:10.1038/s42004-024-01181-8
Información de la revista: Química de las Comunicaciones
Proporcionado por la Universidad de Kyoto