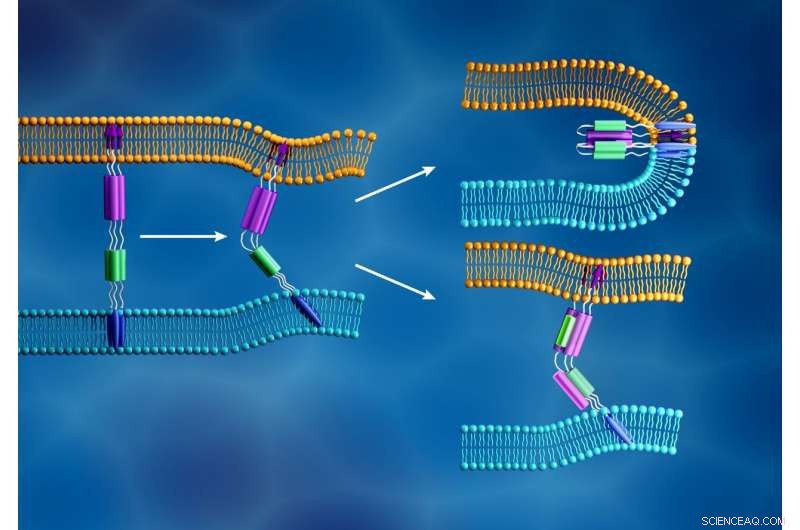
La nueva membrana del coronavirus (azul claro) y la membrana de la célula humana (naranja) se fusionan cuando el péptido de fusión de la subunidad S2 viral (flechas moradas) se inserta en la membrana celular y un componente diferente de la subunidad S2 (morado y verde) se pliega para formar una estructura compacta, como se muestra en la parte superior derecha. A diferencia de, como se ilustra en la parte inferior derecha, Los inhibidores de fusión están diseñados para prevenir la infección viral al interrumpir este proceso. Crédito:ORNL / Jill Hemman
SARS-CoV-2, el coronavirus responsable de la enfermedad COVID-19, está infectando al mundo a un ritmo acelerado. Comprender cómo funciona esta infección a nivel molecular podría ayudar a los expertos a descubrir formas de moderar o detener la propagación.
Un equipo de científicos del Laboratorio Nacional Oak Ridge (ORNL) del Departamento de Energía (DOE) está utilizando la reflectometría de neutrones para hacer precisamente eso.
Los neutrones son capaces de sondear materiales biológicos en condiciones fisiológicas sin dañarlos. Aprovechando estas propiedades, los investigadores pueden medir la dinámica de infección del virus a medida que ocurre.
Su misión es obtener una visión detallada de algunas de las primeras etapas de la infección que ocurren en la membrana celular. Estos hallazgos ayudarán al equipo a probar candidatos a fármacos antivirales que podrían interrumpir este proceso. Los datos obtenidos de estos experimentos también podrían informar otros estudios centrados en el desarrollo de terapias y vacunas.
Los investigadores están centrando su análisis en las proteínas de pico del SARS-CoV-2, proteínas estructurales en forma de púas que cubren la superficie del virus y desencadenan el proceso de infección. La proteína de pico se une a un receptor en la capa externa de la célula huésped y facilita la fusión entre las membranas viral y celular. permitiendo que el virus entre en la célula y libere su material genético. Luego, la maquinaria de producción de proteínas de la célula usa esta información genética para hacer nuevas copias del virus.
Cuando el SARS-CoV-2 secuestra una célula huésped, su proteína de punta se divide en dos subunidades, llamado S1 y S2. Las dos partes son esenciales para la infección. La subunidad S1 contiene un dominio de unión al receptor que reconoce y se adhiere a un receptor celular. Los receptores celulares son proteínas incrustadas dentro de la membrana celular que pueden unirse a moléculas específicas fuera de la célula. Esta conexión puede hacer que los componentes cambien de forma, que a su vez podría inducir cambios en cascada dentro de la célula. Para la proteína de pico SARS-CoV-2, esta conexión activa la subunidad S2, lo que ayuda al virus a fusionar su membrana con la de la célula. Por lo tanto, la función de la proteína de punta es similar a abrir una puerta cerrada, con S1 como la llave que abre la puerta y S2 como la fuerza que empuja la puerta para abrirla.
Aprendiendo de epidemias pasadas
La estructura general de la proteína pico del SARS-CoV-2 es muy similar a la del SARS-CoV, un coronavirus anterior que causó el síndrome respiratorio agudo severo (SARS), y esta semejanza ayudó al equipo a desarrollar su estrategia de investigación.
La subunidad S1 es el foco de muchos estudios de desarrollo de fármacos, ya que se ha demostrado que esta parte de la proteína de pico provoca una respuesta inmunitaria en el cuerpo humano. Sin embargo, Estudios anteriores de SARS-CoV encontraron que la subunidad S1 experimenta altas tasas de mutación, permitiendo que el virus eluda tratamientos basados en anticuerpos mientras mantiene su capacidad de infectar células. "Esta es la lección que aprendimos de la epidemia de SARS original, "dijo Minh Phan, investigador asociado postdoctoral en ORNL e investigador principal de este proyecto.
Phan y sus colegas están estudiando la subunidad S2 porque este componente de la proteína de pico no muta tan rápidamente. Los tratamientos que tienen éxito en la inhibición de la función S2 pueden seguir siendo eficaces durante más tiempo.
Una vista a nanoescala del coronavirus
Para comprender mejor la dinámica de las subunidades virales S2 y las membranas de la célula huésped, los investigadores están empleando el reflectómetro de líquidos (LIQREF) en la fuente de neutrones de espalación (SNS) de ORNL. Midiendo cómo los neutrones se reflejan en diferentes ángulos cuando atraviesan diferentes tipos de materia, el instrumento puede ayudar a arrojar luz sobre la estructura de los materiales biológicos a escala molecular.
El equipo sintetizó primero una membrana lipídica que imita la membrana externa de las células que recubren las superficies dentro de los pulmones humanos. donde puede tener lugar esta infección viral. Identificaron cómo se organizaban los lípidos dentro de la membrana y cómo cambia esta disposición cuando las membranas están expuestas a diferentes condiciones. como la temperatura, presión, y acidez.
En el instrumento LIQREF, los investigadores extendieron la membrana lipídica sobre una fina capa de agua en un aparato llamado abrevadero Langmuir. Luego introducen la subunidad S2 en estas membranas para observar en detalle cómo las membranas S2 y lipídica cambian de forma cuando interactúan.
Los neutrones también son ideales para este estudio porque son sensibles al elemento hidrógeno, común a todas las moléculas biológicas, y sus isótopos. Reemplazando algunos átomos de hidrógeno con átomos de deuterio, los científicos pueden crear contraste en sus muestras y concentrarse selectivamente en diferentes características estructurales. Esta técnica es útil para estudiar muestras que involucran múltiples componentes con densidades similares, como membranas lipídicas.
"Generalmente, estas membranas no son membranas de un solo lípido, "dijo John Ankner, an instrument scientist involved with this study. "They consist of lipids of a certain structure, lipids of another structure, colesterol, proteínas, and things that come in contact with them."
To capture this complexity, the research team is investigating multiple versions of the membrane, changing the contrast of the sample with deuterium each time.

Researchers at ORNL are using neutron scattering at the Spallation Neutron Source to better understand how spike proteins help the COVID-19 virus infect human cells and what drugs could be effective in stopping them. This research team includes John Ankner (left) and Minh Phan (right). Crédito:ORNL / Genevieve Martin
"By taking multiple measurements and assembling all of this information together, you can create a single picture of how these different components go together, " said Ankner.
The information derived from these experiments will then help steer the team's efforts in selecting and testing drug candidates that could block this interaction, such as fusion inhibitors that successfully blocked original SARS-CoV infections. If these inhibitors can stop the new coronavirus from invading healthy cells, existing drugs could potentially be repurposed to treat COVID-19 patients. The results may also help guide the design of new fusion inhibitors.
Capturing infection
While other studies have used protein crystallography to better understand the atomic structure of the coronavirus S2 subunit alone, this project is analyzing how S2 changes shape when interacting with a lipid membrane. A shape change could be important for inducing actions within a cell after the spike S1 subunit binds to the cell receptor. Phan also notes that the LIQREF instrument allows the team to measure these dynamics under physiological conditions, whereas protein crystallography only allows researchers to capture what the S2 subunit looks like in a crystallized form.
"At ORNL, we have the right tools to study the dynamics of the interaction under physiological conditions. This allows us to better understand how the S2 subunit moves and changes shape naturally in a wet environment, " said Phan. "Such information could complement what experts already know about the protein from crystallography. If we can help verify what this mechanism looks like, then we may have a clearer understanding to guide the development of drugs that block the fusion process.
Collaboration is key
Por supuesto, learning more about the S2 subunit and its certain behaviors depends on the ability to grow quality samples, which involves synthesizing S2 subunit proteins, purifying them, and preparing them for experimentation.
Phan and Ankner note that this part of their research has been made possible only through collaboration with labs across ORNL and at outside institutions.
The S2 subunit protein was synthesized in mammalian cell cultures by Steve Foster, a biomedical researcher at the University of Tennessee Medical Center in Knoxville, Tennesse. Through this method, he can develop S2 proteins for research that retain several aspects of its natural structure and function.
"In our lab we routinely use mammalian cell cultures for protein production, so we hope we've produced an S2 protein best suited for this research analysis. Our proximity to ORNL also works well in that the sample doesn't have to travel far, meaning less risk of damaging the protein or distorting its original structure, which is critical for this work, " said Foster.
Following its synthesis, the sample was purified by Jessy Labbé and Michael Melesse Vergara from ORNL's Biosciences Division. Scientists from the ORNL Neutron Sciences Directorate then performed a series of tests to confirm the structure of the sample protein and check its purity. This effort was implemented by Yichong Fan and Wellington Leite from the Bio-Labs team, and Jacob Kinnun and Mary Odom from the SNS team.
"We put an enormous effort into making sure the protein has the right properties going into the experiment. If it does not, we could get spurious results and misinterpret what we're doing, " said Hugh O'Neill, director of ORNL's Center for Structural and Molecular Biology and lead researcher for the Bio-Labs team.
"This virus is extremely delicate in its components, and it's a big challenge to get these materials to the neutron instrument, " said Ankner. "That's why involving various ORNL labs and the University of Tennessee is so crucial. Each step that eventually gets the sample onto our instrument requires the expertise of lots of people."
This project also relied on efforts from the LIQREF instrument staff, who were instrumental in developing the systems, protocols, and modeling frameworks necessary to run the experiments and interpret the data.
"Experts across the division, across ORNL, and from partner institutes have come together for this project, " said Phan. "We couldn't have done this without their support, and it's greater motivation to fulfill our mission."