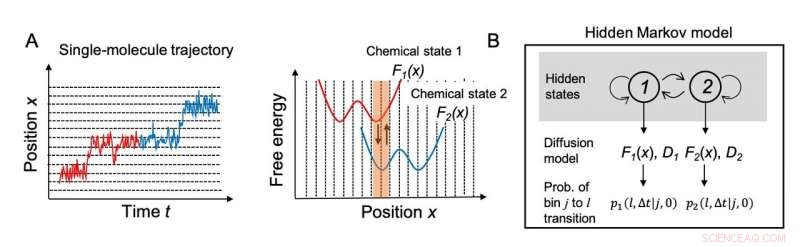
Fig. 1. (A) Trayectoria del movimiento de una sola molécula y los correspondientes perfiles de energía libre dependientes del estado químico. (B) Esquema de un modelo de Markov oculto en el que los estados químicos se consideran estados "ocultos". Crédito:NINS / IMS
Se ha desarrollado un nuevo método de modelado matemático para estimar modelos de funcionamiento de motores biomoleculares a partir de datos de movimiento de imágenes de una sola molécula con el marco de inferencia bayesiano. El mecanismo de funcionamiento de un motor molecular lineal "quitinasa, "que se mueve unidireccionalmente en una cadena de quitina con la degradación de la cadena que pasa, fue dilucidado mediante el modelado matemático de datos de imágenes experimentales con el método.
Los motores biomoleculares en las células generan movimiento unidireccional, consumir energía química obtenida por, por ejemplo, hidrólisis de ATP. Aclaración del principio de funcionamiento de tales motores moleculares, que son nanomáquinas fabricadas por la naturaleza compuestas de proteínas, ha atraído mucha atención. Imágenes de una sola molécula, que puede capturar directamente el movimiento de motores moleculares, es una técnica prometedora para comprender el principio de funcionamiento de los motores moleculares. Sin embargo, todavía no está claro cómo el consumo de energía química, es decir., cambio en los estados químicos de tales proteínas motoras, da lugar al movimiento unidireccional de todos los motores. Investigadores del Instituto de Ciencia Molecular y de la Universidad de Shizuoka han descubierto el cambio de las formas de los perfiles de energía libre a lo largo del movimiento de un motor molecular desencadenado por cambios de estado químico del motor.
Los investigadores primero intentaron establecer un modelo computacional para describir el movimiento de los motores moleculares. El movimiento de un motor puede considerarse como un movimiento de difusión sobre perfiles de energía libre que cambian según los estados químicos de las moléculas que componen el motor. Más específicamente, como se muestra en la Fig. 1A, el motor se mueve primero sobre la superficie de energía libre del estado químico 1 (rojo) de las moléculas del motor, y luego se mueve sobre la superficie de energía libre del estado químico 2 (azul). Sin embargo, este cambio de estado químico no se suele observar en la formación de imágenes de una sola molécula. Los investigadores trataron la transición entre los estados químicos utilizando un modelo de Markov oculto en el que los estados químicos se consideran estados "ocultos" (Fig. 1B).
Usando este modelo oculto de Markov, es posible calcular la "probabilidad, "que evalúa la probabilidad para mostrar qué tan bien el modelo explica la trayectoria del movimiento real de una sola molécula. También es posible incorporar el conocimiento de los perfiles de energía libre como probabilidades previas. Los investigadores han desarrollado un método para estimar el estado químico perfiles de energía libre dependientes, coeficientes de difusión en cada perfil, y las constantes de velocidad de las transiciones entre estos estados dentro del marco de inferencia bayesiano por muestreo de Monte Carlo utilizando probabilidades posteriores expresadas como un producto de la verosimilitud y las probabilidades previas.
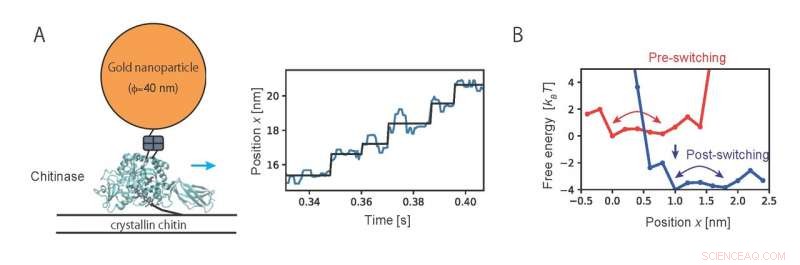
Fig. 2. (A) Movimiento unidireccional observado mediante imágenes de una sola molécula de quitinasa. (B) Perfiles de energía libre dependientes del estado químico estimados a partir de los datos de imágenes. Crédito:NINS / IMS
Luego, Se aplicó el método desarrollado en el presente estudio para analizar el movimiento de la quitinasa, un motor molecular lineal, observado por imágenes de una sola molécula. El análisis de los datos de la trayectoria del movimiento unidireccional de la quitinasa con la degradación de una cadena de quitina reveló los perfiles característicos de energía libre que gobiernan el movimiento (Fig. 2). Los resultados del análisis mostraron que una quitinasa se coloca en un carril de la cadena de quitina sobre una barrera de energía libre relativamente baja por el movimiento browniano. Luego, el movimiento unidireccional se logra cambiando los estados químicos a través de la reacción de hidrólisis de la cadena de quitina y la disociación de los productos de reacción. El presente estudio proporciona una base física para el mecanismo de trinquete browniano de "puente quemado" que los investigadores han informado anteriormente.
"Aplicaremos nuestro método desarrollado en este estudio a varios motores moleculares y esperamos aclarar las similitudes y diferencias en los mecanismos de los motores moleculares. Creemos que nuestro método obtendrá nuevos hallazgos en el futuro y nos dará una pista para los principios generales de funcionamiento de los motores moleculares. Los estudios que utilicen nuestro método allanarán el camino para el diseño de nuevos motores moleculares artificiales, "dijo Okazaki.