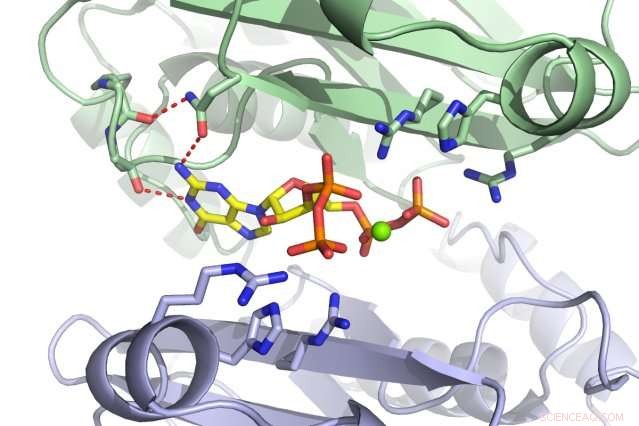
Un equipo del MIT ha descubierto que la molécula pequeña ppGpp (roja) se une a la enzima PurF (subunidades que se muestran en verde y morado). Crédito:Instituto de Tecnología de Massachusetts
En 1969, El científico Michael Cashel estaba analizando los compuestos producidos por bacterias hambrientas cuando notó que aparecían dos manchas en su cromatograma como por arte de magia. Hoy dia, conocemos uno de estos "puntos mágicos, "como los llaman los investigadores, como tetrafosfato de guanosina, o ppGpp para abreviar. También entendemos que es una molécula de señalización presente en prácticamente todas las bacterias, ayudando a ajustar el crecimiento y el tamaño de las células en función de la disponibilidad de nutrientes.
Y todavía, a pesar de décadas de estudio, Precisamente la forma en que ppGpp regula el crecimiento bacteriano sigue siendo bastante misteriosa. Profundizar más requiere una lista más completa de las moléculas a las que se une ppGpp para ejercer sus efectos.
Ahora, colaboradores de los departamentos de Biología y Química del MIT han desarrollado un método para hacer precisamente eso, y utilizó su nuevo enfoque para identificar más de 50 objetivos de ppGpp en Escherichia coli, aproximadamente la mitad de los cuales no se habían identificado previamente. Muchos de estos objetivos son enzimas necesarias para producir nucleótidos, los componentes básicos del ADN y el ARN. Durante los momentos en que las bacterias no tienen suficientes nutrientes para crecer y dividirse normalmente, los investigadores proponen que ppGpp evita que estas enzimas creen nuevos nucleótidos desde cero, ayudando a las células a entrar en un estado inactivo.
"Con moléculas pequeñas o metabolitos como ppGpp, Históricamente ha sido difícil determinar a qué proteínas se unen, "dice Michael Laub, un profesor de biología, un investigador del Instituto Médico Howard Hughes, y el autor principal del estudio. "Este ha sido un problema insoluble que ha frenado el campo durante algún tiempo, pero nuestro nuevo enfoque le permite concretar los posibles objetivos en cuestión de semanas ".
Postdoc Boyuan Wang es el primer autor del estudio, que aparece en Biología química de la naturaleza el 17 de diciembre.
El lugar magico
Desde que se descubrió ppGpp hace casi 50 años, se ha demostrado que suprime la replicación del ADN, transcripción, traducción, y varias vías metabólicas. Frena el crecimiento celular y permite que las bacterias persistan ante la inanición. estrés, y antibióticos. Su influencia sobre numerosos procesos regulatorios sigue siendo un misterio, sin embargo, después de todo, no solo modula una sola vía, sino que coordina múltiples operaciones simultáneamente para orquestar un cierre masivo de la celda.
Para discernir qué proteínas se une ppGpp para efectuar un cambio tan generalizado, los investigadores construyeron lo que ellos llaman "compuestos de captura" que contienen ppGpp, permitiéndoles pescar sus objetivos a partir de extractos bacterianos. Estos compuestos incluían un reticulante fotorreactivo que se adhería firmemente a las proteínas de interés en presencia de luz, y un mango de biotina que ayudó a los científicos a extraer las proteínas para identificarlas. Más importante, se unieron a ppGpp de tal manera que no interferirían con su capacidad para unirse a sus objetivos. Este método es más eficiente y preciso en comparación con los medios más tradicionales para distinguir los objetivos de ppGpp, que son mucho más arduos y carecen de sensibilidad.
"Nuestro enfoque resuelve estos problemas porque ya no es necesario realizar protocolos tan laboriosos para identificar objetivos de ppGpp, y funciona incluso en bacterias más allá de E. coli, "dice Wang." Aunque ppGpp es común entre muchas especies bacterianas, parece ejercer sus efectos a través de diferentes mecanismos, lo que complica las cosas. Nuestros compuestos de captura proporcionan una forma de desentrañar esta diversidad, y en poco tiempo ".
Poniendo freno a la producción de nucleótidos
Aunque los objetivos de 56 ppGpp que Wang identificó en su pantalla controlan una miríada de procesos celulares, se centró en la enzima PurF, que inicia la biosíntesis de nucleótidos de purina que contienen bases de adenina y guanina, también conocido como A y G.
Cuando las bacterias están estresadas o muertas de hambre, entran en un estado latente para sobrevivir. Pero no basta con limitar la traducción y la transcripción; Los nucleótidos todavía se están generando y se acumularán si no se pone en pausa su síntesis. Las células pueden construir nucleótidos de dos formas:recuperando materiales existentes o comenzando completamente desde cero. PurF inicia el primer paso en el último proceso que conduce a los nucleótidos A y G. Sin embargo, cuando ppGpp se une a PurF, hace que la enzima cambie de forma, que le impide hacer su trabajo, reduciendo así la producción de nucleótidos en la célula.
"Esta es la primera vez que una enzima involucrada en esa vía o función específica se identifica como un objetivo de ppGpp, ", Dice Wang." Si limita el consumo de nucleótidos pero no su producción, el grupo de nucleótidos va a explotar, lo cual no es bueno para el celular. Así que hemos demostrado que ppGpp también aborda este problema ".
Además de PurF y otras enzimas necesarias para la producción de nucleótidos, los investigadores notaron que ppGpp también se une a muchas enzimas GTPasa involucradas en la traducción. Esto podría indicar un mecanismo a prueba de fallos que ralentiza la traducción al activar varios enzimas similares de una manera casi redundante frente al hambre.
Mientras Wang continúa perfeccionando su método, su objetivo es aumentar su especificidad y garantizar que sus compuestos de captura se unan exactamente a las mismas proteínas que lo harían dentro de una célula viva. También espera detectar proteínas de unión a ppGpp en otras bacterias, incluidos los patógenos que dependen de ppGpp para sobrevivir dentro de sus huéspedes y propagar enfermedades como la tuberculosis.
"Este es un enfoque químico emocionante para comprender mejor la función de una molécula de señalización conservada estudiada durante mucho tiempo en las bacterias, "dice Jue Wang, profesor de bacteriología en la Universidad de Wisconsin en Madison, que no participó en el estudio. "Sus hallazgos y técnicas son muy relevantes para muchas otras bacterias, y mejorará enormemente el conocimiento de cómo las bacterias utilizan esta molécula de señalización crítica para mediar en todo, desde la supervivencia en el intestino humano hasta la causa de la enfermedad ".
Agrega Laub:"Todavía hoy estamos descubriendo nuevas moléculas de señalización basadas en nucleótidos en bacterias, y cada uno de ellos podría eventualmente derivatizarse de una manera similar para identificar a sus socios vinculantes ".
Esta historia se vuelve a publicar por cortesía de MIT News (web.mit.edu/newsoffice/), un sitio popular que cubre noticias sobre la investigación del MIT, innovación y docencia.