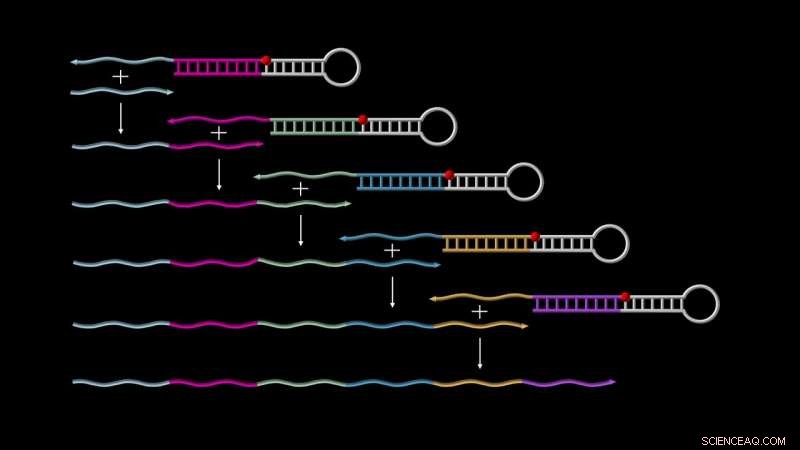
Las cascadas de reacción de intercambio de cebadores (PER) permiten el crecimiento autónomo de ADN monocatenarios. En la parte superior, una 'horquilla PER catalítica' une una primera 'imprimación' (mostrada como una hebra gris corta), desencadena su alargamiento con una secuencia codificada por la propia horquilla, y lo suelta para iniciar otro ciclo con la imprimación ya extendida, Etcétera, hasta que se genere una transcripción larga. Crédito:Instituto Wyss de la Universidad de Harvard
Los biólogos y nanobiólogos sintéticos están reorientando el ADN, el material hereditario presente en casi todas las células del cuerpo, como material de autoensamblaje inteligente y estable para construir nanofábricas, nanoestructuras liberadoras de fármacos y dispositivos moleculares que pueden detectar su entorno y responder de diferentes formas al, por ejemplo, detectar inflamación en el cuerpo o toxinas en el medio ambiente. Estas aplicaciones a nanoescala a menudo implican la síntesis de grandes secuencias que comprenden miles de los componentes básicos de los que está hecho el ADN, conocido como el A, T, Bases de nucleótidos C y G, que se puede doblar y estructurar aún más debido a las habilidades específicas de emparejamiento de bases entre As y Ts, y Cs y Gs, respectivamente.
Sin embargo, hasta aquí, Los investigadores no tienen herramientas a su disposición que permitan que las secuencias monocatenarias más grandes crezcan de manera autónoma y luego se unan de un extremo a otro siguiendo un plan de diseño molecular. una capacidad que podría generar estructuras y dispositivos con diversas capacidades.
Publicado hoy en Química de la naturaleza , La investigación de Peng Yin en el Instituto Wyss de Ingeniería de Inspiración Biológica de Harvard proporciona una solución ampliamente aplicable a este problema. Yin y su equipo han desarrollado un método que permite que las secuencias prediseñadas de ADN crezcan y se concatenen de forma autónoma a lo largo de rutas de ensamblaje específicas. proporcionando así la base para una nueva generación de dispositivos moleculares programables. Poniendo a prueba su nuevo concepto de las llamadas cascadas de 'Reacción de intercambio de cebadores' (PER), diseñaron con éxito un primer conjunto de dispositivos con diversas funciones, como la autoconstrucción de ADN-origami y nanoestructuras de ADN que detectan, amplificar, registrar o evaluar lógicamente las señales ambientales.
Los métodos pasados produjeron copias idénticas de una secuencia fija más pequeña, pero son incapaces de agregar diferentes secuencias sintetizadas entre sí en patrones definidos para generar ensamblajes más grandes de forma autónoma sin la intervención mediada por el usuario. "Las características autónomas y programables que ofrecen las cascadas PER podrían generar una generación completamente nueva de aplicaciones y dispositivos moleculares programables y cerrar brechas en los esfuerzos de diseño, para los que ya existen muchas partes móviles, ", dijo Peng Yin, miembro de la facultad principal del Instituto Wyss, Doctor., quien dirigió el estudio y también es profesor de Biología de Sistemas en la Escuela de Medicina de Harvard (HMS). "Proporcionamos datos de prueba de concepto para PER en una amplia gama de aplicaciones de biología sintética de vanguardia que destacan claramente el amplio potencial de la tecnología".
El equipo del Instituto Wyss utilizó el nuevo concepto para diseñar una serie de transcripciones de ADN PER para aplicaciones muy diversas. incluida la síntesis autónoma de grandes nanoestructuras de ADN conocidas como ADN-origamis, y enfoques de biología sintética, en el que la síntesis de una transcripción de ADN depende de un desencadenante, como un pequeño microARN asociado al cáncer. Su enfoque PER puede incluso generar transcripciones de ADN resultantes de una combinación evaluada lógicamente de diferentes desencadenantes, similar a los dispositivos de ribocomputación de ARN que el equipo de Yin publicó a principios de este año. Curiosamente, Las transcripciones de ADN PER pueden convertirse en catalíticas por sí mismas, ser capaz de cortar un ARN objetivo arbitrario, se convierten en sondas marcadas con fluorescencia que amplifican la presencia de un estímulo molecular particular, o "registradores moleculares" que indican fielmente el orden en el que aparecen determinadas señales moleculares en sus entornos.
Para iniciar la cascada PER, se necesitan dos componentes básicos. Uno se llama "mediador de horquilla de ADN catalítico", que es una molécula de ADN de una sola hebra que se empareja parcialmente consigo misma para formar una estructura de horquilla con una hebra única corta que sobresale. Este voladizo está diseñado para capturar el segundo componente de las cascadas PER, la "cartilla, "que contiene una región que es complementaria al voladizo. A través de una serie de reacciones de alargamiento y desplazamiento, el cebador se extiende con una secuencia proporcionada por el mediador de horquilla catalítico y luego se expulsa. Esto libera el mediador de horquilla catalítico para que pase en cascada la siguiente ronda del proceso, ya sea capturando una nueva imprimación inicial o la imprimación ya alargada, y así sucesivamente.
Estas complejas vías de síntesis proceden de forma autónoma, comparable a un robot molecular que realiza una tarea determinada, y a una sola temperatura, lo que hace que la tecnología sea muy robusta. "El enfoque nos da una tremenda libertad creativa:no solo podemos sintetizar el mismo fragmento de ADN una y otra vez como nuevas adiciones de una secuencia en crecimiento, pero también podemos variar los tipos de secuencias de ADN que se agregarán simplemente cambiando la composición de los ADN en horquilla catalíticos y los cebadores en la mezcla mientras el ensamblaje está en curso. Esto nos permite hacer que la síntesis se ramifique en diferentes direcciones y modelar intrincadamente la composición de la transcripción final del ADN, "dijo la primera autora del estudio, Jocelyn Kishi, quien, como becario de investigación de posgrado de la National Science Foundation (NSF) en HMS, trabaja en el equipo del Instituto Wyss de Yin. "Ahora estamos trabajando para implementar cascadas PER para una variedad de aplicaciones, incluyendo registradores moleculares, diagnósticos sofisticados, y formación de imágenes de tejidos. También esperamos que algún día estos sistemas se puedan utilizar en células vivas como dispositivos que puedan registrar eventos o reprogramar el comportamiento celular de formas específicas. "dijo Kishi.
"Este nuevo avance que muestra cómo las moléculas de ADN pueden programarse para autoensamblarse en estructuras 3D específicas y llevar a cabo funciones y tareas predefinidas representa un gran paso adelante en el campo de la robótica molecular, y ofrece un vistazo al futuro de los dispositivos para aplicaciones médicas y no médicas, "dijo el director fundador del Instituto Wyss, Donald Ingber, MARYLAND., Doctor., quien también es el Profesor Judah Folkman de Biología Vascular en HMS y el Programa de Biología Vascular en el Boston Children's Hospital, así como profesor de Bioingeniería en la Escuela de Ingeniería y Ciencias Aplicadas John A. Paulson de Harvard (SEAS).