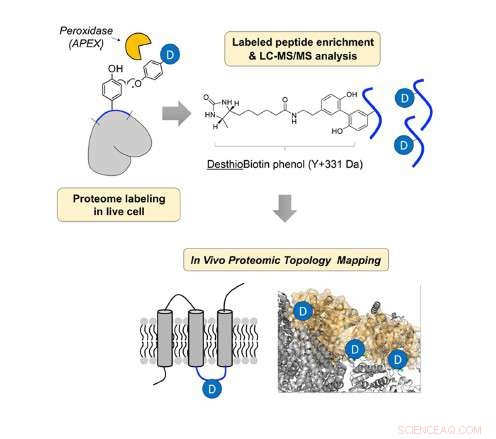
Identificación estructural in vivo de proteína mitocondrial, utilizando peroxidasa y desthiobiotin-fenol. Crédito:UNIST
Las proteínas de membrana constituyen aproximadamente una cuarta parte de todos los productos genéticos y son el objetivo de más del 50 por ciento de todos los fármacos farmacéuticos modernos. El proteoma de la membrana mitocondrial interna (IMM) juega un papel central en el mantenimiento de la fisiología mitocondrial y el metabolismo celular. A pesar de su importancia, no ha habido ningún método para revelar la topología de las proteínas de la membrana mitocondrial en células vivas, hasta ahora.
Un estudio reciente, afiliado a UNIST ha desarrollado una nueva técnica para comprender las arquitecturas correctas de las proteínas IMM, utilizando herramientas químicas especiales. Al resolver la etapa más difícil del desarrollo de nuevos fármacos, su trabajo ayudará a acelerar el desarrollo de nuevas terapias y curas.
Esta investigación ha sido dirigida por el equipo del profesor Hyun-Woo Rhee de Química en UNIST en colaboración con el profesor Jong-Seo Kim en el Centro del Centro de Investigación de ARN, dentro del Instituto de Ciencias Básicas (IBS) de la Universidad Nacional de Seúl y el profesor Jeong-Kon Seo de las Instalaciones Centrales de Investigación de UNIST (UCRF). Los resultados del estudio han aparecido en la edición del 15 de marzo de la Revista de la Sociedad Química Estadounidense ( JACS ).
IMM es uno de los sitios más activos para el metabolismo celular y está profundamente relacionado con diversas enfermedades metabólicas humanas, como el cáncer y las enfermedades neurodegenerativas. Por lo tanto, Es crucial comprender la arquitectura correcta del proteoma IMM en células vivas para el desarrollo exitoso y eficiente de terapias dirigidas a las mitocondrias.
En el estudio, El profesor Lee y su equipo de investigación revelaron la dirección topológica in vivo de 135 proteínas IMM, utilizando una sonda de radicales desthiobiotin-fenoxyl generada in situ con peroxidasa genéticamente dirigida (APEX).
"La determinación de la estructura de la proteína de la membrana es una de las tareas más desafiantes en el análisis de la estructura de la proteína, ", dice el profesor Lee." Nuestra identificación de información estructural en el proteoma de la membrana interna mitocondrial puede proporcionar información valiosa para la arquitectura y el conectoma del proteoma IMM en células vivas ".
El equipo de investigación diseñó una nueva sonda química, desthiobiotin-fenol y lo aplicó a las proteínas IMM en células vivas. Luego, identificaron la estructura de las proteínas de membrana mediante espectrometría de masas (MS).
La peroxidasa puede reaccionar con el peróxido de hidrógeno para producir el radical fenoxilo. Luego, el radical fenoxilo puede reaccionar con el residuo de tirosina en la proteína proximal formando un enlace covalente. En el estudio, el equipo de investigación obtuvo la información de topología analizando el sitio de tirosina marcado de la proteína de membrana.
La mayoría de los análisis de secuencias de proteínas actuales utilizan espectrometría de masas (MS), que digiere la muestra de proteína en péptidos usando una enzima apropiada. Análisis previos, que utilizó ascorbato peroxidasa genéticamente dirigida (APEX), no pudo resolver la identificación estructural porque estos análisis se basaron en la detección de péptidos sin marcar. Sin embargo, solo el péptido marcado puede proporcionar información estructural útil, según el equipo de investigación.
A diferencia de las biomoléculas que están marcadas con biotina-fenol, Las proteínas y otros objetivos que están marcados con desthiobiotin-fenol se pueden eluir sin dureza, condiciones desnaturalizantes. Es más, a medida que aumenta el número de muestras de estructura de proteínas de membrana disponibles obtenidas a través de MS, también aumenta la eficacia de la identificación estructural de las proteínas de membrana.
Debido a la corta vida útil de los radicales fenoxilo generados in situ por APEX dirigido submitocondrial y la impermeabilidad del IMM a moléculas pequeñas, los residuos de tirosina expuestos al disolvente de los lados de la matriz y del espacio intermembrana (IMS) de las proteínas IMM se marcaron exclusivamente con la sonda de radicales en células vivas mediante Matrix-APEX e IMS-APEX, respectivamente e identificados por espectrometría de masas.
A través de este análisis, el equipo de investigación confirmó 58 topologías de proteínas IMM y determinó la dirección topológica de 77 proteínas IMM cuya topología en el IMM no se ha caracterizado completamente.