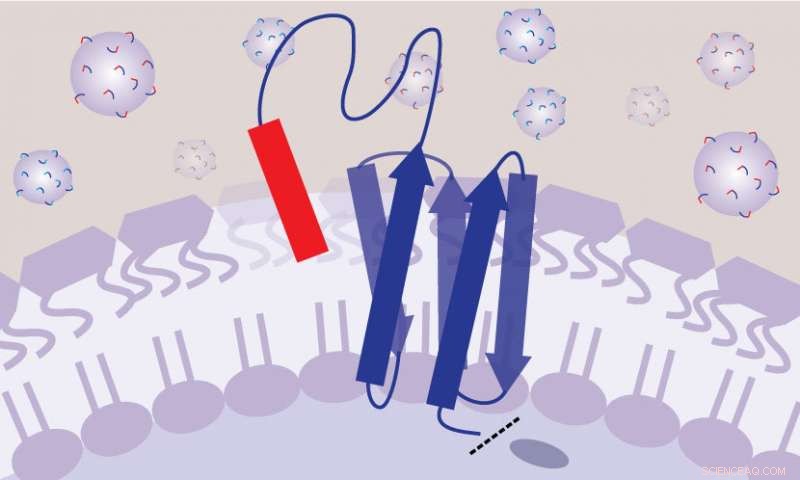
En el método SLAY, cada bacteria está diseñada genéticamente para producir una molécula en la superficie de su célula que es en parte péptido y en parte atadura, como una pelota de juego en el patio de recreo. Esta disposición permite que los péptidos imiten los fármacos que flotan libremente en el cuerpo humano. Crédito:Ashley Tucker
Los biólogos de la Universidad de Texas en Austin han desarrollado un método para detectar rápidamente cientos de miles de medicamentos potenciales para combatir infecciones. una innovación prometedora para combatir el creciente flagelo de las bacterias resistentes a los antibióticos. El método implica manipular bacterias para producir y probar moléculas que son potencialmente tóxicas para ellas mismas.
Una descripción del método aparece en la edición impresa del 25 de enero de la revista. Celda .
No se ha descubierto una nueva clase de antibiótico en 40 años; ya se han encontrado muchos de los más accesibles en la naturaleza. y el proceso para crear y probar nuevos desde cero es lento y laborioso, pero la medicina moderna los necesita con urgencia. Según la Organización Mundial de la Salud, los antibióticos han añadido unos 20 años a la esperanza de vida media de los seres humanos. Pero sus beneficios protectores se están desvaneciendo a medida que las bacterias desarrollan resistencia a los antibióticos.
En su prueba de concepto, el equipo de UT Austin, dirigido por Bryan Davies, proyectado alrededor de 800, 000 moléculas llamadas péptidos para ver si tenían efectos antimicrobianos, lo que significa que mataron bacterias dañinas. De aquellos, varios miles de bacterias E. coli muertas, convirtiéndolos en posibles pistas para los antibióticos. Algunos antibióticos actualmente en uso son péptidos. Será necesaria una investigación de seguimiento para determinar cuál, Si alguna, de los miles de nuevos clientes potenciales son realmente efectivos y seguros en humanos, pero los investigadores demostraron que al menos una de esas moléculas, apodado P7, también mata otras formas de bacterias patógenas y es seguro en ratones.
Con este método, llamado SLAY (pantalla antimicrobiana localizada en superficie), una persona puede analizar cientos de miles de péptidos similares de forma más rápida y rentable que los métodos existentes. A Davies le gustaría que el método se convierta en una herramienta estándar en la búsqueda mundial de nuevos antibióticos.
"Entonces, ¿qué pasa si tenemos mil grupos que usan este sistema para seguir sus propios intereses y sus propios péptidos?" dijo Davies, profesor asistente de biociencias moleculares. "Una vez que habilita una comunidad de ese tamaño, entonces creo que tienes más posibilidades de encontrar un nuevo antibiótico que funcione ".
Un avance clave en este trabajo fue descubrir cómo hacer que las bacterias produzcan moléculas que podrían ser tóxicas para ellas mismas y controlar cómo esas moléculas interactúan con las bacterias anfitrionas.
"Pensamos, ¿No sería genial si una bacteria pudiera sintetizar el compuesto por nosotros? porque las bacterias son baratas y fáciles de cultivar, y luego probar el compuesto en sí mismo e informarnos y decirnos, ¿Era un antimicrobiano o no? ", dijo Davies.
Su solución fue diseñar genéticamente las bacterias para producir una molécula en la superficie celular que es en parte péptido y en parte atadura, como una pelota de juego y su atadura, con un extremo fijado a la membrana celular y el otro libre para flotar. Esto permite que el péptido se mueva y entre en contacto con la superficie de la célula bacteriana. como si flotara libremente como una droga en tu torrente sanguíneo, pero sin interactuar con otras bacterias cercanas.
Asegurándose de que cada versión de la tetherball solo interactúe con las bacterias que la produjeron, los investigadores podrían entonces dar un gran salto en eficiencia. Podrían crear cientos de miles de cepas de bacterias, cada una diseñada genéticamente para producir una versión ligeramente diferente de la bola de tetherball, y poner todas estas cepas en el mismo tubo de ensayo para que crezcan. Al ejecutar cientos de miles de experimentos simultáneamente, su método ahorra una enorme cantidad de espacio, tiempo y costo.
Parte de este proceso se basa en una técnica desarrollada por George Georgiou de UT Austin en la década de 1990 que induce a las bacterias a producir proteínas o péptidos en sus superficies.
Para averiguar qué tetherballs (péptidos) noquean a sus anfitriones, los científicos utilizan la secuenciación de genes para identificar qué versiones están produciendo las bacterias al principio y cuáles se están produciendo al final.
Tras el descubrimiento de que P7 mata a los patógenos, el equipo ahora planea crear miles de variaciones sutiles de esta molécula, llamados derivados, y ejecutarlos por el mismo proceso de selección para buscar una versión aún más eficaz.
La becaria postdoctoral Ashley Tucker dirigió el trabajo experimental para demostrar el uso de la plataforma.
Davies, Tucker y UT Austin han presentado solicitudes de patente para el método SLAY y para las secuencias genéticas específicas de los miles de péptidos antimicrobianos que han descubierto hasta ahora.