En un estudio publicado recientemente en la revista Small , investigadores del Nano Life Science Institute (WPI NanoLSI), Universidad de Kanazawa, Japón, en colaboración con el profesor Sarikaya, Seattle, EE. UU., utilizaron microscopía de fuerza atómica de frecuencia modulada para revelar la arquitectura molecular de péptidos genéticamente diseñados y mutados puntualmente y sus autoorganizaciones cada uno. formando cristales biológicos distintos y de una sola molécula de espesor sobre grafito atómicamente plano y MoS2 superficies, ofreciendo una plataforma potencial para tecnologías híbridas como bioelectrónica, biosensores y matrices de proteínas.
Las proteínas son los componentes moleculares clave en biología, llevan a cabo funciones enzimáticas, transportan iones y electrones y constituyen la infraestructura principal de las arquitecturas celulares, como las bombas de iones. Las funciones de las proteínas dependen de sus secuencias de aminoácidos, que, a su vez, dictan sus estructuras moleculares tridimensionales.
Las secuencias de aminoácidos de las proteínas están codificadas por los genes (código de ADN) de una célula especializada determinada. Por lo tanto, se espera que cualquier defecto en la secuencia de aminoácidos, como una mutación puntual, un cambio en la ubicación de un aminoácido, un aminoácido faltante o la transposición de dos o más aminoácidos, afecte la estructura molecular de la proteína. , lo que se refleja en su función, dando lugar a menudo a enfermedades genéticas.
Al igual que las proteínas, los péptidos también están compuestos de unidades de aminoácidos y cada péptido tiene una secuencia única; sin embargo, son mucho más cortos, oscilan entre 10 y 30 unidades, y están codificados de manera similar por el ADN. Son más fáciles de manipular, diseñar y usar en comparación con las proteínas, que son mucho más grandes (cientos o miles de aminoácidos), difíciles de sintetizar y poco prácticas de usar.
Por lo tanto, es imperativo que si se va a desarrollar la tecnología práctica del futuro basada en las lecciones de la biología a escala molecular, la ciencia fundamental debe establecerse utilizando péptidos, que luego puedan implementarse en aplicaciones del mundo real, como los biosensores. , bioelectrónica y dispositivos lógicos, así como medicamentos, todos ellos basados en lecciones de biología.
Al darse cuenta de esta premisa, el profesor Sarikaya inició un novedoso enfoque científico de convergencia hace dos décadas, que involucra ingeniería genética, biología molecular, informática, aprendizaje automático y ciencia e ingeniería de materiales, en el diseño de péptidos de unión sólida, y demostró su uso en tecnologías híbridas prácticas. /P>
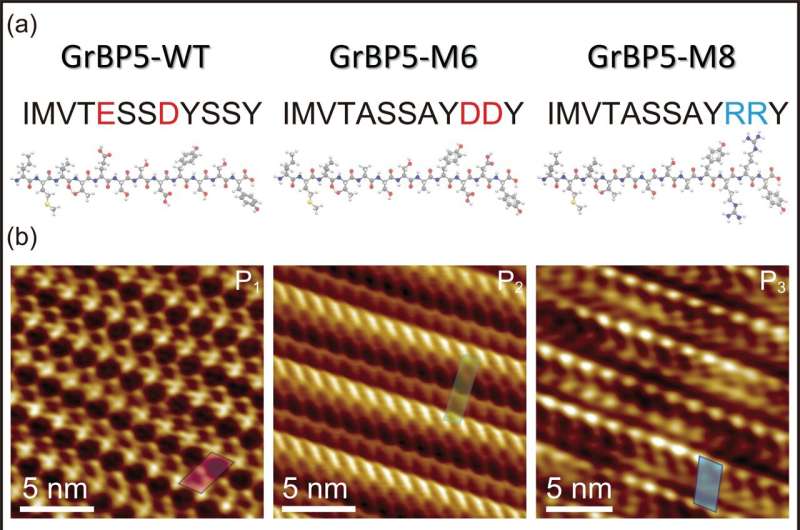
En este nuevo estudio, uno de estos péptidos, diseñado específicamente como un péptido de unión a grafito, se muta en dos péptidos diferentes, M6 y M8, cada uno nominalmente del mismo peso molecular y tamaño, pero que contienen mutaciones puntuales de los aminoácidos cargados, una negativa. y el otro positivo, respectivamente.
Como se predijo, funcionarían de manera diferente, y así fue. Sin embargo, la cuestión que era imposible de predecir a priori era cómo exactamente actuarían de manera diferente entre sí y con el péptido de tipo salvaje (el original), y cuáles serían los detalles de su estructura molecular y su comportamiento cinético en la superficie del sustrato de grafito.
Las respuestas a estas preguntas, cuya falta hasta ahora limitaba la comprensión de la relación entre los mutantes y su comportamiento predecible, requirieron una resolución muy alta y imágenes directas de las moléculas en sus estados nativos, en agua durante un período de segundos a horas. /P>
Aquí es donde entra en escena el laboratorio del profesor Takeshi Fukuma en el WPI Nano Life Science Institute de la Universidad de Kanazawa, donde su equipo ha desarrollado instalaciones de microscopía de fuerza atómica de frecuencia modulada que son capaces de examinar objetos a escala molecular con una resolución de imagen cercana al angstrom y para largos períodos de tiempo en ambientes acuosos:una combinación inusual de capacidades para un sistema experimental, especialmente para la investigación biológica.
Con las altamente competentes habilidades experimentales de los investigadores (con una paciencia inagotable en pruebas de larga duración), liderados por el Dr. Ayhan Yurtsever, el Dr. Kaito Hirata, el Dr. Linhao Sun y el Dr. Takeshi Fukuma, el equipo descubrió el comportamiento del mutantes con un detalle sin precedentes, proporcionando resultados con un significado que abarca varias disciplinas tradicionalmente diversas, desde la ciencia de los materiales hasta la física, la química y la biología.
Primero, a pesar de las mutaciones y de tener individualmente una carga general diferente, cada péptido no solo se unió a la superficie del grafito sino que también se ensambló, formando cristales peptídicos de una sola molécula de espesor. Sin embargo, mientras que el primer mutante, el cargado negativamente, formó una red cristalina oblicua con dos moléculas (envueltas entre sí) en cada red, el segundo mutante, el cargado positivamente, formó una red oblicua diferente que contenía un solo péptido. /P>
Lo realmente valioso aquí es que el comportamiento de los péptidos está simple y directamente relacionado con su arquitectura molecular, modelada mediante dinámica molecular. Por lo tanto, los investigadores pudieron describir matemáticamente la relación entre cada uno de los péptidos (cada uno formando una red molecular oblicua diferente) y la red hexagonal del sustrato de grafito, que se describe exclusivamente como reconocimiento quiral.
En efecto, esto significa que se ha demostrado que cada péptido forma una interfaz híbrida con el sustrato de grafito, que se describe mejor como cristalográficamente coherente sin espacios notables entre ellas, a diferencia de las interacciones ligando-receptor o proteína/ADN en biología.
Ni el reconocimiento molecular de objetos en estado sólido por parte de una molécula "viva" se había observado tan claramente antes, ni sus detalles se habían descrito como quirales, lo que aumenta la importancia de los resultados presentados en esta publicación.
Por un lado, desde el punto de vista de la biología, ahora existen abundantes datos y detalles sobre cómo las mutaciones conducen a cambios conformacionales moleculares y la función resultante de los péptidos, que pueden ampliarse al comportamiento de las proteínas, con importancia en las mutaciones genéticas y en la eficacia. diseño de fármacos.
Y, por otro lado, la hibridación coherente sugiere que los objetos animados e inanimados a nanoescala están presentes de manera estable en la misma interfaz suave, formando un puente molecular exclusivo que podría describirse mejor integrando perfectamente la biología con dispositivos de estado sólido.
Si es así, basándose en este estudio, se espera que los científicos y tecnólogos amplíen enormemente el repertorio de las bases moleculares de los futuros bio/nanodispositivos mediante el uso de la sencilla herramienta de biología molecular de la mutación.
Más información: Ayhan Yurtsever et al, Dinámica del autoensamblaje molecular de péptidos cortos en interfaces líquido-sólido:efecto de las mutaciones puntuales de aminoácidos cargados, pequeñas (2024). DOI:10.1002/smll.202400653
Información de la revista: Pequeño
Proporcionado por la Universidad de Kanazawa