
Esta micrografía de AFM de capas 2-D S ensambladas en mica muestra dos vías diferentes para la cristalización, uno en el que los dominios son 2-3 nanómetros más altos (círculos de puntos blancos) que el otro. Diferencias en los dos perfiles de altura, que se midieron a lo largo de las líneas negras punteadas horizontales, fueron el resultado del atrapamiento cinético. Crédito:Fundición Molecular
(Phys.org) —Las proteínas pueden autoensamblarse en una amplia gama de estructuras altamente ordenadas que presentan una diversa gama de propiedades. A través de la biomimetismo, innovación tecnológica inspirada en la naturaleza, los humanos esperan emular proteínas y producir nuestra propia versión de moléculas autoensambladas. Una clave para lograr esto es comprender cómo el plegamiento de proteínas, un proceso crítico para la forma y función de una proteína, se extiende desde proteínas individuales hasta ensamblajes complejos.
Investigadores del Laboratorio Nacional Lawrence Berkeley (Berkeley Lab) del Departamento de Energía de EE. UU. (DOE) han demostrado ahora que un concepto ampliamente aceptado que describe el plegamiento de una única proteína individual también es aplicable al autoensamblaje de múltiples proteínas. Sus hallazgos proporcionan pautas importantes para futuros esfuerzos de biomimetismo, particularmente para la fabricación de dispositivos y la síntesis a nanoescala.
"Hemos hecho las primeras observaciones directas de que el concepto de un embudo de plegado con trampas de energía cinética para proteínas individuales se puede aplicar igualmente al ensamblaje de estructuras proteicas ordenadas". "dice Jim DeYoreo, un científico de la Fundición Molecular, un centro de nanociencia del DOE en Berkeley Lab, quien dirigió esta investigación junto con la química Carolyn Bertozzi del Berkeley Lab. "Nuestros resultados nos dicen que los esfuerzos para descubrir y codificar las reglas de diseño para el autoensamblaje de sistemas moleculares complejos deberán tener en cuenta el impacto de las trampas cinéticas asociadas con las transformaciones conformacionales".
DeYoreo y Bertozzi son los autores correspondientes de un artículo publicado por el procedimientos de la Academia Nacional de Ciencias (PNAS) que informó esta investigación. El artículo se titula "Observación directa de trampas cinéticas asociadas con transformaciones estructurales que conducen a múltiples vías de ensamblaje de la capa S". Los coautores del artículo fueron Seong-Ho Shin, Sungwook Chung, Babak Sanii y Luis Comolli.
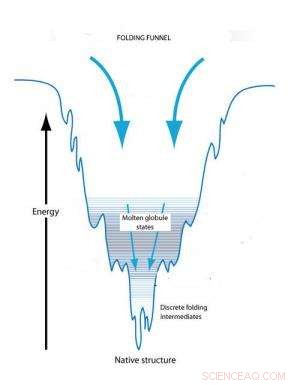
El concepto de embudo plegable explica el plegamiento de proteínas sobre la base de cambios conformacionales para alcanzar un estado de energía libre mínima. Una proteína desplegada comienza en un estado de alta libertad y alcanza su estado original con una energía mínima. Las trampas cinéticas a lo largo del camino pueden crear glóbulos fundidos transitorios e intermedios plegables.
Las proteínas son esencialmente nanomáquinas biomoleculares capaces de realizar numerosas tareas debido a su capacidad para plegarse en una multitud de formas y formas. Cuando las proteínas individuales se autoensamblan en estructuras ordenadas, el conjunto resultante a menudo adopta conformaciones que son bastante distintas de las de los componentes individuales.
"Por ejemplo, matrices de colágeno, que constituyen los andamios orgánicos de huesos y dientes, se construyen a partir de triples hélices de monómeros de colágeno individuales, "Dice DeYoreo." Estas hélices se ensamblarán aún más en fibrillas retorcidas altamente organizadas que exhiben una simetría pseudohexagonal ".
El concepto de embudo plegable explica el plegamiento de proteínas individuales sobre la base de cambios conformacionales para alcanzar un estado de energía libre mínima. Una proteína desplegada comienza en un estado de alta energía libre que hace que su conformación sea inestable. Inicialmente, hay una serie de posibles conformaciones tridimensionales que reducirían esta energía libre. Sin embargo, a medida que la proteína comienza a plegarse, la energía libre comienza a caer y el número de posibles conformaciones comienza a disminuir como el ancho de encogimiento de un embudo. El fondo del embudo se alcanza cuando se minimiza la energía libre y solo hay una conformación disponible. A medida que cae la energía libre, sin embargo, puede haber trampas cinéticas a lo largo del camino que pueden detener el proceso de plegado y mantener la proteína en conformaciones parcialmente plegadas, conocidos como glóbulos fundidos e intermedios plegables, durante largos períodos de tiempo. Eventualmente, estos estados conformacionales atrapados se transformarán en una conformación estable, pero la forma y la forma de esa conformación final están influenciadas por las trampas cinéticas.
"En un embudo de plegado de proteínas, Se presume que las paredes del embudo no son lisas y las protuberancias y valles resultantes definen trampas cinéticas, "Dice DeYoreo." Esta imagen física del plegamiento se ha explorado con cierto detalle a nivel de una sola molécula, pero no se ha considerado para el autoensamblaje de proteínas en arquitecturas extendidas a pesar de que las transformaciones conformacionales son parte integral del proceso de autoensamblaje ".
DeYoreo, Bertozzi y sus colegas tomaron medidas para corregir este déficit de conocimiento mediante el estudio de las proteínas de la capa superficial (capa S) que se autoensamblan en una membrana cristalina alrededor de las células individuales de bacterias y arqueas. Esta membrana externa sirve como el primer punto de contacto entre el microbio y su entorno y es clave para la capacidad del microbio para sobrevivir. Usando microscopía de fuerza atómica in situ (AFM), Los investigadores obtuvieron imágenes en tiempo real y en el atrapamiento cinético de nivel molecular durante el autoensamblaje 2D de estructuras de proteínas de la capa S en superficies de mica.
"Observamos que el autoensamblaje de las proteínas de la capa S sigue dos vías diferentes, uno que conduce directamente a la final de baja energía, estado ordenado, y el otro conduce a una trampa cinética ocupada por un estado transitorio de larga duración que es más desordenado, "Dice DeYoreo." Aunque cualquiera de los estados es fácilmente accesible durante la nucleación del cristal, si el sistema cae en el estado de alta energía, escapar a la final, el estado de baja energía está fuertemente impedido a temperatura ambiente. Esto demuestra la importancia de las trampas cinéticas para determinar la vía de cristalización de la capa S y sugiere que el concepto de embudos de plegado es igualmente válido para el autoensamblaje de estructuras proteicas extendidas ".