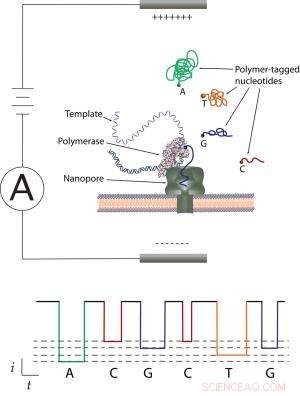
Esquema de una membrana artificial, a través del cual un voltaje fuerza un fluido ionizado a través del nanoporo. Los nucleótidos en una hebra de ADN se marcan primero con polímeros de diferentes tamaños, y luego la hebra se pasa cerca de la abertura del nanoporo, donde una polimerasa escinde los polímeros y los pasa uno por uno a través del nanoporo. Mientras pasan el poro produce una firma de bloqueo de corriente iónica única debido a la estructura química distintiva de la etiqueta, determinando así la secuencia de ADN. Crédito:NIST
(Phys.org) —Rapid, La secuenciación genética precisa pronto estará al alcance de todos los consultorios médicos si las investigaciones recientes del Instituto Nacional de Estándares y Tecnología (NIST) y la Escuela de Ingeniería y Ciencias Aplicadas de la Universidad de Columbia se pueden comercializar de manera efectiva. El equipo ha demostrado un potencial bajo costo, forma confiable de obtener las secuencias de ADN completas de cualquier individuo utilizando una especie de lector de cinta de teletipo molecular, potencialmente permitiendo una fácil detección de marcadores de enfermedades en el ADN de un paciente.
Si bien la secuenciación del genoma de una especie animal por primera vez es tan común que ya casi no es noticia, es menos conocido que secuenciar el ADN de cualquier individuo es un asunto costoso, cuesta muchos miles de dólares utilizando la tecnología actual. El genoma de un individuo lleva marcadores que pueden proporcionar una advertencia anticipada del riesgo de enfermedad, pero necesitas un ayuno Una forma confiable y económica de secuenciar los genes de cada paciente para aprovecharlos al máximo. Igualmente importante es la necesidad de secuenciar continuamente el ADN de un individuo a lo largo de su vida. porque el código genético puede ser modificado por muchos factores.
El nuevo método determina las secuencias de ADN uniendo distintas "etiquetas" moleculares a cada uno de los cuatro componentes químicos, o "bases, "que comprenden la información genética en una hebra de ADN, abreviada como A, GRAMO, C y T. Cada una de estas etiquetas de polímero se puede cortar de la hebra y pasar, uno a uno, a través de un agujero del tamaño de un nanómetro en una membrana. Un flujo constante de fluido e iones fluye a través de este "nanoporo, "que es lo suficientemente grande como para contener solo una etiqueta a la vez. Como las etiquetas de polímero son de diferentes tamaños, el cambio en la corriente eléctrica causado por el flujo de fluido alterado muestra cuál de las cuatro bases se encuentra en cada punto de la hebra de ADN.
Los nanoporos y su interacción con las moléculas de polímero han sido un foco de investigación desde hace mucho tiempo del científico del NIST John Kasianowicz. Su grupo colaboró con un equipo dirigido por Jingyue Ju, director del Centro de Tecnología Genómica e Ingeniería Biomolecular de Columbia, al que se le ocurrió la idea de etiquetar los componentes básicos del ADN para la secuenciación de una sola molécula mediante la detección de nanoporos. Kasianowicz demostró la capacidad de discriminar entre las etiquetas de polímero, su colega del NIST Joseph Robertson, y otros. La Universidad de Columbia ha solicitado patentes para la comercialización de la tecnología.
Kasianowicz estima que la técnica podría identificar un bloque de construcción de ADN con una precisión extremadamente alta con una tasa de error de menos de uno en 500 millones. y el equipo necesario estaría al alcance de cualquier proveedor médico. "El corazón del secuenciador sería un amplificador operacional que costaría mucho menos de $ 1, 000 por una compra única, " él dice, "y el costo de los materiales y el software debería ser trivial".
Kasianowicz agrega que una empresa privada podría crear una gran variedad de nanoporos que puedan analizar el genoma de un solo individuo dividido en muchas hebras cortas de ADN, cada uno de los cuales podría secuenciarse rápidamente. Una matriz de este tipo podría proporcionar potencialmente la secuenciación de bajo costo necesaria para el uso médico de rutina.