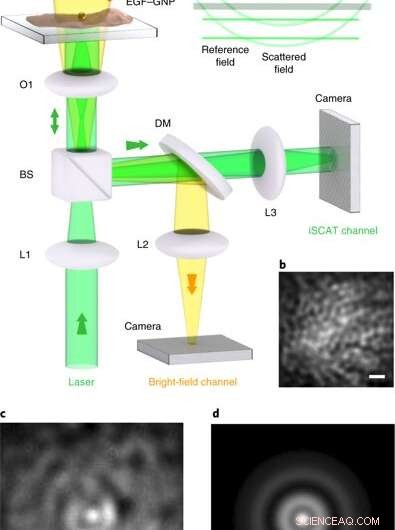
Microscopía iSCAT en células vivas. a, Disposición experimental del microscopio iSCAT para la obtención de imágenes de células vivas. Las células se cultivan en placas en una placa con fondo de vidrio bajo medio Leibowitz. (a) la micropipeta entrega las sondas EGF-GNP directamente en el cultivo celular, donde se dirigen específicamente a la proteína EGFR en la membrana celular. El canal de iluminación de campo brillante desde arriba ayuda a inspeccionar el cultivo, pero no es necesario para la obtención de imágenes iSCAT. L1 – L3, lentes; O1, × 100 objetivo; BS, Divisor de haz 90:10; DM, Espejo dicroico de paso corto de 590 nm. Las imágenes iSCAT se realizaron con intensidades de iluminación de 1 a 8 kW cm − 2, que se sabe que son viables para HeLa en la longitud de onda de interés. Recuadro, frentes de onda de los campos que contribuyen a la señal iSCAT. (B), Una sección de la membrana de la célula HeLa antes del etiquetado, visto a través de iSCAT de reflexión. (C), Imagen iSCAT de la membrana celular que incluye una sonda EGF-GNP unida. (D), El PSF extraído de c. Las barras de escala en b – d son 1 µm. Crédito: Fotónica de la naturaleza , doi:10.1038 / s41566-019-0414-6
Las funciones celulares están dictadas por el intrincado movimiento de las proteínas en las membranas que abarcan una escala de nanómetros a micrómetros, dentro de un marco de tiempo de microsegundos a minutos. Sin embargo, este rico parámetro del espacio es inaccesible usando microscopía de fluorescencia, aunque está al alcance del seguimiento de partículas por dispersión interferométrica (iSCAT). La nueva técnica iSCAT es, sin embargo, altamente sensible a proteínas simples y sin marcar, provocando así una tinción de fondo no específica como un desafío sustancial durante la formación de imágenes celulares.
En un estudio reciente, Richard W. Taylor y sus colegas de los departamentos interdisciplinarios de Física y Biología en Alemania desarrollaron un nuevo enfoque de procesamiento de imágenes para superar esta dificultad. Utilizaron el método para rastrear el receptor del factor de crecimiento epidérmico transmembrana (EGFR) con precisión de escala nanométrica en tres dimensiones (3-D). La técnica permitió obtener imágenes de microsegundos a minutos. Los científicos proporcionaron ejemplos de movimiento y confinamiento a nanoescala utilizando el método para obtener imágenes de procesos ubicuos como la difusión en las membranas plasmáticas, transporte en filopodios y movimiento de rotación durante la endocitosis. Los resultados ahora se publican en Fotónica de la naturaleza .
Si bien el progreso constante en la microscopía de fluorescencia ha permitido a los científicos monitorear los eventos celulares a escala nanométrica, Aún queda mucho por hacer con los sistemas de imágenes avanzados. Los desafíos de la microscopía de fluorescencia se produjeron debido a la tasa de emisión finita de una fuente fluorescente (molécula de tinte o punto cuántico semiconductor), donde muy pocas emisiones de fotones durante un período de tiempo muy pequeño impidieron la obtención de imágenes efectivas o prolongadas. La principal dificultad de la microscopía basada en la dispersión es relativa a la sonda nanoscópica, que compite con el ruido de fondo y una baja relación señal / ruido (SNR); limitando el potencial de las imágenes a solo unos pocos nanómetros en experimentos de seguimiento de alta velocidad.

Vídeo sin procesar de una nanopartícula de oro con factor de crecimiento epidérmico (EGFR-GNP) que se difunde en una membrana celular HeLa. Crédito: Fotónica de la naturaleza , doi:10.1038 / s41566-019-0414-6
En el presente trabajo, Taylor y col. utilizó microscopía de dispersión interferométrica (iSCAT) para rastrear proteínas en las membranas de células vivas. El método podría visualizar interacciones sonda-celda para comprender la dinámica entre la difusión y la topología local. Durante los experimentos, Los científicos utilizaron nanopartículas de oro (GNP) para marcar los receptores del factor de crecimiento epidérmico (EGFR) en las células HeLa. Los EGFR son proteínas transmembrana de tipo I que pueden detectar y responder a señales extracelulares, cuya señalización aberrante está relacionada con una variedad de enfermedades. Taylor y col. mostró la proteína etiquetada con GNP como un 'nano-rover' que mapeó la nano-topología de características celulares como terrenos de membrana, estructuras de filopodia y clatrina. Proporcionaron ejemplos de subdifusión y movimiento de confinamiento nanoscópico de una proteína en 3-D a alta resolución temporal y largos puntos de tiempo.
En los experimentos, Taylor y col. introdujo las sondas de nanopartículas de oro del factor de crecimiento epidérmico (EGF-GNP) en la cámara de muestras del microscopio utilizando una micropipeta para marcar los EGFR (receptores del factor de crecimiento epidérmico) en las células HeLa y verificó que las sondas estimulaban los EGFR. Estudios anteriores ya habían indicado que el tamaño de la sonda podría influir en las tasas de difusión de lípidos en membranas sintéticas, aunque no afectaron al modo de difusión. Adicionalmente, en células vivas, el apiñamiento molecular fue insignificante para partículas iguales o menores de 50 nm.
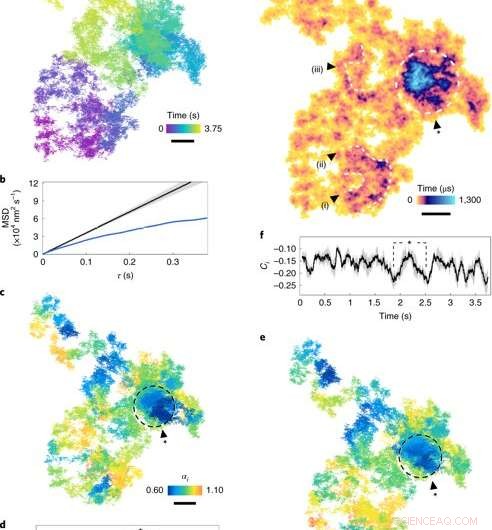
Difusión en la membrana plasmática. (a), Una trayectoria de difusión lateral (tiempo de exposición de 17,5 μs, consulte la escala de colores para ver la cronología). (B), MSD (desplazamiento cuadrático medio) frente a τ. La curva azul muestra el MSD de a. La curva negra es difusión normal simulada (α =1), con el sobre gris que indica la incertidumbre. (C), El exponente de difusión de las ventanas rodantes (escala de colores) sobre la trayectoria. Las regiones de subdifusión (α <1) se indican con tonos más oscuros. (D), αi a través del tiempo. El sombreado gris representa una incertidumbre media de 7 ± 4%, correspondiente a un intervalo de confianza del 95% para una ventana de 100 ms (1, 000 fotogramas) y τ =250 μs. Los puntos marcados con el asterisco corresponden al círculo en c. (mi), La dirección del paso Ci para las ventanas rodantes a lo largo de la trayectoria. (F), La dirección del paso Ci trazada a través del tiempo, con el sombreado que denota incertidumbre. (gramo), Gráfica de ocupación ATOM con tiempo de residencia (escala de colores). El tamaño del contenedor corresponde al error de localización. Regiones notables de ocupación extendida, marcados como bucles y remolinos (i) - (iii), son indicativos de estructuras nanoscópicas persistentes. La región cerrada representa un parche denso de notable subdifusión. Barras de escala, 100 nm. Crédito: Fotónica de la naturaleza , doi:10.1038 / s41566-019-0414-6
Taylor y col. verificó estos dos casos concretos en el presente trabajo comparando PNB de diámetros variables a 48 nm y 20 nm. Luego, los científicos realizaron estudios fluorescentes y bioquímicos para sugerir que los GNP recubiertos de EGF activaban la señalización de EGFR, al igual que los EGF disponibles gratuitamente, lo que indica que la etiqueta no obstaculizó las funciones biológicas. Para superar el ruido de fondo relacionado con las imágenes moleculares, los científicos implementaron un nuevo algoritmo, que extrajo la función de dispersión de puntos iSCAT completa (iSCAT-PSF) directamente de cada cuadro para mayor claridad. Dado que las técnicas existentes no pueden visualizar características con alta resolución espacial y temporal, muchos detalles sobre la actividad intracelular siguen siendo objeto de debate. En respuesta, el nuevo método de Taylor et al. ofreció una gran cantidad de heterogeneidades dinámicas en 3-D para arrojar luz sobre el movimiento de las proteínas intracelulares.
Los científicos primero estudiaron cuantitativamente la subdifusión en la membrana plasmática al considerar un ejemplo 2-D del viaje del EGFR en la membrana de una célula HeLa viva. Para esto, calcularon el desplazamiento cuadrático medio (MSD) para toda la trayectoria del movimiento. Taylor y col. no necesitó hacer suposiciones sobre la naturaleza de la difusión o su paisaje geográfico durante el cálculo. Ellos midieron la ocurrencia de confinamientos y barreras biológicas difractivas observando el grado de correlación direccional entre dos pasos vectoriales a lo largo de un lapso de tiempo.

Difusión sobre un filopodio. Crédito: Fotónica de la naturaleza , doi:10.1038 / s41566-019-0414-6
Luego, los científicos evaluaron la popularidad de cada píxel de trayectoria en el espacio mediante la introducción de un mapa de ocupación temporal acumulada (ATOM). En esta técnica, dividieron el plano lateral de la trayectoria en contenedores de tamaño nanométrico y contaron la presencia de la partícula en cada contenedor. Los resultados indicaron la disposición de nanoestructuras en bucles y giros dentro de una vida útil mínima de 250 nanosegundos (5000 cuadros) para representar potencialmente un paso preendocítico. En total, las observaciones simuladas mostraron cómo la difusión de proteínas se ve afectada por la subestructura de la célula.
La técnica de microscopía iSCAT permitió a los científicos registrar efectos durante un período de tiempo muy largo, que utilizaron junto con las capacidades de imágenes en 3-D para seguir los EGFR en un filopodio. Los filopodios son protuberancias celulares parecidas a varillas biológicamente que contienen haces de filamentos de actina de hasta 100 a 300 nm de diámetro y 100 µm de longitud. Las nanoestructuras pueden detectar estímulos mecánicos para la quimioatracción o repulsión en el microambiente celular mientras proporcionan sitios para la unión celular. La unión de ligandos y la activación de EFGR en filopodios ocurrieron a bajas concentraciones de EGF, seguido de su asociación con filamentos de actina y transporte retrógrado de EFGR al cuerpo celular.
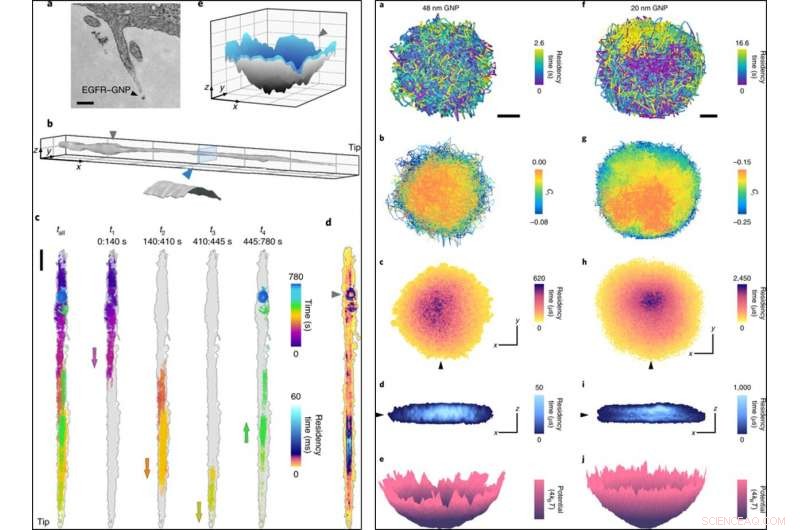
IZQUIERDA:(a) Imagen TEM (microscopio electrónico de transmisión) de un filopodio que incluye un EGFR-GNP. (B), Una superficie de filopodio reconstruida a partir de 780, 000 puntos de trayectoria con un error de localización de σx, y =2 nm registrados en 1, 000 fps. Recuadro, Corte transversal que representa una superficie cilíndrica de 150 nm de diámetro después de tener en cuenta el tamaño del PNB. (C), Una trayectoria cruda de 13 minutos (izquierda) dividida en cuatro partes posteriores que revelan el viaje hacia y desde la punta, con flechas que marcan la dirección del movimiento de la red. (D), Una gráfica de ATOM de c, corregido por la deriva del filopodio. (mi), Una interpolación de superficie de los últimos 80 s. El confinamiento en forma de anillo en la fase final (marcado con un triángulo) es un pozo en 3D. Las barras de escala son 200 nm (a), 1 μm (x, y) y 200 nm (z) (b), 1, 000 nm (c) y 100 nm (x, y) y 50 nm (z) (e). DERECHA:(a), Una trayectoria lateral de una sonda GNP de 48 nm. Barra de escala, 100 nm. Un muestreo temporal más bajo de este confinamiento habría subestimado la extensión del límite. (B), Ci de la trayectoria (utilizando un intervalo de tiempo de cinco fotogramas), que muestra una difusión parcialmente obstaculizada con una propensión a una difusión más libre en el centro. (C), Un diagrama de ATOM de a. (D), Un corte a través del gráfico 3D-ATOM a lo largo de la línea del triángulo negro en c muestra que la ocupación favorece una región más interna parecida a un disco. Los ejes denotan 100 nm tanto en c como en d. (mi), Conversión de la ocupación temporal 2D de c en una distribución de energía potencial efectiva. (f – j), Equivalente a a – e, pero para una sonda GNP de 20 nm. Crédito: Fotónica de la naturaleza , doi:10.1038 / s41566-019-0414-6
De este modo, los científicos obtuvieron información sobre los detalles nanoscópicos de la difusión a lo largo del filopodio y registraron los datos durante 13 minutos. Analizaron la trayectoria 3-D para crear la topografía del filopodio utilizando nanopartículas de oro como un 'nano rover' y mapearon la topología de la superficie de las estructuras celulares para un examen más profundo. Trazaron la trayectoria ATOM (mapa de ocupación temporal acumulada) y encontraron que la representación 3-D era consistente con el paso biológico de la invaginación de la membrana pre-endocítica.
Las técnicas de microscopía de alta velocidad como iSCAT son necesarias para obtener información temporal de alta resolución y evitar efectos borrosos durante la obtención de imágenes basadas en la localización de nanopartículas. Los científicos demostraron esta característica al registrar la difusión confinada a 30, 000 fps (fotogramas por segundo) con GNP de 48 nm y 20 nm. Siguieron los experimentos con rastreo 3D de proteínas a ultra alta velocidad a 66, 000 fps utilizando un tiempo de exposición corto de 10 µs dentro de un tiempo de duración de 3,5 segundos. Las imágenes de microscopía iSCAT rápidas proporcionaron más evidencia para revelar las intrincadas características de los eventos endocíticos en relación con la endocitosis mediada por clatrina en las células HeLa cuando se simulan mediante bajas concentraciones de EGF.
De este modo, Taylor y col. señaló que la nueva técnica podría registrar fielmente información nano-topográfica. Los resultados coincidieron con las observaciones registradas con microscopía electrónica de transmisión (TEM) sin diferencias significativas en la reducción del tamaño de la sonda de 48 nm a 20 nm, al tiempo que proporciona nuevos conocimientos. Los nuevos conocimientos incluyeron detalles de subdifusión, confinamiento nanoscópico, Contornos tridimensionales de estructuras de filopodios y clatrinas a nanoescala. Los científicos tienen la intención de combinar iSCAT con microscopía de fluorescencia de superresolución in situ para comprender las trayectorias de las proteínas, virus y otras entidades biológicas nanoscópicas. Taylor y col. tienen como objetivo avanzar en los métodos de análisis de imágenes para rastrear PNB menores de 20 nm en el futuro y creen que la nueva tecnología y la optimización adicional les permitirá comprender específicamente el ciclo de vida de los virus sin usar una etiqueta externa para el rastreo.
© 2019 Science X Network