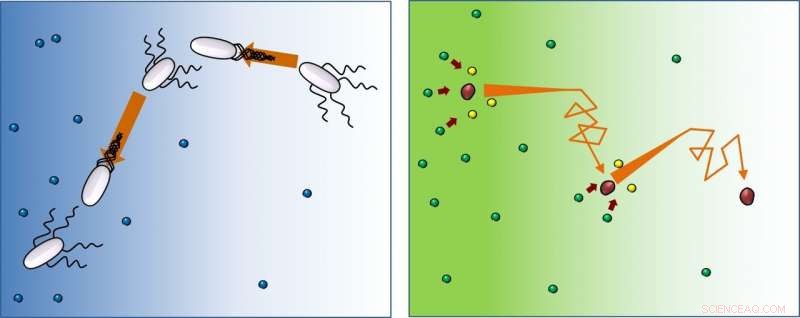
En presencia de comida (azul), las bacterias (blancas) nadan en línea recta, luego cambia de orientación (caída), y siga este patrón una y otra vez. Este estudio muestra que las enzimas (rojo) se mueven de la misma manera, pero después de reaccionar con sustratos (de verde a amarillo), continúan corriendo y cayendo lejos de la mayor concentración de sustratos. Crédito:IBS
Aunque juntar las palabras "aleatorio" y "sesgado" puede parecer un conflicto, estos son los atributos que describen cómo las bacterias navegan y buscan su alimento. Siguen un camino aleatorio pero sesgado hacia la dirección de la fuente de nutrientes. Científicos del Center for Soft and Living Matter, dentro del Instituto de Ciencias Básicas (IBS, Corea del Sur), observó que las enzimas, las moléculas que ayudan a que las reacciones biológicas ocurran más rápidamente, moverse con un patrón similar, pero en la ruta opuesta, viajando aleatoriamente hacia el área con menos sustratos. Estos resultados, obtenido con nuevas tecnologías ópticas y publicado como artículo inaugural en Actas de la Academia Nacional de Ciencias (PNAS) , son particularmente intrigantes, ya que suponen una desviación de la visión tradicional.
Miles de enzimas promueven una multitud de reacciones en nuestro cuerpo, cada uno de los cuales sustenta nuestras vidas. Las enzimas actúan sobre otras sustancias químicas llamadas sustratos y aceleran las reacciones químicas que convierten los sustratos en productos. Para hacer eso, las enzimas y los sustratos deben encontrarse. Originalmente se creía que las enzimas y sus sustratos correspondientes chocaban casualmente entre sí:"Los libros de texto estándar mencionan que se supone que las enzimas y los sustratos se acercan" de alguna manera, " interactuar, y así es como ocurren las reacciones. Intentamos profundizar en la parte "de alguna manera" de este proceso. Después de recopilar miles de eventos, llegamos a la conclusión de que la caminata de las enzimas está sesgada al azar, lo que significa que son microscópicamente direccionales, como bacterias, "explica Ah-Young Jee, primer autor de este estudio.
El movimiento de las bacterias que nadan se conoce como "correr y caer":correr, gira y repite. Proceden en una dirección, luego cambia de dirección al azar, y repetir. Sin embargo, mientras que las bacterias se orientan hacia el suministro de alimentos, las enzimas se mueven en la dirección de menor concentración de sustrato. "Las moléculas carecen de capacidad para tomar decisiones, pero sorprendentemente se mueven hacia áreas con menos sustrato. En efecto, el sustrato introduce el sesgo de dirección, y cuanto mayor sea la concentración de sustrato que usamos en el experimento, cuanto más fuerte es esta tendencia. Suponemos que podría ser una forma de hacer que la concentración de producto sea uniforme alrededor del medio, incluso cuando la concentración de sustrato no lo es, "dice el profesor Tsvi Tlusty, quien ofreció la comprensión teórica de esta investigación.
Como el paso de cada enzima tiene solo 50 nanómetros de longitud, Se necesitan dispositivos extremadamente precisos para medir su velocidad y orientación. El equipo de investigación dirigido por Steve Granick, observó el movimiento enzimático con una tecnología de microscopía de superresolución, conocida como espectroscopia de correlación de fluorescencia de reducción de emisión estimulada, o STED-FCS para abreviar.

A) Enzima-sustrato-tampón (E + S + B) entra en la entrada izquierda, y se inyecta tampón enzimático (E + B) en la entrada derecha. De este modo, la concentración de enzima es uniforme en todo el chip, mientras que la concentración de sustrato es mayor en el lado izquierdo (azul) disminuyendo gradualmente hacia el derecho (blanco). El rectángulo de guiones indica el área donde se estudia con gran detalle el movimiento de la enzima utilizando la tecnología de microscopía STED-FCS. B) La concentración de la enzima ureasa (negra) y la concentración de urea (su sustrato, gris) se trazan contra la posición dentro de la zona FCS, entre 0 y 2,5 milímetros del rayo láser. La escala en cada punto es una barra de error, mostrando la desviación estándar en cinco mediciones repetidas. C) El coeficiente de difusión (Da), que indica qué tan rápido se mueve la enzima, no cambia en ausencia de sustrato, pero aumenta cuando el sustrato está presente, lo que significa que la enzima se mueve más rápido hacia la derecha. Crédito:IBS, publica en PNAS
En el método FCS, las enzimas están decoradas con moléculas fluorescentes y su paso a través de un pequeño punto es detectado por un rayo láser. Se recopila información estadística, como el número de enzimas que pasan por el punto y su velocidad (tasa de difusión). El equipo complementó FCS con tecnología STED, uno de los puntos fuertes de este centro de investigación. STED concentra el rayo láser en un área extremadamente pequeña, permitiendo una medición más refinada de la posición de las enzimas. El rayo láser FCS cubre un área de alrededor de 250 nanómetros de diámetro, que es aproximadamente 25 veces el diámetro de la enzima (10 nanómetros). STED-FCS es más preciso ya que reduce el diámetro del haz a 50 nanómetros, que es comparable a la distancia del paso de una enzima.
Los investigadores también diseñaron un nuevo chip de microfluidos, formado por canales de un espesor de un micrómetro por donde pueden pasar los líquidos. Estudios previos, utilizando FCS sin STED y un chip de microfluidos diferente, sugirió que las enzimas se muevan hacia el área con mayor concentración de sustrato. Sin embargo, esta nueva tecnología permite observaciones más profundas y mostró lo contrario. Los autores piensan que los resultados difieren porque la información detallada sobre el movimiento de la enzima se canceló utilizando un rayo láser más grande. Sería similar a localizar a alguien dentro de 50 kilómetros cuadrados o dentro de 2 kilómetros cuadrados, el último es más preciso. Crearon condiciones donde la enzima se distribuye uniformemente dentro del chip, pero la concentración de sustrato varía de izquierda a derecha:desde una concentración mínima en el lado derecho del chip, a una concentración máxima a la izquierda. Luego, utilizaron FCS para observar la concentración y la velocidad de la enzima dentro del chip, y encontró que la enzima tiende a acelerarse hacia el área con menor concentración de sustrato (es decir, hacia el lado derecho del chip). En lugar de, cuando no hay sustrato presente, las enzimas no cambian su velocidad. Es más, las capacidades de superresolución de STED-FCS mostraron una dinámica de carrera y caída.
El equipo observó el mismo comportamiento con dos parejas enzima-sustrato por separado:ureasa-urea y acetilcolinesterasa (AChE) -acetilcolina. Algunos microorganismos utilizan la primera enzima para transformar la urea en amoniaco y ácido carbónico. este último juega un papel fundamental en las uniones neuromusculares.
En el futuro, El equipo de investigación tiene como objetivo ampliar la complejidad del estudio y probar las condiciones de forma más similar a la vida real:"Este estudio analizó el comportamiento de una enzima en respuesta a un sustrato. A continuación, planeamos mirar varias enzimas al mismo tiempo. Nuestro cuerpo funciona con una cascada de reacciones, lo que significa que una reacción desencadena la siguiente:la enzima A desencadena la enzima B, luego B dispara C, etcétera. El análisis STED-FCS podría ser una herramienta clave para avanzar en la investigación metabólica al revelar cómo estos múltiples sistemas enzimáticos están relacionados orgánicamente. "concluye Jee.