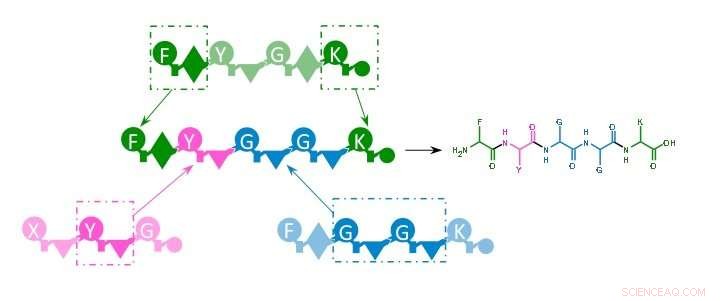
Diagrama esquemático del “sistema de caja de herramientas” de las enzimas NRPS para la producción de nuevos principios activos. Fragmentos de sistemas naturales (verde, magenta, azul) se vuelven a ensamblar en un nuevo orden (centro) y luego producen un producto natural que no se ha formado así en la naturaleza antes (derecha). Crédito:Goethe-Universität Frankfurt am Main
Los microorganismos a menudo producen productos naturales paso a paso, similar a una línea de montaje. Ejemplos de tales enzimas son péptido sintetasas no ribosomales (NRPS). Los investigadores de la Universidad Goethe de Frankfurt han logrado diseñar estas enzimas de tal manera que puedan producir productos naturales completamente nuevos.
Muchas terapias importantes, como antibióticos o fármacos inmunosupresores y anticancerígenos, se derivan de microorganismos. Este es también el caso de varios péptidos diferentes que se producen en la célula microbiana con la ayuda de las enzimas NRPS. Un NRPS funciona como una línea de ensamblaje en una fábrica de automóviles moderna:se agregan piezas nuevas al chasis básico en cada estación de trabajo hasta que un automóvil terminado sale de la planta al final. En el caso del NRPS, se selecciona un determinado aminoácido, activados y procesados en cada estación (conocidos como módulos) de modo que lineal, Al final surgen péptidos cíclicos o más modificados que también pueden transportar aminoácidos inusuales.
Aunque los principios fundamentales de NRPS se conocen desde hace mucho tiempo, hasta la fecha apenas era posible modificar estas enzimas. En los pocos casos en los que se intercambiaron con éxito módulos individuales, la producción de productos naturales modificados disminuyó notablemente. Ensamblar enzimas completamente nuevas, que a su vez produciría productos naturales completamente nuevos, parecía totalmente imposible. El grupo de investigación dirigido por el profesor Helge Bode, Profesor de Merck Endowment de Biotecnología Molecular en la Universidad Goethe de Frankfurt, ahora ha logrado esto.
"En principio, usamos sistemas NRPS naturales de bacterias solo como bloques de construcción que reensamblamos de una nueva manera usando nuevas interfaces que hemos identificado, "dice Bode, explicando el enfoque de la investigación. Los rendimientos son comparables con la producción natural de estas sustancias naturales.
Mientras tanto, el método es tan sofisticado que incluso los principiantes pueden usarlo para producir nuevos péptidos y, por lo tanto, fármacos potenciales poco después de una introducción básica. Sin embargo, ha sido un largo camino. "Tuve la suerte de tener un equipo trabajando conmigo en este proyecto que no se dejó desanimar, fue muy diligente y capaz de pensar fuera de la caja, ", dice Bode." La interfaz que finalmente seleccionamos para ensamblar los bloques de construcción individuales es tal que ya no se respeta el orden modular clásico de la biosíntesis ".
El siguiente paso es modificar los primeros fármacos clínicos con este método y utilizar la biotecnología para producirlos. Es más, Se recopilará nueva información sobre la estructura de estos NRPS como parte del enfoque de investigación de LOEWE MegaSyn dirigido por Bode y el profesor Martin Grininger, también de la Universidad Goethe de Frankfurt. Esto permitirá mejorar aún más el método para permitir la modificación de clases relacionadas de productos naturales o incluso para producir bibliotecas completas de productos naturales. Los primeros resultados son muy prometedores.