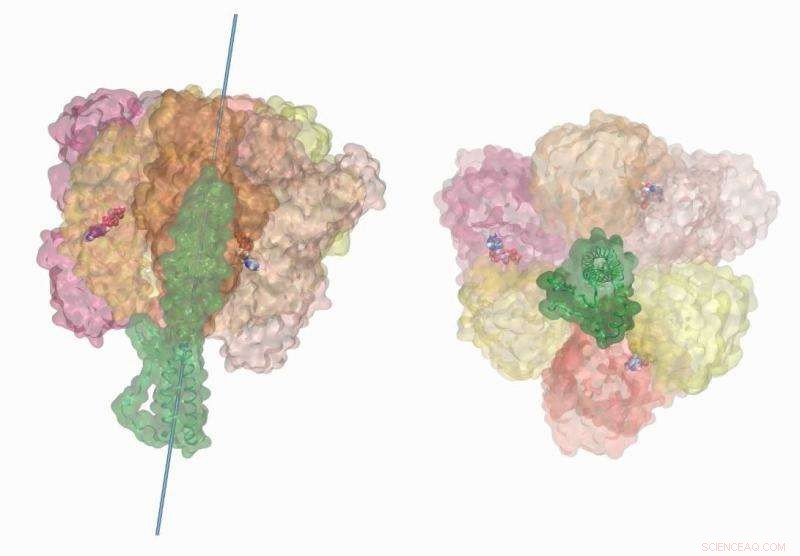
La rotación impulsada por la hidrólisis de ATP del tallo central en la ATP sintasa de tipo V de la bacteria Enterococcus hirae representa el acoplamiento quimio-mecánico del motor molecular. Estas simulaciones se llevaron a cabo en la supercomputadora Titán utilizando NAMD2.12. La visualización y el procesamiento de películas se completaron en VMD1.9.3. Crédito:Barry Isralewitz, Universidad de Illinois en Urbana-Champaign
A pesar de la gran diversidad entre los organismos vivos, la molécula utilizada para almacenar y transmitir energía dentro de aeróbicos, o el uso de oxígeno, células es notablemente el mismo. De bacterias a hongos, plantas y animales, trifosfato de adenosina (ATP) sirve como la moneda de energía universal de la vida, alimentando los procesos que las células necesitan para sobrevivir y funcionar.
En el transcurso de un día, un individuo normalmente utilizará el equivalente de su peso corporal en ATP; sin embargo, el cuerpo humano transporta solo una pequeña cantidad de la molécula a la vez. Eso significa que las células deben reciclarse constantemente o reponer su capacidad limitada, confiando en un motor molecular altamente eficiente llamado ATP sintasa para hacer el trabajo.
Como parte de un proyecto dedicado a modelar cómo las bacterias púrpuras unicelulares convierten la luz en alimento, un equipo de científicos computacionales de la Universidad de Illinois en Urbana-Champaign (UIUC) simuló una ATP sintasa completa con detalles de todos los átomos. El trabajo se basa en la primera fase del proyecto, un orgánulo fotosintético de 100 millones de átomos llamado cromatóforo, y brinda a los científicos una visión sin precedentes de una máquina biológica cuya eficiencia energética supera con creces la de cualquier sistema artificial.
Propuesto por primera vez bajo el liderazgo del difunto Klaus Schulten, pionero en el campo de la biofísica computacional y fundador del Grupo de Biofísica Teórica y Computacional en UIUC, la investigación ha progresado bajo la dirección de Abhishek Singharoy, co-investigador principal y becario postdoctoral de la National Science Foundation con el Centro de Física de Células Vivas de la UIUC.
Además de Singharoy, el equipo incluye miembros de los grupos de profesores de la UIUC Emad Tajkhorshid, Zaida Luthey-Schulten y Aleksei Aksimentiev; el científico investigador Melih Sener; y los desarrolladores Barry Isralewitz, Jim Phillips, y John Stone. El colaborador experimental Neil Hunter de la Universidad de Sheffield en Inglaterra también participó en el proyecto.
El equipo liderado por UIUC construyó y probó su megamodelo bajo una asignación de varios años otorgada a través del programa Innovative and Novel Computational Impact on Theory and Experiment en la supercomputadora Titan. un Cray XK7 administrado por la Instalación de Computación de Liderazgo de Oak Ridge (OLCF) del Departamento de Energía de EE. UU. (DOE), una instalación para usuarios de la Oficina de Ciencias del DOE ubicada en el Laboratorio Nacional Oak Ridge del DOE.
Usando Titán, el equipo produjo una herramienta virtual que puede predecir con todo detalle la producción de energía química de un sistema fotosintético en función de la cantidad de luz solar absorbida. La investigación podría algún día contribuir a la tecnología avanzada de energía limpia que incorpore conceptos biológicos.
"La naturaleza ha diseñado el cromatóforo de tal manera que puede generar suficientes ATP para que estas bacterias sobrevivan en entornos con poca luz, como el fondo de estanques y lagos, ", Dijo Singharoy." Nuestro trabajo capturó este proceso de conversión de energía con todos los detalles de los átomos y nos permitió predecir su eficiencia ".
Luz en movimiento
A menudo conocida como la planta de energía de la celda, La ATP sintasa es una enzima compleja que acelera la síntesis de sus precursores moleculares, difosfato de adenosina (ADP) y fosfato. Incrustado dentro de la membrana interna y externa del cromatóforo, el motor enzimático consta de tres partes principales:un rotor de iones, un tallo central, y un anillo de proteínas.
Similar a una rueda hidráulica que se hace girar por la fuerza de una corriente que fluye, el rotor de ATP sintasa aprovecha el movimiento estimulado electroquímicamente de iones, como protones o sodio, de alta concentración a baja concentración a través de la membrana. La energía mecánica resultante se transfiere al tallo central, que ayuda al anillo proteico a sintetizar ATP.
Notablemente, el proceso funciona igual de bien a la inversa. Cuando se acumulan demasiados iones en el lado exterior del cromatóforo, el anillo de la proteína ATP sintasa descompondrá el ATP en ADP, un proceso llamado hidrólisis, y los iones fluirán de regreso al lado interno.
"Normalmente, esperaría mucha pérdida de energía durante este proceso, como en cualquier motor hecho por el hombre, pero resulta que la ATP sintasa tiene muy poco desperdicio, "Dijo Singharoy." Cómo está diseñado este motor para minimizar la pérdida de energía es la pregunta que comenzamos a hacer ".
Similar a un técnico que desmonta un motor para comprender mejor cómo funciona, El equipo de Singharoy rompió el 300, 000 átomos de enzima en sus partes constituyentes. Basándose en décadas de investigación sobre la ATP sintasa, modelos pasados, y nuevos datos experimentales proporcionados por un equipo japonés dirigido por Takeshi Murata del Centro RIKEN para Tecnologías de Ciencias de la Vida, el equipo construyó y simuló las piezas del rompecabezas de la ATP sintasa de forma independiente y conjunta en Titán.
Para capturar procesos importantes que se desarrollan en escalas de tiempo de milisegundos, Singharoy, en colaboración con Christophe Chipot del Centro Nacional Francés de Investigación Científica y Mahmoud Moradi de la Universidad de Arkansas, implementó estratégicamente el código de dinámica molecular NAMD. El equipo ejecutó una estrategia de conjunto, siguiendo el movimiento de alrededor de 1, 000 réplicas de ATP sintasa simultáneamente con pasos de tiempo de 2 femtosegundos, o 2, 000 billonésimas de segundo. En total, el equipo acumuló 65 microsegundos (65 millonésimas de segundo) de tiempo de simulación, utilizando esta información para extrapolar los movimientos que ocurren en el transcurso de un milisegundo (1 milésima de segundo).
Como resultado, el equipo identificó movimientos giratorios previamente indocumentados en el anillo de proteínas que ayudan a explicar la eficiencia del motor molecular. Similar, Las simulaciones del equipo capturaron la elasticidad similar a una banda elástica del tallo central de la enzima. El equipo de Singharoy estimó que cuando se combina con el anillo de proteínas, el tallo absorbe alrededor del 75 por ciento de la energía liberada durante la hidrólisis.
Adicionalmente, Las simulaciones del anillo de proteínas por sí mismas revelaron una unidad que puede funcionar de forma independiente, un hallazgo informado en experimentos pero no en detalle computacional. "Incluso en ausencia del tallo central, el anillo proteico en sí mismo es capaz de hidrólisis de ATP. No es muy eficiente, pero tiene la capacidad, "Dijo Singharoy.
El panorama
Después de simular su modelo completo de ATP sintasa, el equipo de UIUC incorporó la enzima en su modelo de cromatóforo previamente construido para obtener la imagen más completa de un sistema fotosintético hasta la fecha.
Con este panel solar biológico virtual, el equipo podría medir cada paso del proceso de conversión de energía, desde la recolección de luz, a la transferencia de electrones y protones, a la síntesis de ATP y comprender mejor sus fundamentos mecánicos.
El cromatóforo de la naturaleza está diseñado para poca intensidad de luz, absorbiendo únicamente entre el 3 y el 5 por ciento de la luz solar en un día normal. El equipo, a través de los esfuerzos de Sener, encontró que esta tasa de absorción se traduce en alrededor de 300 ATP por segundo, que es la cantidad que necesita una bacteria para mantenerse viva.
Habiendo estudiado el diseño de la naturaleza, el equipo ahora quería ver si podía mejorarlo. Suponiendo la misma cantidad de intensidad de luz, el equipo diseñó un cromatóforo artificial con una composición de proteínas decididamente antinatural, potenciando la presencia de dos tipos de proteínas especializadas. El análisis del nuevo diseño predijo una triplicación de la producción de ATP del sistema fotosintético, abriendo la posibilidad de optimización guiada por humanos del cromatóforo.
"Podría potencialmente modificar genéticamente un cromatóforo o cambiar su concentración de proteínas, ", Dijo Singharoy." Estas predicciones prometen generar nuevos desarrollos en la fotosíntesis artificial ".
Bajo su última asignación INCITE, el equipo de UIUC está girando hacia la conversión de energía en una forma de vida diferente:los animales. Tomando lo que ha aprendido al modelar la fotosíntesis en bacterias púrpuras, el equipo está modelando la respiración celular, el proceso que utilizan las células animales para convertir los nutrientes en ATP.
"Tienes al menos dos proteínas en común entre la respiración y la fotosíntesis, "dijo Singharoy, quien continúa su participación en el proyecto como profesor asistente en la Universidad Estatal de Arizona. "La pregunta es ¿qué principios de diseño se trasladan a los organismos superiores?"
Vida in situ
La simulación del cromatóforo, completa con ATP sintasa, marca un cambio continuo en la biofísica computacional del análisis de partes celulares individuales (p. Ej., proteínas individuales y cientos de átomos) para analizar sistemas celulares completos (por ejemplo, cientos de proteínas y millones de átomos).
Schulten, que falleció en octubre de 2016, entendió mejor que la mayoría de la gente la importancia de usar computadoras para simular la naturaleza. En una entrevista en 2015, expuso su razón fundamental para modelar el cromatóforo. "La motivación es comprender un paso clave de la vida en la Tierra del que depende toda la vida en la actualidad. El 95 por ciento de la vida en la Tierra, en términos de energía, depende de la fotosíntesis, incluidos los humanos, " él dijo.
Schulten también entendió el hito de un orgánulo especializado representado en el camino hacia la simulación de un organismo unicelular completo. "No tenemos nada más pequeño que una celda a la que llamaríamos viva, ", dijo." Es la entidad viviente más pequeña, y queremos entenderlo ".
Con supercomputadoras de próxima generación, incluida la Cumbre de la OLCF, que estará en línea en 2018, el grupo de investigación que Schulten fundó en 1989 se prepara para asumir el gran desafío de simular una célula.
Bajo el liderazgo de Tajkhorshid, el equipo planea simular la primera celda de mil millones de átomos, incluidos los componentes básicos que una célula necesita para sobrevivir y crecer. Las mejoras a NAMD y el trabajo que se está realizando en el marco del programa Center for Accelerated Application Readiness del OLCF están ayudando a hacer realidad la visión de Schulten y otros.
"Seguimos avanzando, ", Dijo Singharoy." Nuestro estudio exhaustivo de un orgánulo completo con detalles de todos los átomos ha abierto la puerta a una celda completa con detalles de todos los átomos ".