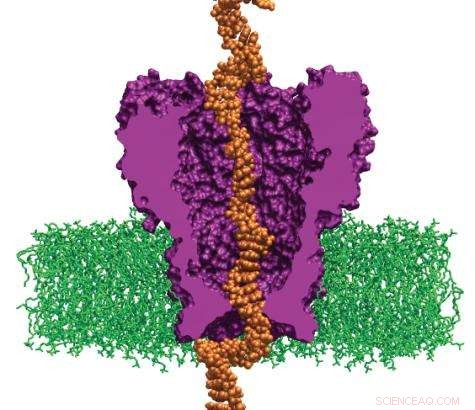
Los científicos simulan la interacción del ADN con una proteína modificada. El sistema puede ralentizar las hebras de ADN que viajan a través de los poros lo suficiente como para leer el genoma individual de un paciente. (Imagen cortesía de Aleksei Aksimentiev)
(PhysOrg.com) - El Proyecto Genoma Humano allanó el camino para la genómica, el estudio del genoma de un organismo. La genómica personalizada puede establecer la relación entre las variaciones de la secuencia de ADN entre los individuos y sus condiciones de salud y respuestas a medicamentos y tratamientos. Para hacer de la secuenciación del genoma un procedimiento de rutina, sin embargo, el tiempo debe reducirse a menos de un día y el costo a menos de $ 1, 000, una hazaña que no es posible con los conocimientos y tecnologías actuales.
En 2008, un equipo de investigación dirigido por Aleksei Aksimentiev, profesor asistente en el departamento de física de la Universidad de Illinois-Urbana-Champaign, inició un proyecto para crear máquinas para la secuenciación del genoma personal que serán más accesibles para los hospitales. Usando Jaguar del Laboratorio Nacional Oak Ridge, una de las supercomputadoras más rápidas del mundo, Aksimentiev y su equipo están desarrollando un enfoque de nanoporos, lo que promete una reducción drástica en el tiempo y los costos de secuenciación del ADN. Su investigación revela la forma del ADN que se mueve a través de un solo nanoporo, un poro de proteína de una milmillonésima parte de un metro de ancho que atraviesa una membrana. A medida que el ADN atraviesa el poro, un detector lee la secuencia de nucleótidos (componentes básicos del ADN).
"El principal obstáculo de la secuenciación utilizando las generaciones anteriores de nanoporos biológicos y sintéticos fue la incapacidad de identificar la secuencia de ADN con una resolución de un solo nucleótido, ", Dijo Aksimentiev." Los nucleótidos pasaron demasiado rápido a través del nanoporo para que los científicos pudieran secuenciar el ADN ".
El grupo de Aksimentiev utiliza el nanoporo MspA, una proteína de ingeniería. Su secuencia debe modificarse para unirse con más fuerza a la hebra de ADN en movimiento. MspA es una plataforma ideal para secuenciar el ADN porque los científicos ahora pueden medir las presas en el poro, lo que podría ralentizar el viaje del ADN a través de la proteína. Alterar la proteína MspA para optimizar las presas requiere mucho tiempo y es costoso en un laboratorio, pero simple en una computadora. Por ejemplo, alterar la proteína de alguna manera, los científicos deben determinar si la mutación particular que introducen es estable y si la idea es razonable. Por lo tanto, los científicos primero simulan MspA para decidir sobre una mutación para inducir y probar ideas de alto riesgo antes de implementarlas en un experimento.
El equipo de investigación utiliza el código NAMD, que calcula los estados de energía mínima de los átomos en un gran sistema biomolecular y es un indicador de qué formas serían las más cómodas asumiendo las moléculas. El equipo primero construye un modelo de la proteína MspA sumergida en una bicapa de lípidos y una solución de electrolitos. A continuación, se enhebra una hebra de ADN de una secuencia de nucleótidos deseada a través del nanoporo MspA. A continuación, los científicos simulan el efecto de un campo eléctrico que conduce iones y ADN a través del nanoporo MspA.
La simulación emplea dinámica molecular, o cálculos del movimiento de cada átomo en un sistema molecular siguiendo las leyes físicas de la naturaleza, para imitar el sistema experimental. Los resultados de las simulaciones se pueden comparar directamente con los de los experimentos porque ambos enfoques miden la corriente iónica, según Aksimentiev. Al conocer las posiciones de cada átomo e ion de ADN, Los científicos obtienen una ventaja:pueden optimizar la secuenciación de nanoporos utilizando un diseño racional para producir un poro que se adhiera al ADN con más fuerza. ralentizar el viaje de la molécula a través del poro a una velocidad que permita la resolución de un solo nucleótido.
El trabajo de secuenciación está financiado por el Instituto Nacional de Investigación del Genoma Humano de los Institutos Nacionales de Salud. El desarrollo del método del proyecto está financiado en parte por la National Science Foundation. Los colaboradores del proyecto incluyen dos grupos experimentales:uno dirigido por Jens Gundlach en la Universidad de Washington-Seattle y el otro por Michael Niederweis en la Universidad de Alabama-Birmingham.
La investigación recibió 10 millones de horas de procesador en Jaguar a través del Impacto computacional innovador y novedoso en la teoría y el experimento, o INCITE, programa, que otorga asignaciones considerables en algunas de las supercomputadoras más poderosas del mundo para proyectos que abordan grandes desafíos en ciencia e ingeniería. Con la dotación INCITE, los científicos pudieron reproducir las presas en el nanoporo MspA para el tipo de nucleótidos de ADN confinado a él, ralentizando el movimiento de la secuencia a través del nanoporo.
"Hemos llevado a cabo un estudio piloto sobre varias variantes del nanoporo MspA y hemos observado una reducción considerable de la velocidad de la cadena de ADN, ", Dijo Aksimentiev." Estos resultados muy preliminares sugieren que lograr una reducción de 100 veces de la velocidad del ADN, que debería ser suficiente para leer la secuencia de ADN con resolución de un solo nucleótido, está al alcance. Los estudios futuros se dirigirán hacia este objetivo ".
El equipo espera lograr el objetivo de este proyecto para 2013 y planea perseguir una serie de emocionantes proyectos derivados, Dijo Aksimentiev. La capacidad de hacer asequible la secuenciación del genoma permitirá programas como el Cancer Genome Project, que caracteriza las mutaciones del ADN en las células cancerosas en varios tejidos a lo largo de todas las etapas del desarrollo del cáncer.